Introduction à R
Last updated on 2025-05-05 | Edit this page
Estimated time: 120 minutes
Overview
Questions
- Premières commandes dans R
Objectives
- Définir les termes suivants relatifs à R : objet, affectation, appel, fonction, arguments, options.
- Attribuer des valeurs aux objets dans R.
- Apprendre à nommer des objets.
- Utiliser des commentaires pour informer le script.
- Résoudre des opérations arithmétiques simples dans R.
- Appeler des fonctions et utiliser des arguments pour modifier leurs options par défaut.
- Inspecter le contenu des vecteurs et manipulez leur contenu.
- Extraire un sous-ensemble ou des valeurs à partir de vecteurs.
- Analyser des vecteurs contenant des données manquantes.
Cet épisode est basé sur le cours Data Analysis and Visualization in R for Ecologists (Analyse de données et Visualisation en R pour les écologistes) de Data Carpentries.
Créer des objets dans R
En R, vous pouvez obtenir des résultats simplement en tapant des opérations mathématiques directement dans la console :
R
3 + 5
OUTPUT
[1] 8R
12 / 7
OUTPUT
[1] 1.714286Cependant, pour faire des analyses utiles et intéressantes, il est avantageux d’attribuer des valeurs à des objets. Pour créer un objet, nous devons lui donner un nom suivi de l’opérateur d’assignation `←
R
poids_kg <- 55
<- est l’opérateur d’assignation (affectation). Il
attribue des valeurs à droite aux objets à gauche. Ainsi, après avoir
exécuté x <- 3, la valeur de x est
3. La flèche peut être lue comme 3 entre
dans x. Pour des raisons historiques, vous pouvez
également utiliser = pour les assignations, mais pas dans
tous les contextes. En raison de légères
différences dans la syntaxe, une bonne pratique est de toujours
utiliser <- pour les assignations.
Dans RStudio, sur un PC, taper Alt + - (appuyer
sur Alt en même temps que la touche - ) écrira
<- en une seule frappe, tandis que taper
Option + - (appuyer sur Option en même
temps que la touche - ) fera de même sur un Mac.
Nommer les variables
Les objets peuvent avoir n’importe quel nom tel que « x », «
current_temperature » ou « subject_id ». Il est souhaitable que les noms
de vos objets soient explicites et pas trop longs. Ils ne peuvent pas
commencer par un nombre (2x n’est pas un nom valide, mais
x2 l’est). R est sensible à la casse (par exemple,
weight_kg est différent de Weight_kg).
Certains noms ne peuvent pas être utilisés car ils sont les noms de
fonctions fondamentales dans R (par exemple, if,
else, for, voir ici
pour une liste complète des noms réservés). En général, même si c’est
autorisé, il est préférable de de ne pas utiliser d’autres noms de
fonctions (par exemple, c, T,
mean, data, df,
weight). En cas de doute, consultez l’aide pour voir si le
nom est déjà utilisé. Il est également préférable d’éviter les points
(.) dans un nom d’objet comme dans my.dataset
(utilisez plutôt my_dataset par exemple). Vous remarquerez
cependant qu’il existe de nombreuses fonctions dans R avec des points
dans leurs noms. Ces noms de fonction sont conservés pour des raisons
historiques, mais comme les points ont une signification particulière en
R (pour les méthodes) et autres langages de programmation, il est
préférable de les éviter. Il est également recommandé d’utiliser des
noms pour les noms d’objets et des verbes pour les noms de fonctions. Il
est important d’être cohérent dans le style de votre code (où vous
placez les espaces, comment vous nommez les objets, etc.). L’utilisation
d’un style de codage cohérent rend votre code plus clair à lire pour vos
collaborateurs, mais aussi pour vous-même dans le futur (vous vous
remercierez plus tard). En R, il existe plusieurs guides de styles
populaires comme le guides de style de Google, du
tidyverse ou le Bioconductor
style guide. Le style Tidyverse est très complet mais peut sembler un
peu lourd au début. Vous pouvez installer le package lintr
qui vérifiera et corrigera automatiquement les problèmes ou incohérences
dans le style de votre code.
Objets et variables : ce que l’on appelle des « objets » en « R » sont connus sous le nom de « variables » dans de nombreux autres langages de programmation. Selon le contexte, « objet » et « variable » peuvent avoir des significations radicalement différentes. Cependant, dans cette leçon, les deux mots sont utilisés de manière synonyme. Pour plus d’informations voir ce lien.
Lors de l’attribution d’une valeur à un objet, R n’imprime rien dans la console. Vous pouvez forcer R à imprimer la valeur d’un objet en utilisant des parenthèses ou en tapant le nom de l’objet :
R
weight_kg <- 55 # n'imprime rien
(weight_kg <- 55) # mais mettre des parenthèses autour de l'appel imprime la valeur de `weight_kg`
OUTPUT
[1] 55R
weight_kg # et taper également le nom du objet
OUTPUT
[1] 55Maintenant que R a weight_kg en mémoire, nous pouvons
faire des opérations arithmétiques sur cet objet. Par exemple, nous
pouvons vouloir convertir ce poids en livres (le poids en livres est 2,2
fois le poids en kg) :
R
2.2 * weight_kg
OUTPUT
[1] 121On peut également changer la valeur d’un objet en lui attribuant une nouvelle valeur :
R
weight_kg <- 57.5
2.2 * weight_kg
OUTPUT
[1] 126.5L’attribution d’une valeur à un objet ne modifie pas les valeurs
d’autres objets. Par exemple, stockons le poids en livres dans un nouvel
objet weight_lb :
R
weight_lb <- 2.2 * weight_kg
puis remplacons weight_kg par 100.
R
weight_kg <- 100
Défi:
Selon vous, quel est maintenant le contenu de l’objet
weight_lb ? 126.5 ou 220?
Commentaires
En R, le caractère de commentaire est #. Tout ce qui se
trouve à droite d’un # dans un script sera ignoré par R.
Les commentaires sont utiles pour laisser des notes et explications dans
vos scripts.
RStudio permet de facilement commenter ou “décommenter” un paragraphe : après avoir sélectionné les lignes que vous souhaitez commenter (ou décommenter), appuyez simultanément sur les touches Ctrl + Shift + C. Si vous ne souhaitez commenter qu’une seule ligne, vous pouvez placer le curseur à n’importe quel emplacement de cette ligne (il n’est pas nécessaire de sélectionner la ligne entière), puis appuyez sur Ctrl + Shift + C.
Défi
Quelles sont les valeurs des objets après chaque instruction ci-dessous ?
R
mass <- 47.5 # mass?
age <- 122 # age?
mass <- mass * 2.0 # mass?
age <- age - 20 # age?
mass_index <- mass/age # mass_index?
Les fonctions et leurs arguments
Les fonctions sont des “scripts prédéfinis” qui automatisent des
ensembles de commandes plus complexes, y compris les affectations
d’opérations. De nombreuses fonctions sont prédéfinies ou peuvent être
rendues disponibles en important des packages R (nous en
parlerons plus tard). Une fonction “prend” généralement une ou plusieurs
entrées appelées arguments. Les fonctions renvoient souvent
(mais pas toujours) une valeur. Un exemple typique serait la
fonction sqrt(). L’entrée (l’argument) doit être un nombre
et la valeur de retour (la “sortie”) est la racine carrée de ce nombre.
Exécuter une fonction est appelé appeler la fonction (“call
a function” en anglais). Un exemple d’appel de fonction est :
R
b <- sqrt(a)
Ici, la valeur de a est donnée à la fonction
sqrt(), la fonction sqrt() en calcule la
racine carrée, et renvoie cette valeur qui est ensuite attribuée à
l’objet b. Cette fonction est simple car elle ne prend
qu’un seul argument.
La valeur de retour d’une fonction (sa sortie) n’est pas
nécessairement numérique (comme celle de sqrt()), et elle
n’est pas forcément un élément unique : une sortie peut être un ensemble
de choses, ou même un jeu de données. Nous en verrons un exemple lorsque
nous lirons des fichiers de données dans R.
Les arguments peuvent aussi être divers, pas uniquement des nombres ou noms de fichiers, mais aussi d’autres objets (listes, jeux de données, etc.). La signification exacte de chaque argument diffère selon la fonction et doit être recherchée dans la documentation (voir ci-dessous). Certaines fonctions ont des arguments qui peuvent être spécifiés par l’utilisateur ou prendre une valeur par défaut : ces arguments optionnels sont appelés options. Les options sont généralement utilisées pour modifier le fonctionnement de la fonction, par exemple si elle doit ignorer les « mauvaises valeurs » ou quel symbole utiliser dans un graphique. Si vous souhaitez un comportement spécifique pour une fonction, vous pouvez spécifier une valeur de votre choix qui sera utilisée à la place de la valeur par défaut.
Essayons maintenant une fonction qui peut prendre plusieurs arguments
: round().
R
round(3.14159)
OUTPUT
[1] 3Ici, nous avons appelé round() avec un seul argument,
3.14159, et il a renvoyé la valeur 3. En
effet, le comportement par défaut de la fonction round()
est d’arrondir au nombre entier le plus proche. Si nous souhaitons
conserver quelques chiffres décimaux, nous pouvons voir comment procéder
en obtenant des informations sur la fonction round.
args(round) ou ?round nous permettront
d’obternir des informations sur les arguments (optionnels) de la
fonction round.
R
args(round)
OUTPUT
function (x, digits = 0, ...)
NULLR
?round
Nous voyons que si nous souhaitons deux chiffres décimaux, nous
pouvons taper digits=2.
R
round(3.14159, digits = 2)
OUTPUT
[1] 3.14Si vous fournissez les arguments exactement dans le même ordre que celui dans lequel ils sont définis, vous n’avez pas besoin de les nommer :
R
round(3.14159, 2)
OUTPUT
[1] 3.14Et si vous nommez les arguments, vous pouvez changer leur ordre :
R
round(digits = 2, x = 3.14159)
OUTPUT
[1] 3.14Il est recommandé de placer les arguments non facultatifs (comme le nombre que vous arrondissez) en premier dans votre appel de fonction et de spécifier les noms de tous les arguments facultatifs. Si vous ne le faites pas, quelqu’un qui lit votre code devra peut-être rechercher la définition d’une fonction avec des arguments inconnus pour comprendre ce que vous faites. En spécifiant le nom des arguments, vous protégez également votre code contre d’éventuelles modifications futures dans l’interface de la fonction, qui peuvent potentiellement ajouter de nouveaux arguments entre ceux existants.
Vecteurs et types de données
Le vecteur est le type de données le plus courant, basique et central
de R. Un vecteur est composé d’une série de valeurs, telles que des
nombres ou caractères. Nous pouvons attribuer une série de valeurs à un
vecteur en utilisant la fonction c(). Par exemple, nous
pouvons créer un vecteur de poids d’animaux et l’attribuer à un nouvel
objet weight_g :
R
weight_g <- c(50, 60, 65, 82)
weight_g
OUTPUT
[1] 50 60 65 82Un vecteur peut également contenir des caractères :
R
molecules <- c("dna", "rna", "protein")
molecules
OUTPUT
[1] "dna" "rna" "protein"Les guillemets autour de "dna", "rna", etc.
sont ici essentiels. Sans les guillemets, R supposera qu’il existe des
objets appelés dna, rna et
protein. Comme ces objets n’existent pas dans la mémoire de
R, il y aura un message d’erreur.
Il existe de nombreuses fonctions qui vous permettent d’inspecter le
contenu d’un vecteur. length() vous indique combien
d’éléments se trouvent dans un vecteur particulier :
R
length(weight_g)
OUTPUT
[1] 4R
length(molecules)
OUTPUT
[1] 3Une caractéristique importante d’un vecteur est que tous les éléments
sont du même type de données. La fonction class() indique
la classe (le type d’élément) d’un objet :
R
class(weight_g)
OUTPUT
[1] "numeric"R
class(molecules)
OUTPUT
[1] "character"La fonction str() fournit un aperçu de la structure d’un
objet et de ses éléments. C’est une fonction utile lorsque vous
travaillez avec des objets volumineux et complexes :
R
str(weight_g)
OUTPUT
num [1:4] 50 60 65 82R
str(molecules)
OUTPUT
chr [1:3] "dna" "rna" "protein"Vous pouvez utiliser la fonction c() pour ajouter
d’autres éléments à votre vecteur :
R
weight_g <- c(weight_g, 90) # ajout à la fin du vecteur
weight_g <- c(30, weight_g) # ajout au début du vecteur
weight_g
OUTPUT
[1] 30 50 60 65 82 90Dans la première ligne, nous prenons le vecteur d’origine
weight_g, y ajoutons la valeur 90 à la fin et
enregistrons le résultat dans weight_g. Dans la deuxième
ligne, nous ajoutons la valeur « 30 » au début, en enregistrant à
nouveau le résultat dans « weight_g ».
Nous pouvons continuer à compléter ainsi un vecteur ou utiliser cette syntaxe pour assembler des données. Cela peut être utile pour ajouter les résultats que nous collectons ou calculons au fur et à mesure de notre analyse.
Un vecteur atomique est le type de
données R le plus simple; il s’agit d’un vecteur linéaire
composé d’éléments du même type. Plus tôt, nous avons vu 2 des 6
principaux types de vecteurs atomiques que R utilise :
"character" (caractères) et "numeric"
(chiffres, décimaux ou entiers) (ou "double" (réels)). Ce
sont les éléments de base à partir desquels tous les objets R sont
construits. Les 4 autres types de vecteurs atomiques
sont :
-
"logical"pourTRUEetFALSE(le type de données booléen) -
"integer"pour les nombres entiers (par exemple,2L, leLindique à R que c’est un entier) -
"complex"pour représenter des nombres complexes avec des parties réelles et imaginaires (par exemple,1 + 4i); et c’est tout ce que dirons à leur sujet -
"raw"pour les “flux de bits” (bitstreams) dont nous ne parlerons pas davantage
Vous pouvez vérifier le type de votre vecteur en utilisant la
fonction typeof() et en saisissant votre vecteur comme
argument.
Les vecteurs sont l’une des nombreuses structures de
données utilisées par R. Les autres structures importantes sont
les listes (list), les matrices (matrix), les
tables de données (data.frame), les facteurs
(factor) et les tableaux (array ).
Défi:
Nous avons vu que les vecteurs atomiques peuvent être de type caractère, numérique (ou double), entier et logique. Que se passe-t-il si nous essayons de mélanger ces types dans un seul vecteur ?
R les convertit implicitement pour qu’ils soient tous du même type
Défi:
Que se passera-t-il dans chacun de ces exemples ? (indice : utilisez
la fonction class() pour vérifier le type de données des
objets) :
R
num_char <- c(1, 2, 3, "a")
num_logical <- c(1, 2, 3, TRUE, FALSE)
char_logical <- c("a", "b", "c", TRUE)
tricky <- c(1, 2, 3, "4")
R
class(num_char)
OUTPUT
[1] "character"R
num_char
OUTPUT
[1] "1" "2" "3" "a"R
class(num_logical)
OUTPUT
[1] "numeric"R
num_logical
OUTPUT
[1] 1 2 3 1 0R
class(char_logical)
OUTPUT
[1] "character"R
char_logical
OUTPUT
[1] "a" "b" "c" "TRUE"R
class(tricky)
OUTPUT
[1] "character"R
tricky
OUTPUT
[1] "1" "2" "3" "4"Défi:
Pourquoi pensez-vous que cela arrive ?
Les éléments des vecteurs ne peuvent appartenir qu’à un seul type de données. R essaie donc de convertir le contenu de ce vecteur pour trouver un dénominateur commun qui n’amène à aucune perte d’information.
Défi:
Combien de valeurs dans combined_logical sont
"TRUE" (sous forme de caractère) dans l’exemple
suivant :
R
num_logical <- c(1, 2, 3, TRUE)
char_logical <- c("a", "b", "c", TRUE)
combined_logical <- c(num_logical, char_logical)
Seulement un. Il n’y a pas de mémoire des types de données passés et
la conversion (“coercition”) se produit la première fois que le vecteur
est évalué. Par conséquent, le TRUE dans
num_logical est converti en 1 avant d’être
converti en "1" dans combined_logical.
R
combined_logical
OUTPUT
[1] "1" "2" "3" "1" "a" "b" "c" "TRUE"Défi:
Dans R, nous appelons la conversion d’objets d’une classe vers une autre classe coercition. Ces conversions se produisent selon une hiérarchie, selon laquelle certains types sont préférentiellement contraints vers d’autres types. Pouvez-vous dessiner un diagramme qui représente la hiérarchie de la façon dont ces types de données sont forcés ?
logique → numérique → caractère ← logique
Extraire des valeurs d’un vecteur (Subsetting)
Si l’on veut extraire une ou plusieurs valeurs d’un vecteur, il faut fournir un ou plusieurs indices entre crochets. Par exemple:
R
molecules <- c("dna", "rna", "peptide", "protein")
molecules[2]
OUTPUT
[1] "rna"R
molecules[c(3, 2)]
OUTPUT
[1] "peptide" "rna" On peut également répéter les indices pour créer un objet avec plus d’éléments que celui d’origine :
R
more_molecules <- molecules[c(1, 2, 3, 2, 1, 4)]
more_molecules
OUTPUT
[1] "dna" "rna" "peptide" "rna" "dna" "protein"En R, les indices commencent à 1. Les langages de programmation comme Fortran, MATLAB, Julia et R commencent à compter à 1, car c’est ce que font généralement les êtres humains. Les langages de la famille C (y compris C++, Java, Perl, et Python) comptent à partir de 0 car c’est plus simple à faire pour les ordinateurs.
En utilisant des indices négatifs, on peut obtenir tous les éléments d’un vecteur sauf ceux spécifiés par les indices négatifs :
R
molecules ## all molecules
OUTPUT
[1] "dna" "rna" "peptide" "protein"R
molecules[-1] ## all but the first one
OUTPUT
[1] "rna" "peptide" "protein"R
molecules[-c(1, 3)] ## all but 1st/3rd ones
OUTPUT
[1] "rna" "protein"R
molecules[c(-1, -3)] ## all but 1st/3rd ones
OUTPUT
[1] "rna" "protein"Sous-ensemble conditionnel
Une autre méthode courante de sous-ensemble consiste à utiliser un
vecteur logique. TRUE sélectionnera l’élément avec le même
index, tandis que FALSE ne le fera pas :
R
weight_g <- c(21, 34, 39, 54, 55)
weight_g[c(TRUE, FALSE, TRUE, TRUE, FALSE)]
OUTPUT
[1] 21 39 54Généralement, ces vecteurs logiques ne sont pas tapés à la main, mais sont le résultat d’autres fonctions ou tests logiques. Par exemple, si vous souhaitez sélectionner uniquement les valeurs supérieures à 50 :
R
## will return logicals with TRUE for the indices that meet
## the condition
weight_g > 50
OUTPUT
[1] FALSE FALSE FALSE TRUE TRUER
## so we can use this to select only the values above 50
weight_g[weight_g > 50]
OUTPUT
[1] 54 55Vous pouvez combiner plusieurs tests en utilisant &
(les deux conditions sont vraies, AND) ou | (au moins une
des conditions est vraie, OR) :
R
weight_g[weight_g < 30 | weight_g > 50]
OUTPUT
[1] 21 54 55R
weight_g[weight_g >= 30 & weight_g == 21]
OUTPUT
numeric(0)Ici, < signifie “inférieur à”, >
signifie “supérieur à”, >= signifie “supérieur ou égal
à” et == signifie “égal à”. Le double signe égal
== est un test d’égalité numérique entre les côtés gauche
et droit, et ne doit pas être confondu avec le signe simple
=, qui effectue une affectation de variable (similaire à
<-).
Une tâche courante consiste à rechercher certaines chaînes de
caractères dans un vecteur. On pourrait utiliser l’opérateur “ou”
| pour tester l’égalité de plusieurs valeurs, mais cela
peut rapidement devenir fastidieux. La fonction %in% permet
de tester si l’un des éléments d’un vecteur de recherche est trouvé
:
R
molecules <- c("dna", "rna", "protein", "peptide")
molecules[molecules == "rna" | molecules == "dna"] # returns both rna and dna
OUTPUT
[1] "dna" "rna"R
molecules %in% c("rna", "dna", "metabolite", "peptide", "glycerol")
OUTPUT
[1] TRUE TRUE FALSE TRUER
molecules[molecules %in% c("rna", "dna", "metabolite", "peptide", "glycerol")]
OUTPUT
[1] "dna" "rna" "peptide"Défi:
À votre avis, pourquoi est-ce "four" > "five"
("quatre" > "cinq") renvoie TRUE ?
R
"four" > "five"
OUTPUT
[1] TRUELorsque vous utilisez > ou < sur des
chaînes de caractère, R compare leur ordre alphabétique. Ici,
"four" (quatre) vient alphabétiquement après
"five" (cinq), et est donc supérieur à cinq.
Noms d’éléments d’un vecteur
Il est possible de nommer chaque élément d’un vecteur. Le code ci-dessous montre un exemple d’un vecteur dont les éléments n’ont initialement aucun nom, et comment on peut ensuite définir et récupérer les noms des éléments.
R
x <- c(1, 5, 3, 5, 10)
names(x) ## no names
OUTPUT
NULLR
names(x) <- c("A", "B", "C", "D", "E")
names(x) ## now we have names
OUTPUT
[1] "A" "B" "C" "D" "E"Lorsqu’un vecteur possède des noms, il est possible d’accéder aux éléments par leur nom, en plus de par leur index.
R
x[c(1, 3)]
OUTPUT
A C
1 3 R
x[c("A", "C")]
OUTPUT
A C
1 3 Données manquantes
Comme R a été conçu pour analyser des jeux de données, il inclut le
concept de données manquantes (ce qui est rare dans d’autres langages de
programmation). Les données manquantes sont représentées dans les
vecteurs par NA (pour not applicable).
Lorsque vous effectuez des opérations sur des nombres, la plupart des
fonctions renverront NA si les données avec lesquelles vous
travaillez incluent des valeurs manquantes. Cette fonctionnalité par
défaut rend plus difficile l’ignorance des cas où vous avez affaire à
des données manquantes. Vous pouvez ajouter l’argument
na.rm = TRUE pour calculer le résultat en ignorant les
valeurs manquantes (na.rm peut se traduire par “remove
NA”, où “remove” signifie “enlever” en anglais).
R
heights <- c(2, 4, 4, NA, 6)
mean(heights)
OUTPUT
[1] NAR
max(heights)
OUTPUT
[1] NAR
mean(heights, na.rm = TRUE)
OUTPUT
[1] 4R
max(heights, na.rm = TRUE)
OUTPUT
[1] 6Si vos données incluent des valeurs manquantes, vous souhaiterez
peut-être vous familiariser avec les fonctions is.na(),
na.omit() et complete.cases(). Voici quelques
exemples.
R
## Extrait les éléments qui ne sont pas manquants.
heights[!is.na(heights)]
OUTPUT
[1] 2 4 4 6R
## Renvois un objet dont les cas incomplets ont été supprimés.
## L'objet renvoyé est un vecteur atomique du type "numeric"
## (ou "double").
na.omit(heights)
OUTPUT
[1] 2 4 4 6
attr(,"na.action")
[1] 4
attr(,"class")
[1] "omit"R
## Extrait les éléments qui représentent des cas complets.
## L'objet renvoyé est un vecteur atomique du type "numeric"
## (ou "double").
heights[complete.cases(heights)]
OUTPUT
[1] 2 4 4 6Défi:
- En utilisant ce vecteur de hauteurs en pouces (inches), créez un nouveau vecteur en supprimant les NA.
R
heights <- c(63, 69, 60, 65, NA, 68, 61, 70, 61, 59, 64, 69, 63, 63, NA, 72, 65, 64, 70, 63, 65)
- Utilisez la fonction
median()pour calculer la médiane du vecteurheights. - Utilisez R pour déterminer combien de personnes dans l’échantillon mesurent plus de 67 pouces.
R
heights_no_na <- heights[!is.na(heights)]
## or
heights_no_na <- na.omit(heights)
R
median(heights, na.rm = TRUE)
OUTPUT
[1] 64R
heights_above_67 <- heights_no_na[heights_no_na > 67]
length(heights_above_67)
OUTPUT
[1] 6Génération de vecteurs
Constructeurs
Il existe quelques fonctions pour générer des vecteurs de différents
types. Pour générer un vecteur de valeurs numériques, on peut utiliser
le constructeur numeric(), en lui fournissant la longueur
du vecteur de sortie comme paramètre. Les valeurs seront initialisées à
0.
R
numeric(3)
OUTPUT
[1] 0 0 0R
numeric(10)
OUTPUT
[1] 0 0 0 0 0 0 0 0 0 0Notez que si l’on demande un vecteur de numériques de longueur 0, on obtient exactement cela :
R
numeric(0)
OUTPUT
numeric(0)Il existe des constructeurs similaires pour les caractères et les
logiques, nommés respectivement character() et
logical().
Défi:
Quelles sont les valeurs par défaut pour les vecteurs de caractères et les vecteurs logiques ?
R
character(2) ## the empty character
OUTPUT
[1] "" ""R
logical(2) ## FALSE
OUTPUT
[1] FALSE FALSERépliquer des éléments
La fonction rep permet de répéter une valeur un certain
nombre de fois. Si nous voulons initier un vecteur de numériques de
longueur 5 avec la valeur -1, par exemple, nous pourrions faire ce qui
suit :
R
rep(-1, 5)
OUTPUT
[1] -1 -1 -1 -1 -1De même, pour générer un vecteur rempli de valeurs manquantes, ce qui est souvent une bonne façon de commencer, sans poser d’hypothèses sur les données qui seront collectées :
R
rep(NA, 5)
OUTPUT
[1] NA NA NA NA NArep peut prendre en entrée des vecteurs de n’importe
quelle longueur (ci-dessus, nous avons utilisé des vecteurs de longueur
1) et de n’importe quel type. Par exemple, si nous voulions répéter cinq
fois les valeurs 1, 2 et 3, nous procéderions comme suit :
R
rep(c(1, 2, 3), 5)
OUTPUT
[1] 1 2 3 1 2 3 1 2 3 1 2 3 1 2 3Défi:
Et si nous voulions répéter les valeurs 1, 2 et 3 cinq fois, mais
obtenir cinq 1, cinq 2 et cinq 3 dans cet ordre, comment devrions-nous
procéder ? Il existe deux possibilités: voir ?rep ou
?sort pour obtenir de l’aide.
R
rep(c(1, 2, 3), each = 5)
OUTPUT
[1] 1 1 1 1 1 2 2 2 2 2 3 3 3 3 3R
sort(rep(c(1, 2, 3), 5))
OUTPUT
[1] 1 1 1 1 1 2 2 2 2 2 3 3 3 3 3Génération de séquences
Une autre fonction très utile est seq, pour générer une
séquence de nombres. Par exemple, pour générer une séquence d’entiers de
1 à 20 par pas de 2, on utiliserait :
R
seq(from = 1, to = 20, by = 2)
OUTPUT
[1] 1 3 5 7 9 11 13 15 17 19La valeur par défaut de by est 1 et, étant donné que la
génération d’une séquence d’une valeur à une autre avec des pas de 1 est
fréquemment utilisée, il existe un raccourci :
R
seq(1, 5, 1)
OUTPUT
[1] 1 2 3 4 5R
seq(1, 5) ## default by
OUTPUT
[1] 1 2 3 4 5R
1:5
OUTPUT
[1] 1 2 3 4 5Pour générer une séquence de nombres de 1 à 20 de longueur finale de 3, on utiliserait :
R
seq(from = 1, to = 20, length.out = 3)
OUTPUT
[1] 1.0 10.5 20.0Échantillons aléatoires et permutations
Un dernier groupe de fonctions utiles sont celles qui génèrent des
données aléatoires. La première, sample, génère une
permutation aléatoire d’un autre vecteur. Par exemple, pour tirer au
sort un ordre aléatoire de 10 étudiants pour leur examen oral,
j’attribue d’abord à chaque étudiant·e un numéro de 1 à dix (par exemple
en fonction de l’ordre alphabétique de leurs noms) puis :
R
sample(1:10)
OUTPUT
[1] 9 4 7 1 2 5 3 10 6 8Sans autres arguments, sample renverra une permutation
de tous les éléments du vecteur. Si je veux un échantillon aléatoire
d’une certaine taille, je définirais cette valeur comme deuxième
argument. Ci-dessous, j’échantillonne 5 lettres aléatoires de l’alphabet
contenu dans le vecteur letters prédéfini :
R
sample(letters, 5)
OUTPUT
[1] "s" "a" "u" "x" "j"Si je voulais une sortie plus grande que le vecteur d’entrée, ou
pouvoir tirer au sort certains éléments plusieurs fois, je devrais
définir l’argument replace à TRUE :
R
sample(1:5, 10, replace = TRUE)
OUTPUT
[1] 2 1 5 5 1 1 5 5 2 2Défi:
En essayant les fonctions ci-dessus, vous aurez réalisé que les
échantillons sont effectivement aléatoires et qu’on n’obtient pas deux
fois la même permutation. Pour pouvoir reproduire ces tirages
aléatoires, on peut définir manuellement la “graine d’échantillonage” de
nombres aléatoires avec la fonction set.seed() avant de
tirer l’échantillon aléatoire.
Testez cette fonctionnalité avec votre voisin. Tirez d’abord deux permutations aléatoires de « 1:10 » indépendamment et observez que vous obtenez résultats différents.
Définissez maintenant la graine avec, par exemple,
set.seed(123) et répétez le tirage au sort. Observez que
vous obtenez désormais les mêmes tirages.
Répétez en définissant une graine différente.
Différentes permutations
R
sample(1:10)
OUTPUT
[1] 9 1 4 3 6 2 5 8 10 7R
sample(1:10)
OUTPUT
[1] 4 9 7 6 1 10 8 3 2 5Mêmes permutations avec la graine 123
R
set.seed(123)
sample(1:10)
OUTPUT
[1] 3 10 2 8 6 9 1 7 5 4R
set.seed(123)
sample(1:10)
OUTPUT
[1] 3 10 2 8 6 9 1 7 5 4Une graine différente
R
set.seed(1)
sample(1:10)
OUTPUT
[1] 9 4 7 1 2 5 3 10 6 8R
set.seed(1)
sample(1:10)
OUTPUT
[1] 9 4 7 1 2 5 3 10 6 8Échantillonner à partir d’une distribution normale
La dernière fonction que nous allons voir est rnorm, qui
tire un échantillon aléatoire à partir d’une distribution normale. Deux
distributions normales de moyennes 0 et 100 et d’écarts types 1 et 5,
notées N(0, 1) et N(100, 5), sont présentées
graphiquement ci-dessous.
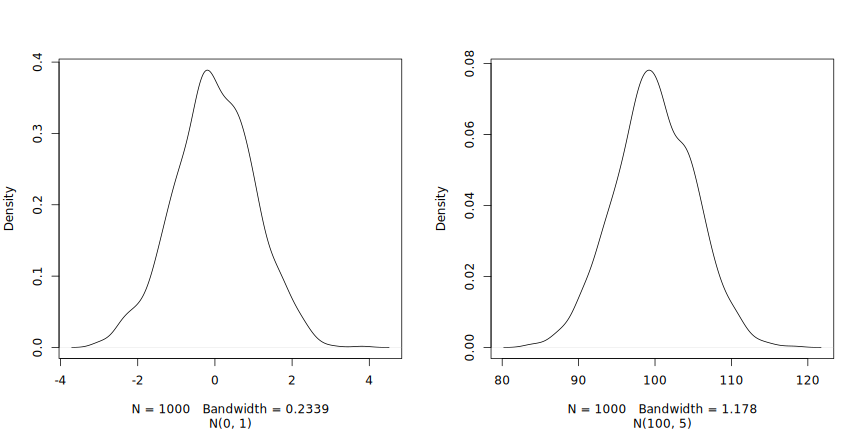
Les trois arguments, n, mean et
sd, définissent la taille de l’échantillon, et les
paramètres de la distribution normale, c’est-à-dire sa moyenne et son
écart type. Les valeurs par défaut de ces derniers sont 0 et 1.
R
rnorm(5)
OUTPUT
[1] 0.69641761 0.05351568 -1.31028350 -2.12306606 -0.20807859R
rnorm(5, 2, 2)
OUTPUT
[1] 1.3744268 -0.1164714 2.8344472 1.3690969 3.6510983R
rnorm(5, 100, 5)
OUTPUT
[1] 106.45636 96.87448 95.62427 100.71678 107.12595Maintenant que nous avons appris à écrire des scripts et connaissons les bases des structures de données de R, nous sommes prêts à commencer à travailler avec des données plus volumineuses et à en apprendre davantage sur les “data frames” (tables de données).
Key Points
- Comment interagir avec R
