R et RStudio
Dernière mise à jour le 2024-09-12 | Modifier cette page
Vue d'ensemble
Questions
- Que sont R et RStudio ?
Objectifs
- Décrivez l’objectif des volets RStudio Script, Console, Environment et Plots.
- Organisez les fichiers et les répertoires pour un ensemble d’analyses en tant que projet R et comprenez le but du répertoire de travail.
- Utilisez l’interface d’aide intégrée de RStudio pour rechercher plus d’informations sur les fonctions R.
- Montrez comment fournir suffisamment d’informations pour le dépannage avec la communauté des utilisateurs R.
Cet épisode est basé sur la leçon Analyse des données et Visualisation dans R pour les écologistes de Data Carpentries.
Qu’est-ce que R ? Qu’est-ce que RStudio ?
Le terme R est utilisé pour désigner le langage de programmation, l’environnement de calcul statistique et le logiciel qui interprète les scripts écrits à l’aide de celui-ci.
RStudio est actuellement un moyen très populaire non seulement d’écrire vos scripts R mais aussi d’interagir avec le logiciel R 1. Pour fonctionner correctement, RStudio a besoin de R et donc les deux doivent être installés sur votre ordinateur.
La RStudio IDE Cheat Sheet fournit beaucoup plus d’informations que ce qui sera couvert ici, mais peut être utile pour apprendre les raccourcis clavier et découvrir de nouvelles fonctionnalités.
Pourquoi apprendre R ?
R n’implique pas beaucoup de pointage et de clic, et c’est une bonne chose
The learning curve might be steeper than with other software, but with R, the results of your analysis do not rely on remembering a succession of pointing and clicking, but instead on a series of written commands, and that’s a good thing! Ainsi, si vous souhaitez refaire votre analyse parce que vous avez collecté plus de données, vous n’avez pas besoin de vous rappeler sur quel bouton vous avez cliqué dans quel ordre pour obtenir vos résultats ; il vous suffit de réexécuter votre script.
Travailler avec des scripts rend les étapes que vous avez utilisées dans votre analyse claires, et le code que vous écrivez peut être inspecté par quelqu’un d’autre qui peut vous donner des commentaires et repérer les erreurs.
Travailler avec des scripts vous oblige à avoir une compréhension plus profonde de ce que vous faites et facilite votre apprentissage et votre compréhension des méthodes que vous utilisez.
Le code R est idéal pour la reproductibilité
La reproductibilité signifie que quelqu’un d’autre (y compris votre futur moi) peut obtenir les mêmes résultats à partir du même ensemble de données en utilisant le même code d’analyse .
R s’intègre à d’autres outils pour générer des manuscrits ou des rapports à partir de votre code . Si vous collectez plus de données ou corrigez une erreur dans votre ensemble de données, les chiffres et les tests statistiques de votre manuscrit ou rapport sont mis à jour automatiquement.
Un nombre croissant de revues et d’agences de financement s’attendent à ce que les analyses soient reproductibles, donc connaître R vous donnera un avantage avec ces exigences.
R est interdisciplinaire et extensible
Avec plus de 10 000 packages2 pouvant être installés pour étendre ses capacités, R fournit un cadre qui vous permet de combiner des approches statistiques de nombreuses disciplines scientifiques pour s’adapter au mieux à le cadre analytique dont vous avez besoin pour analyser vos données. Par exemple, R propose des packages pour l’analyse d’images, le SIG, les séries chronologiques, la génétique de population et bien plus encore.
, the Comprehensive R Archive Network. From the R Journal, Volume 10/2, December 2018.](fig/cran.png)
R fonctionne sur des données de toutes formes et tailles
Les compétences que vous apprenez avec R évoluent facilement avec la taille de votre ensemble de données . Que votre ensemble de données comporte des centaines ou des millions de lignes, cela ne fera pas beaucoup de différence pour vous.
R est conçu pour l’analyse des données. Il est livré avec des structures de données spéciales et des types de données qui facilitent la gestion des données manquantes et des facteurs statistiques .
R peut se connecter à des feuilles de calcul, des bases de données et à de nombreux autres formats de données, sur votre ordinateur ou sur le Web.
R produit des graphiques de haute qualité
Les fonctionnalités de traçage de R sont étendues et vous permettent d’ajuster n’importe quel aspect de votre graphique pour transmettre le plus efficacement possible le message de vos données.
R a une communauté nombreuse et accueillante
Des milliers de personnes utilisent R quotidiennement. Beaucoup d’entre eux sont prêts à vous aider via des listes de diffusion et des sites Web tels que Stack Overflow, ou sur le RStudio communauté. Ces larges communautés d’utilisateurs s’étendent à des domaines spécialisés tels que la bioinformatique. L’un de ces sous-ensembles de la communauté R est Bioconductor, un projet scientifique pour l’analyse et la compréhension « des données provenant d’essais biologiques actuels et émergents ». Cet atelier a été développé par des membres de la communauté Bioconductor ; pour plus d’informations sur Bioconductor, veuillez consulter l’atelier complémentaire “The Bioconductor Project”.
Non seulement R est gratuit, mais il est également open source et multiplateforme
N’importe qui peut inspecter le code source pour voir comment R fonctionne. Grâce à cette transparence, il y a moins de risques d’erreurs, et si vous (ou quelqu’un d’autre) en trouvez, vous pouvez signaler et corriger des bugs.
Connaître RStudio
Commençons par découvrir RStudio, qui est un environnement de développement intégré (IDE) permettant de travailler avec R.
Le produit open source RStudio IDE est gratuit sous la Affero General Public License (AGPL) v3. L’IDE RStudio est également disponible avec une licence commerciale et une assistance prioritaire par courrier électronique de Posit, Inc.
Nous utiliserons l’IDE RStudio pour écrire du code, parcourir les fichiers sur notre ordinateur, inspecter les variables que nous allons créer et visualiser les tracés que nous allons générer. RStudio peut également être utilisé pour d’autres choses (par exemple, le contrôle de version, le développement de packages, l’écriture d’applications Shiny) que nous n’aborderons pas pendant l’atelier.
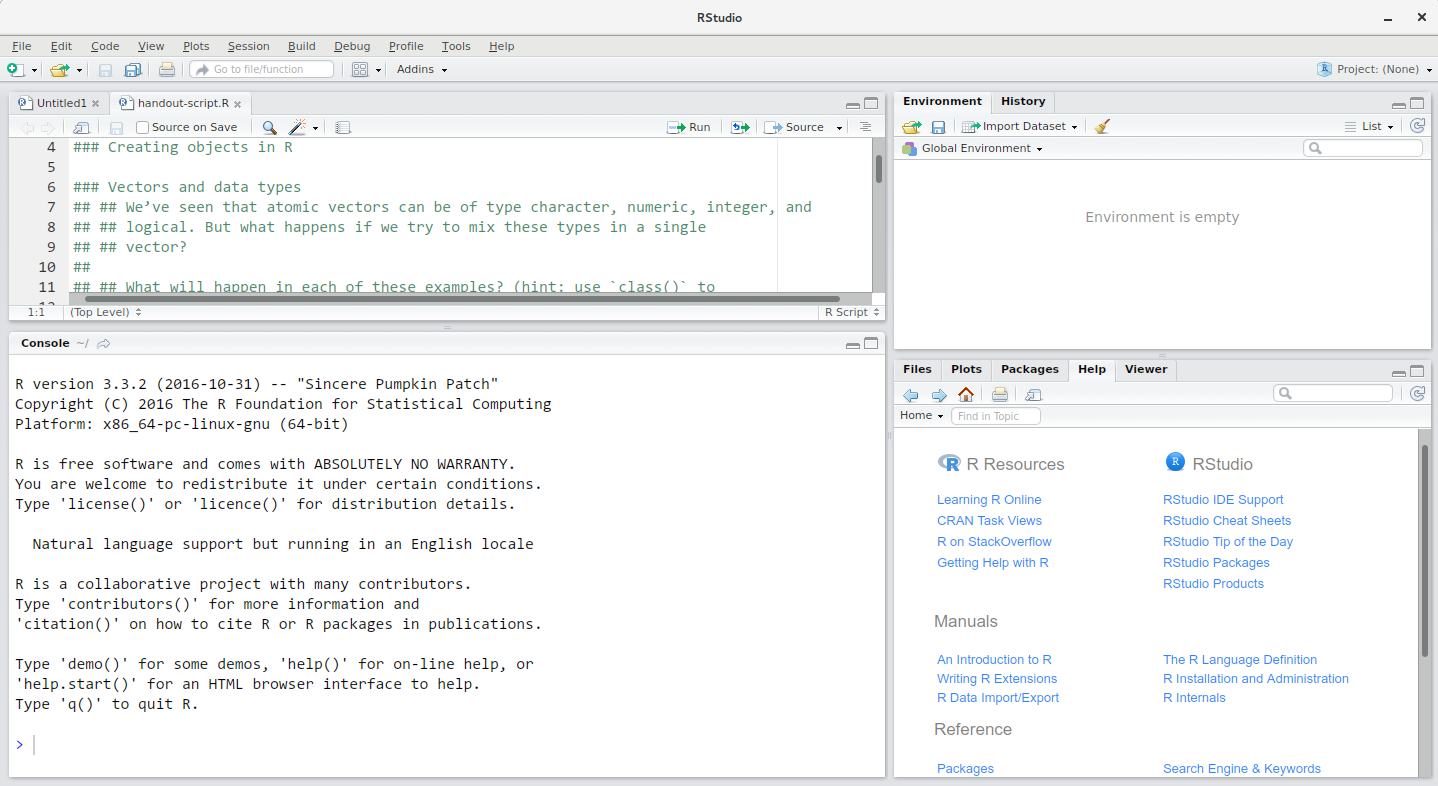
La fenêtre RStudio est divisée en 4 “Volets” :
- la Source de vos scripts et documents (en haut à gauche, dans la mise en page par défaut )
- votre Environnement/Historique (en haut à droite),
- vos Fichiers/Tracés/Packages/Aide/Visionneuse (en bas à droite), et
- la R Console (en bas à gauche).
L’emplacement de ces volets et leur contenu peuvent être
personnalisés (voir le menu ,
Outils -> Options globales -> Disposition des volets).
L’un des avantages de l’utilisation de RStudio est que toutes les informations dont vous avez besoin pour écrire du code sont disponibles dans une seule fenêtre. De plus, avec de nombreux raccourcis, la complétion automatique et la mise en surbrillance pour les principaux types de fichiers que vous utilisez lors du développement dans R, RStudio facilitera la saisie de et moins sujet aux erreurs.
Mise en place
Il est recommandé de conserver un ensemble de données, d’analyses et de textes connexes autonomes dans un seul dossier, appelé **répertoire de travail **. Tous les scripts de ce dossier peuvent alors utiliser chemins relatifs vers les fichiers qui indiquent où dans le projet se trouve un fichier (par opposition aux chemins absolus, qui pointent vers l’endroit où se trouve un fichier ). se trouve sur un ordinateur spécifique). Travailler de cette façon rend beaucoup plus facile le déplacement de votre projet sur votre ordinateur et le partage avec d’autres sans vous soucier de savoir si les scripts sous-jacents fonctionneront toujours.
RStudio fournit un ensemble d’outils utiles pour ce faire via son interface “Projets” , qui non seulement crée un répertoire de travail pour vous, mais mémorise également son emplacement (vous permettant d’y accéder rapidement ) et conserve éventuellement les paramètres personnalisés et les fichiers ouverts pour faciliter la reprise du travail après une pause. Suivez les étapes de création d’un “Projet R” pour ce tutoriel ci-dessous.
- Démarrez RStudio.
- Dans le menu « Fichier », cliquez sur « Nouveau projet ». Choisissez
Nouveau répertoire, puisNouveau projet. - Entrez un nom pour ce nouveau dossier (ou “répertoire”) et
choisissez un emplacement pratique pour celui-ci. Ce sera votre
répertoire de travail pour cette session (ou tout le
cours) (par exemple,
bioc-intro). - Cliquez sur « Créer un projet ».
- (Facultatif) Définissez les préférences sur « Jamais » pour enregistrer l’espace de travail dans RStudio.
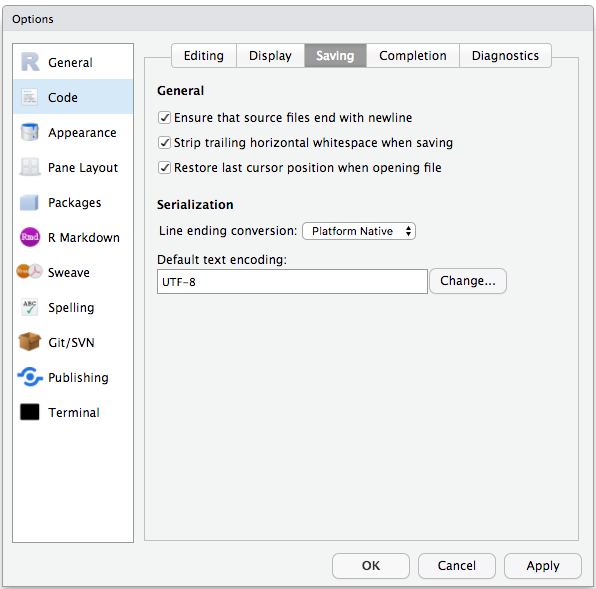
Les préférences par défaut de RStudio fonctionnent généralement bien, mais enregistrer un espace de travail dans .RData peut être fastidieux, surtout si vous travaillez avec des ensembles de données plus volumineux. Pour désactiver cela, allez dans Outils –> « Options globales » et sélectionnez l’option « Jamais » pour « Enregistrer l’espace de travail dans .RData » à la sortie.

Pour éviter les problèmes d’encodage des caractères entre Windows et d’autres systèmes d’exploitation, nous allons définir UTF-8 par défaut :
Organiser votre répertoire de travail
L’utilisation d’une structure de dossiers cohérente dans vos projets aidera à garder les choses organisées et facilitera également la recherche/le classement des éléments à l’avenir. Ce peut être particulièrement utile lorsque vous avez plusieurs projets. En général, vous pouvez créer des répertoires (dossiers) pour les scripts, données et documents.
-
data/Utilisez ce dossier pour stocker vos données brutes et les ensembles de données intermédiaires que vous pouvez créer pour les besoins d’une analyse particulière. Par par souci de transparence et de provenance, vous devez toujours conserver une copie de votre données brutes accessibles et effectuez autant de le nettoyage et le prétraitement de vos données par programme (c’est-à-dire avec scripts, plutôt que manuellement) que possible. Séparer les données brutes des données traitées est également une bonne idée. Par exemple, vous pourriez avoir les fichiersdata/raw/tree_survey.plot1.txtet...plot2.txtconservés séparés d’undata/processed/tree.survey. fichier csvgénéré par le scriptscripts/01.preprocess.tree_survey.R. -
documents/Ce serait un endroit pour conserver les plans, les brouillons, les et d’autres textes. -
scripts/(ousrc) Ce serait l’emplacement où conserver vos scripts R pour différentes analyses ou traçages, et potentiellement un dossier séparé pour vos fonctions (plus nous y reviendrons plus tard).
Vous souhaiterez peut-être des répertoires ou sous-répertoires supplémentaires en fonction de les besoins de votre projet, mais ceux-ci devraient constituer l’épine dorsale de votre répertoire de travail .
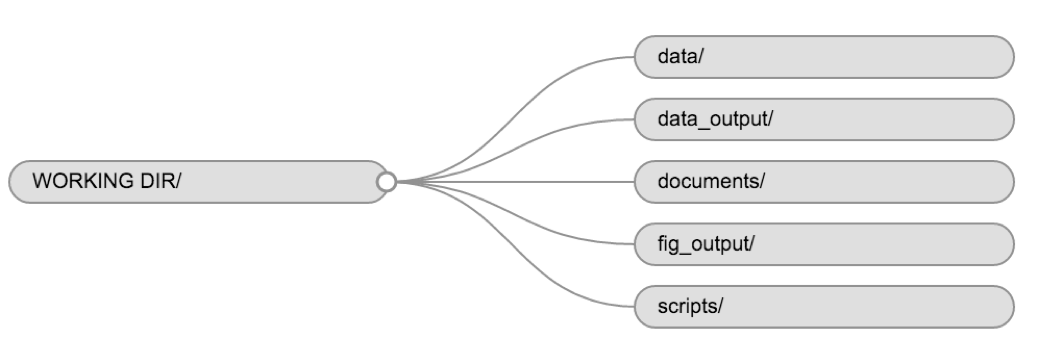
Pour ce cours, nous aurons besoin d’un dossier data/
pour stocker nos données brutes, et nous utiliserons
data_output/ lorsque nous apprendrons à exporter des
données sous forme de fichiers CSV, et Dossier fig_output/
pour les figures que nous allons enregistrer.
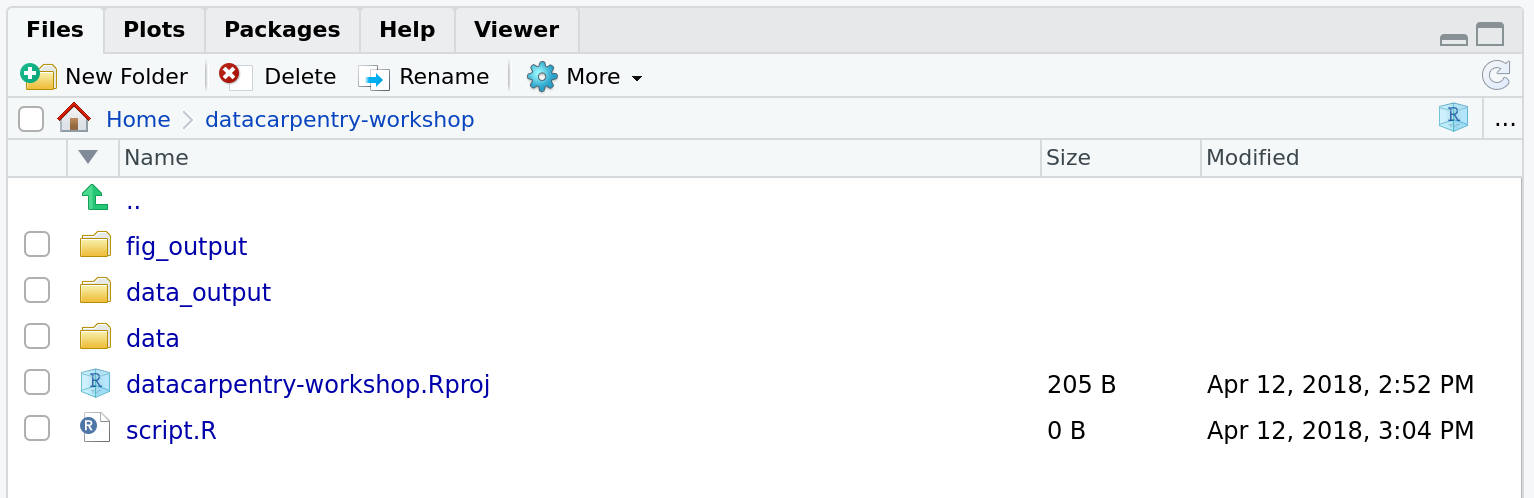
Défi : créer la structure de répertoires de votre projet
Sous l’onglet « Fichiers » à droite de l’écran, cliquez sur « Nouveau
dossier » et créez un dossier nommé « données » dans votre répertoire de
travail nouvellement créé (par exemple, « ~/bioc
-intro/données). (Vous pouvez également taperdir.create(“data”)sur votre console R.) Répétez ces opérations pour créer un dossierdata_output/et unfig_output`.
Nous allons conserver le script à la racine de notre répertoire de travail car nous n’allons utiliser qu’un seul fichier et cela rendra les choses plus faciles.
Votre répertoire de travail devrait maintenant ressembler à ceci :
La gestion de projet s’applique également aux projets de bioinformatique, bien sûr3. William Noble (@Noble:2009) propose la structure de répertoires suivante :
Les noms de répertoires sont en gros caractères et les noms de fichiers sont en caractères plus petits . Seul un sous-ensemble des fichiers est affiché ici. Notez que les dates sont formatées
<year>-<month>-<day>afin qu’elles puissent être triées par ordre chronologique. Le code sourcesrc/ms-analysis.cest compilé pour créerbin/ms-analysiset est documenté dansdoc/ms-analysis.html. Les fichiersREADMEdans les répertoires de données précisent qui a téléchargé les fichiers de données à partir de quelle URL et à quelle date . Le script du piloteresults/2009-01-15/runallgénère automatiquement les trois sous-répertoires split1, split2 et split3, correspondant à trois divisions de validation croisée. Le scriptbin/parse-sqt.pyest appelé par les deux scripts du piloterunall.
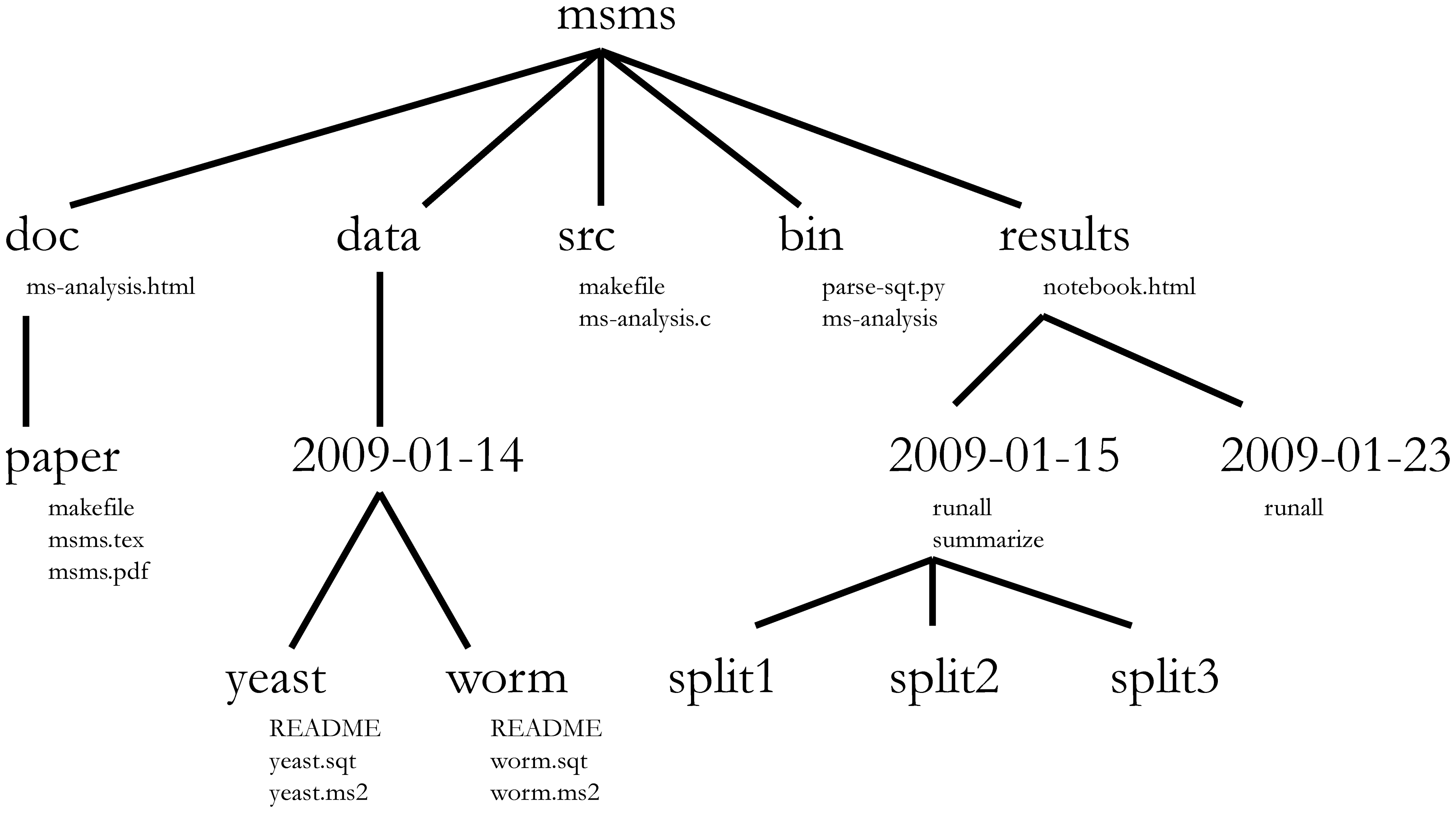
L’aspect le plus important d’un répertoire de projet bien défini et bien documenté est de permettre à quelqu’un qui n’est pas familier avec le projet 4 de
comprendre en quoi consiste le projet, quelles données sont disponibles, quelles analyses ont été effectuées et quels résultats ont été produits et, plus important encore,
répétez l’analyse à nouveau - avec de nouvelles données ou en modifiant certains paramètres d’analyse .
Le répertoire de travail
Le répertoire de travail est un concept important à comprendre. C’est l’endroit à partir duquel R recherchera et enregistrera les fichiers. Lorsque vous écrivez du code pour votre projet, il doit faire référence à des fichiers en relation avec la racine de votre répertoire de travail et n’a besoin que de fichiers au sein de cette structure .
L’utilisation de projets RStudio facilite cela et garantit que votre
répertoire de travail est correctement défini. Si vous avez besoin de le
vérifier, vous pouvez utiliser getwd(). If for some reason
your working directory is not what it should be, you can change it in
the RStudio interface by navigating in the file browser where your
working directory should be, and clicking on the blue gear icon
More, and select Set As Working Directory.
Vous pouvez également utiliser
setwd("/path/to/working/directory") pour réinitialiser
votre répertoire de travail. Cependant, vos scripts ne doivent pas
inclure cette ligne car elle échouera sur l’ordinateur de quelqu’un
d’autre.
Exemple
Le schéma ci-dessous représente le répertoire de travail
bioc-intro avec les sous-répertoires data et
fig_output, et 2 fichiers dans ce dernier :
bioc-intro/data/
/fig_output/fig1.pdf
/fig_output/fig2.pngSi on était dans le répertoire de travail, on pourrait faire
référence au fichier fig1.pdf en utilisant le chemin
relatif bioc-intro/fig_output/fig1.pdf ou le chemin absolu
/ accueil/user/bioc-intro/fig_output/fig1.pdf.
Si nous étions dans le répertoire data, nous
utiliserions le chemin relatif ../fig_output/fig1.pdf ou le
même chemin absolu
/home/user/bioc-intro /fig_output/fig1.pdf.
Interagir avec R
La base de la programmation est que nous écrivons les instructions que l’ordinateur doit suivre, puis nous disons à l’ordinateur de suivre ces instructions . Nous écrivons, ou codeons, des instructions dans R car c’est un langage commun que l’ordinateur et nous pouvons comprendre. Nous appelons les instructions commandes et nous disons à l’ordinateur de suivre les instructions en exécutant (également appelé exécutant) ces commandes.
Il existe deux manières principales d’interagir avec R : en utilisant la console ou en utilisant des scripts (fichiers texte brut contenant votre code). Le volet de la console (dans RStudio, le panneau inférieur gauche) est l’endroit où les commandes écrites en langage R peuvent être saisies et exécutées immédiatement par l’ordinateur. C’est également là que les résultats seront affichés pour les commandes exécutées. Vous pouvez taper des commandes directement dans la console et appuyer sur « Entrée » pour exécuter ces commandes , mais elles seront oubliées lorsque vous fermerez la session.
Parce que nous voulons que notre code et notre flux de travail soient reproductibles, il est préférable de taper les commandes souhaitées dans l’éditeur de script et d’enregistrer le script . De cette façon, il existe un enregistrement complet de ce que nous avons fait, et n’importe qui (y compris notre futur moi !) peuvent facilement reproduire les résultats sur leur ordinateur. Notez cependant que le simple fait de taper les commandes dans le script ne les exécute pas automatiquement - elles doivent quand même être envoyées à la console pour exécution.
RStudio vous permet d’exécuter des commandes directement depuis
l’éditeur de script en utilisant le raccourci Ctrl +
Entrée (sur Mac, Cmd + Return
fonctionnera également). La commande sur la ligne actuelle du script
(indiquée par le curseur) ou toutes les commandes dans le texte
actuellement sélectionné seront envoyées à la console et exécutées
lorsque vous appuyez sur Ctrl + Entrer. Vous
pouvez trouver d’autres raccourcis clavier dans cette aide-mémoire
RStudio sur l’IDE RStudio .
À un moment donné de votre analyse, vous souhaiterez peut-être
vérifier le contenu d’une variable ou la structure d’un objet, sans
nécessairement en conserver un enregistrement dans votre script. Vous
pouvez taper ces commandes et les exécuter directement dans la console.
RStudio fournit les raccourcis Ctrl + 1 et
Ctrl + 2 vous permettant de passer entre le
script et les volets de la console .
Si R est prêt à accepter les commandes, la console R affiche une
invite >. If it receives a command (by typing,
copy-pasting or sending from the script editor using Ctrl +
Enter), R will try to execute it, and when ready, will show
the results and come back with a new > prompt to wait
for new commands.
Si R attend toujours que vous saisissiez plus de données parce que
n’est pas encore terminé, la console affichera une invite « + ». Cela
signifie que vous n’avez pas fini de saisir une commande complète. This
is because you have not ‘closed’ a parenthesis or quotation, i.e. you
don’t have the same number of left-parentheses as right-parentheses, or
the same number of opening and closing quotation marks. Lorsque cela se
produit et que vous pensez avoir fini de taper votre commande, cliquez
dans la fenêtre de la console et appuyez sur « Échap » ; cela annulera
la commande incomplète et vous ramènera à l’invite
>.
Comment en savoir plus pendant et après le cours ?
Le matériel que nous aborderons au cours de ce cours vous donnera un premier aperçu de la façon dont vous pouvez utiliser R pour analyser des données pour votre propre recherche. Cependant, vous devrez en apprendre davantage pour effectuer des opérations avancées telles que nettoyer votre ensemble de données, utiliser des méthodes statistiques, ou créer de superbes graphiques[^dans ce cours]. La meilleure façon de devenir compétent et efficace en R, comme avec tout autre outil, est de l’utiliser pour répondre à vos questions de recherche réelles. En tant que débutant, il peut sembler intimidant de devoir écrire un script à partir de zéro, et étant donné que de nombreuses personnes rendent leur code disponible en ligne, modifiant le code existant pour répondre à vos objectifs. cela pourrait vous permettre de démarrer plus facilement.

Cherche de l’aide
Utilisez l’interface d’aide intégrée de RStudio pour rechercher plus d’informations sur les fonctions R.
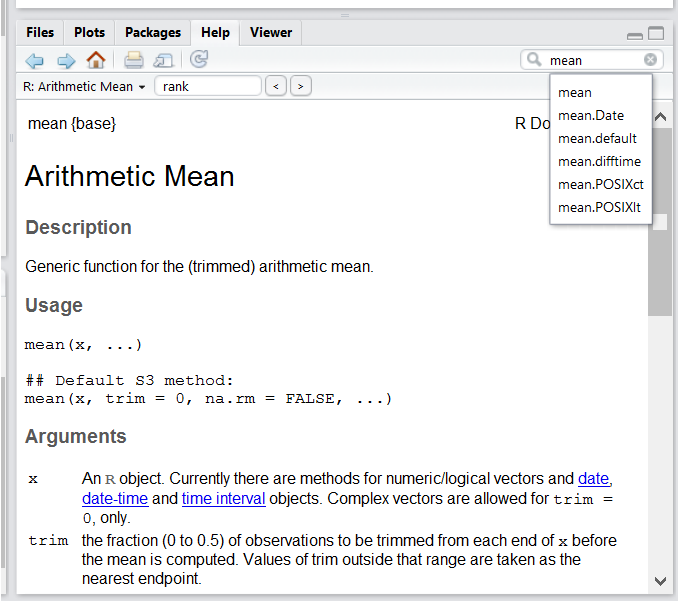
L’un des moyens les plus rapides d’obtenir de l’aide consiste à utiliser l’interface d’aide RStudio . Ce panneau par défaut se trouve dans le panneau inférieur droit de RStudio. Comme le montre la capture d’écran, en tapant le mot “Mean”, RStudio essaie également de donner un certain nombre de suggestions qui pourraient vous intéresser . La description s’affiche alors dans la fenêtre d’affichage .
Je connais le nom de la fonction que je souhaite utiliser, mais je ne sais pas comment l’utiliser
Si vous avez besoin d’aide avec une fonction spécifique, disons
barplot(), vous pouvez taper :
R
?barplot
Si vous avez juste besoin de vous rappeler les noms des arguments, vous pouvez utiliser :
R
args(lm)
Je veux utiliser une fonction qui fait X, il doit y avoir une fonction pour ça mais je ne sais pas laquelle…
Si vous recherchez une fonction pour effectuer une tâche
particulière, vous pouvez utiliser la fonction
help.search(), qui est appelée par le double point
d’interrogation ??. Cependant, cela ne recherche dans les
packages installés que les pages d’aide avec une correspondance avec
votre demande de recherche.
R
??kruskal
Si vous ne trouvez pas ce que vous cherchez, vous pouvez utiliser le site Web rdocumentation.org qui recherche dans les fichiers d’aide de tous les forfaits disponibles.
Enfin, une recherche générique sur Google ou sur Internet “R <task>” vous enverra souvent soit à la documentation du package appropriée, soit à un forum utile où quelqu’un d’autre a déjà posé votre question.
Je suis coincé… Je reçois un message d’erreur que je ne comprends pas
Commencez par rechercher le message d’erreur sur Google. Cependant, cela ne fonctionne pas toujours très bien car souvent, les développeurs de packages s’appuient sur la détection d’erreurs fournie par R. Vous vous retrouvez avec des messages d’erreur généraux qui pourraient ne pas être très utiles pour diagnostiquer un problème. problème (par exemple “indice hors limites”). Si le message est très générique, vous pouvez également inclure le nom de la fonction ou du package que vous utilisez dans votre requête.
Cependant, vous devriez vérifier Stack Overflow. Recherchez en
utilisant la balise [r]. La plupart des questions ont déjà
reçu une réponse, mais le défi consiste à utiliser les bons mots dans la
recherche pour trouver les réponses :
http://stackoverflow.com/questions/tagged/r
The Introduction to R can also be dense for people with little programming experience but it is a good place to understand the underpinnings of the R language.
La FAQ R est dense et technique mais elle regorge d’informations utiles.
Demander de l’aide
La clé pour recevoir de l’aide de quelqu’un est qu’il comprenne rapidement votre problème. Vous devez faire en sorte qu’il soit aussi simple que possible d’identifier où pourrait se situer le problème.
Essayez d’utiliser les mots corrects pour décrire votre problème. Par exemple, un package n’est pas la même chose qu’une bibliothèque. La plupart des gens comprendront ce que vous vouliez dire, mais d’autres ont des sentiments très forts à propos de la différence de sens. Le point clé est que cela peut rendre les choses déroutantes pour les personnes qui essaient de vous aider. Soyez aussi précis que possible lorsque vous décrivez votre problème.
Si possible, essayez de réduire ce qui ne fonctionne pas à un simple *exemple reproductible *. Si vous pouvez reproduire le problème en utilisant un très petit cadre de données au lieu de celui de 50 000 lignes et 10 000 colonnes, fournissez le petit avec la description de votre problème. Le cas échéant, essayez de généraliser ce que vous faites afin que même les personnes qui ne font pas partie de votre domaine puissent comprendre la question. Par exemple, au lieu d’utiliser un sous-ensemble de votre ensemble de données réel, créez un petit (3 colonnes, 5 lignes) générique. Pour plus d’informations sur la façon d’écrire un exemple reproductible, voir cet article de Hadley Wickham.
Pour partager un objet avec quelqu’un d’autre, s’il est relativement
petit, vous pouvez utiliser la fonction dput(). Il produira
du code R qui peut être utilisé pour recréer exactement le même objet
que celui en mémoire :
R
## iris is an example data frame that comes with R and head() is a
## function that returns the first part of the data frame
dput(head(iris))
SORTIE
structure(list(Sepal.Length = c(5.1, 4.9, 4.7, 4.6, 5, 5.4),
Sepal.Width = c(3.5, 3, 3.2, 3.1, 3.6, 3.9), Petal.Length = c(1.4,
1.4, 1.3, 1.5, 1.4, 1.7), Petal.Width = c(0.2, 0.2, 0.2,
0.2, 0.2, 0.4), Species = structure(c(1L, 1L, 1L, 1L, 1L,
1L), levels = c("setosa", "versicolor", "virginica"), class = "factor")), row.names = c(NA,
6L), class = "data.frame")If the object is larger, provide either the raw file (i.e., your CSV file) with your script up to the point of the error (and after removing everything that is not relevant to your issue). Alternativement, en particulier si votre question n’est pas liée à un bloc de données, vous pouvez enregistrer n’importe quel objet R dans un fichier[^export] :
R
saveRDS(iris, file="/tmp/iris.rds")
Le contenu de ce fichier n’est cependant pas lisible par l’homme et
ne peut pas être publié directement sur Stack Overflow. Au lieu de cela,
il peut être envoyé à quelqu’un par email qui pourra le lire avec la
commande readRDS() (ici, suppose que le fichier téléchargé
se trouve dans un dossier Téléchargements dans le
répertoire personnel de l’utilisateur) :
R
some_data <- readRDS(file="~/Downloads/iris.rds")
Dernier point, mais non le moindre, incluez toujours la
sortie de sessionInfo() car elle fournit des
informations critiques sur votre plate-forme, les versions de R et les
packages que vous utilisez. utilisation, et d’autres informations qui
peuvent être très utiles pour comprendre votre problème.
R
sessionInfo()
SORTIE
R version 4.4.1 (2024-06-14)
Platform: x86_64-pc-linux-gnu
Running under: Ubuntu 22.04.4 LTS
Matrix products: default
BLAS: /usr/lib/x86_64-linux-gnu/blas/libblas.so.3.10.0
LAPACK: /usr/lib/x86_64-linux-gnu/lapack/liblapack.so.3.10.0
locale:
[1] LC_CTYPE=C.UTF-8 LC_NUMERIC=C LC_TIME=C.UTF-8
[4] LC_COLLATE=C.UTF-8 LC_MONETARY=C.UTF-8 LC_MESSAGES=C.UTF-8
[7] LC_PAPER=C.UTF-8 LC_NAME=C LC_ADDRESS=C
[10] LC_TELEPHONE=C LC_MEASUREMENT=C.UTF-8 LC_IDENTIFICATION=C
time zone: Asia/Tokyo
tzcode source: system (glibc)
attached base packages:
[1] stats graphics grDevices utils datasets methods base
loaded via a namespace (and not attached):
[1] compiler_4.4.1 tools_4.4.1 highr_0.11 knitr_1.48
[5] xfun_0.47 evaluate_0.24.0Où demander de l’aide ?
- La personne assise à côté de vous pendant le cours. N’hésitez pas à parler à votre voisin pendant l’atelier, comparer vos réponses, et demander de l’aide.
- Vos collègues amicaux : si vous connaissez quelqu’un avec plus d’expérience que vous, il pourra et voudra peut-être vous aider.
- Stack Overflow : si votre question n’a pas reçu de réponse auparavant et est bien conçue, il y a de fortes chances que vous obteniez un réponse en moins de 5 minutes. N’oubliez pas de suivre leurs directives sur comment poser une bonne question.
- La liste de diffusion R-help : elle est lue par un grand nombre de personnes (dont la plupart des l’équipe principale de R), beaucoup de gens y publient des messages, mais le ton peut être assez sec, et il n’est pas toujours très accueillant pour les nouveaux utilisateurs. Si votre question est valide, vous avez de chances d’obtenir une réponse très rapidement, mais ne vous attendez pas à ce qu’elle vienne avec des visages souriants. Aussi, ici plus qu’ailleurs, veillez à d’utiliser un vocabulaire correct (sinon vous pourriez obtenir une réponse pointant vers une mauvaise utilisation de vos mots plutôt que de répondre à votre question). Vous aurez également plus de succès si votre question concerne une fonction de base plutôt qu’un package spécifique.
- Si votre question concerne un package spécifique, vérifiez s’il
existe une liste de diffusion pour celui-ci. Habituellement, il est
inclus dans le fichier DESCRIPTION du package accessible en utilisant
packageDescription("name-of-package"). Vous pouvez également essayer d’envoyer un e-mail directement à l’auteur du package ou d’ouvrir un ticket sur le référentiel de code (par exemple, GitHub). - Il existe également quelques listes de diffusion thématiques (SIG, phylogénétique, etc…), la liste complète est ici.
Davantage de ressources
Le Guide de publication pour les listes de diffusion R.
-
Comment demander de l’aide R
directives utiles.
Ce billet de blog de Jon Skeet contient des conseils assez complets sur la façon dont pour poser des questions de programmation.
Le package reprex est très utile pour créer des exemples reproductibles lorsque vous demandez de l’aide à . The rOpenSci community call “How to ask questions so they get answered” (Github link and video recording) includes a presentation of the reprex package and of its philosophy.
Forfaits R
Chargement des paquets
Comme nous l’avons vu plus haut, les packages R jouent un rôle
fondamental dans R. Les utilisent les fonctionnalités d’un package, en
supposant qu’il soit installé, il faut d’abord le charger pour pouvoir
l’utiliser . Cela se fait avec la fonction library().
Ci-dessous, nous chargeons ggplot2.
R
library("ggplot2")
Installation des packages
Le référentiel de packages par défaut est The Comprehensive R
Archive Network (CRAN), et tout package disponible sur CRAN peut
être installé avec la fonction install.packages().
Ci-dessous, par exemple, , nous installons le package dplyr
que nous découvrirons plus tard.
R
install.packages("dplyr")
Cette commande installera le package dplyr ainsi que
toutes ses dépendances, c’est à dire tous les packages sur lesquels il
s’appuie pour fonctionner.
Un autre référentiel majeur de packages R est géré par Bioconductor.
Packages
Bioconductor sont gérés et installés à l’aide d’un package dédié, à
savoir BiocManager, qui peut être installé à partir de CRAN
avec
R
install.packages("BiocManager")
Des packages individuels tels que SummarizedExperiment
(nous l’utiliserons plus tard), DESeq2 (pour l’analyse
RNA-Seq) et tout autre de Bioconductor ou CRAN peuvent ensuite être
installés avec BiocManager :: installer.
R
BiocManager::install("SummarizedExperiment")
BiocManager::install("DESeq2")
Par défaut, BiocManager::install() vérifiera également
tous vos packages installés et verra si des versions plus récentes sont
disponibles. S’il y en a, il vous les montrera et vous demandera si vous
souhaitez « Mettre à jour tout/certains/aucun ? [a/s/n] :` et attendez
votre réponse. Bien que vous deviez vous efforcer de disposer des
versions de packages les plus à jour, en pratique, nous vous
recommandons de mettre à jour les packages uniquement lors d’une
nouvelle session R avant le chargement des packages.
Au lieu d’utiliser R directement depuis la console de ligne de commande . Il existe d’autres logiciels qui s’interfacent et intègrent avec R, mais RStudio est particulièrement bien adapté aux débutants tout en proposant de nombreuses fonctionnalités très avancées.↩︎
c’est-à-dire des modules complémentaires qui confèrent à R de nouvelles fonctionnalités, telles que l’analyse de données bioinformatiques.↩︎
Dans ce cours, nous considérons la bioinformatique comme une science des données appliquée aux données biologiques ou bio-médicales.↩︎
Cette personne pourrait être, et sera très probablement votre futur moi, quelques mois ou années après que les analyses aient été effectuées.↩︎
