Content from イントロダクションとセットアップ
Last updated on 2025-09-23 | Edit this page
Overview
Questions
- このレッスンのために正しいバージョンのRを使用していますか?
- なぜ私のRのバージョンが重要なのですか?
- このレッスンで使用されるファイルをどうやって取得しますか?
Objectives
- 受講者がこのレッスンの内容を正確に再現するために、正しいバージョンのRを使用していることを確認してください。
- このレッスンのサンプルファイルをダウンロードしてください。
Rのバージョン
このレッスンはR version 4.4.3 (2025-02-28)を使用して開発およびテストされました。
RStudioを起動し、あなたが4.4.xのRバージョンを使用しているか確認してください。xは任意のパッチバージョン、例えば4.4.3です。
R
R.version.string
OUTPUT
[1] "R version 4.4.3 (2025-02-28)"これは重要です。なぜなら、Bioconductorは現在のセッションで実行されているRのバージョンを使用して、現在のRセッションに関連付けられたRライブラリにインストールできるBioconductorパッケージのバージョンを決定するからです。 このレッスンに従いながら異なるバージョンのRを使用すると、予期しない結果が生じる可能性があります。
ファイルをダウンロードする
このレッスンのいくつかのエピソードは、参加者がダウンロードする必要があるサンプルファイルに依存しています。
以下のコードを実行して、現在の作業ディレクトリにdataというフォルダーをプログラムで作成し、そのフォルダー内にレッスンファイルをダウンロードします。
R
dir.create("data", showWarnings = FALSE)
download.file(
url = "https://raw.githubusercontent.com/Bioconductor/bioconductor-teaching/master/data/TrimmomaticAdapters/TruSeq3-PE-2.fa",
destfile = "data/TruSeq3-PE-2.fa"
)
download.file(
url = "https://raw.githubusercontent.com/Bioconductor/bioconductor-teaching/master/data/ActbGtf/actb.gtf",
destfile = "data/actb.gtf"
)
download.file(
url = "https://raw.githubusercontent.com/Bioconductor/bioconductor-teaching/master/data/ActbOrf/actb_orfs.fasta",
destfile = "data/actb_orfs.fasta"
)
download.file(
url = "https://raw.githubusercontent.com/Bioconductor/bioconductor-teaching/devel/data/SummarizedExperiment/counts.csv",
destfile = "data/counts.csv"
)
download.file(
url = "https://raw.githubusercontent.com/Bioconductor/bioconductor-teaching/devel/data/SummarizedExperiment/gene_metadata.csv",
destfile = "data/gene_metadata.csv"
)
download.file(
url = "https://raw.githubusercontent.com/Bioconductor/bioconductor-teaching/devel/data/SummarizedExperiment/sample_metadata.csv",
destfile = "data/sample_metadata.csv"
)
注意
理想的には、参加者は新しいRStudioプロジェクトを作成し、そのプロジェクトのサブディレクトリにレッスンファイルをダウンロードしたいと思うかもしれません。
RStudioプロジェクトを使用すると、作業ディレクトリがそのプロジェクトのルートディレクトリに設定されます。 その結果、コードはそのルートディレクトリに対して実行されるため、ファイルからデータをインポート/エクスポートするために絶対パスを使用する必要がない場合がよくあります。
- 参加者は、このレッスンで説明されているBioconductorパッケージのバージョンをインストールし、その出力を正確に再現するには、正しいバージョンのRを使用する必要があります。
- このレッスンで使用されるファイルは、Rセッションから簡単にアクセスできるローカルパスにダウンロードする必要があります。
Content from Bioconductorの概要
Last updated on 2025-09-23 | Edit this page
Overview
Questions
- Bioconductorプロジェクトには何が含まれていますか?
- BioconductorプロジェクトはCRANリポジトリとどのように関連していますか?
- Bioconductorのパッケージを効果的に使用するにはどうすればいいですか?
- Bioconductorコミュニティに参加し、コミュニケーションをとるにはどうすればいいですか?
Objectives
- Bioconductorプロジェクトについて全体を説明する。
- RエコシステムにおけるBioconductorプロジェクトの全体像を把握する。
- Bioconductorプロジェクトの今後の更新情報を監視するための情報源を特定する。
Bioconductorとは何ですか?
Bioconductorの簡単な歴史
Bioconductorプロジェクトは2001年秋に始まり、計算生物学とバイオインフォマティクスのための拡張可能なソフトウェアの共同制作のためのイニシアティブとして立ち上げられました (Gentleman, Carey, Bates, Bolstad, Dettling, Dudoit, Ellis, Gautier, Ge, Gentry, Hornik, Hothorn, Huber, Iacus, Irizarry, Leisch, Li, Maechler, Rossini, Sawitzki, Smith, Smyth, Tierney, Yang, and Zhang, 2004)。 プロジェクトの初めから、記載された使命は、大規模データセットと厳密かつ堅牢に設計された実験の技術的成果物のための統計分析と理解のためのツールを開発することでした。 統計分析を超えて、統計結果の解釈は、生物学的文脈、視覚化、および再現性を提供するパッケージによってサポートされています。
年月が経つにつれて、Bioconductorプロジェクトに貢献したソフトウェアパッケージは、マイクロアレイから単一細胞ゲノミクスまで、さまざまな高スループット技術の進化と出現を反映してきました。シーケンシング実験の多くのバリエーション(例:RNA-seq、ChIP-seq、DNA-seq)、分析(例:塩基配列のばらつき、コピー数の変動、単一ヌクレオチド多型)、およびデータモダリティ(例:フローサイトメトリー、プロテオミクス、顕微鏡および画像分析)を通じて。
重要なのは、プロジェクトは新しい統計テストと方法論を実装するソフトウェアパッケージをリリースするだけでなく、分子注釈と実験データセットのデータベースへのアクセスを提供する多様なパッケージタイプを生産してきたことです。
Bioconductorプロジェクトは、夏に北アメリカで開催される年次会議でクライマックスに達し、地域会議はいずれもヨーロッパ、アジア、北アメリカでのネットワーキングに優れた機会を提供します。 このプロジェクトは多様で包括的なコミュニティの促進に取り組んでおり、行動規範を含む、行動規範委員会によって施行されています。
Major Bioconductor milestonesと技術の進歩のタイムライン。 タイムラインの上部には、主要イベントの初めの発生が示されています。 タイムライン内では、コアインフラを提供するパッケージの名前がリリース日を示しています。 タイムラインの下部では、主要な技術的進歩がBioconductorプロジェクトの進化を時系列で文脈化します。
科学プロジェクト
元の出版物では、プロジェクトの設立時の目的と方法が説明されています Gentleman, Carey, Bates et al. (2004)。
Huber, Carey, Gentleman, Anders, Carlson, Carvalho, Bravo, Davis, Gatto, Girke, Gottardo, Hahne, Hansen, Irizarry, Lawrence, Love, MacDonald, Obenchain, Oles, Pages, Reyes, Shannon, Smyth, Tenenbaum, Waldron, and Morgan (2015)は、コアインフラとケーススタディの説明を含むプロジェクトの進展を、ユーザーと開発者の両方の視点から説明しています。
Amezquita, Lun, Becht, Carey, Carpp, Geistlinger, Marini, Rue-Albrecht, Risso, Soneson, Waldron, Pages, Smith, Huber, Morgan, Gottardo, and Hicks (2020)は、単一細胞ゲノミクス技術の目覚めに続くプロジェクトのさらなる発展をレビューしています。
多くの出版物や書籍の章がBioconductorプロジェクトを引用しており、最近の例はBioconductorウェブサイトにリストされています。
パッケージリポジトリ
概要およびCRANとの関係
間違いなく、ソフトウェアパッケージはBioconductorプロジェクトの最も知られている側面です。 2001年に設立されて以来、リポジトリは時間とともに成長し、数千のパッケージをホストするようになりました。
Bioconductorプロジェクトは、Rパッケージが主にバイオインフォマティクスと計算生物学の分析を対象にしている既存のCRANリポジトリを拡張しました。
Bioconductorリリースサイクル
Bioconductorプロジェクトは、CRANリポジトリのパッケージ管理インフラを拡張して、ユーザーのレベルでのパッケージの展開と管理をより良くサポートしました (Gentleman, Carey, Bates et al., 2004)。 特に、Bioconductorプロジェクトは、6か月のリリースサイクル(通常は4月と10月頃)を特徴としており、Bioconductorリポジトリ内のすべてのパッケージの現在のバージョンのスナップショットが特定のRバージョン用に指定されています。 R自体は年に1回リリースされ(通常は4月頃)、Rの各リリースごとに2つのBioconductorパッケージの互換性のあるリリースが利用可能になります。
そのため、Bioconductorパッケージ開発者は、次のBioconductorプロジェクトのリリースに関連付けられるRのバージョンを常に使用する必要があります。 これは、10月から4月の間はRの開発バージョンを使用し、4月から10月の間はRのリリースバージョンを使用することを意味します。
重要なのは、厳格なBioconductorリリースサイクルが、ユーザーが互いに非常に遠く離れたバージョンのパッケージをインストールできないようにし、テストを受ける可能性が低いパッケージをインストールできなくすることです。 この慣行は、CRANとBioconductorの両方のパッケージの開発サイクルを反映しており、同時のパッケージが自動化されたシステムによって定期的にテストされ、パッケージ依存関係内の最新のソフトウェア更新が下流のパッケージに壊れることがないことを保証します。これにより、パッケージのメンテナが自分のソフトウェアを更新することを促します。
各Bioconductorリリースの前に、必要な自動テストスイートを通過しないパッケージは廃止され、その後リポジトリから削除されます。 これにより、各Bioconductorリリースは、相互に互換性があり、追跡可能で、関連するRのバージョンで機能するパッケージのスイートを提供することが保証されます。
選択されたBioconductorおよびRのバージョンのリリース日タイムライン。 タイムラインの上部には、Rプロジェクトのバージョンとおおよそのリリース日が示されています。 タイムラインの下部には、Bioconductorプロジェクトのバージョンとリリース日が示されています。 出典: Bioconductor。
パッケージタイプ
パッケージは、主に4つの主要カテゴリに分けられます。
- ソフトウェア
- 注釈データ
- 実験データ
- ワークフロー
ソフトウェアパッケージ自体は、データを保存してアクセスするためのインフラ(すなわち、クラス)を提供するパッケージと、そのデータ構造に保存されたデータを処理するための方法論的ツールを提供するパッケージに再分割できます。 この構造と分析の分離はBioconductorプロジェクトの中心にあり、新しい方法論的ソフトウェアパッケージの開発者が可能な限り既存のデータコンテナを再利用することを奨励し、異なるデータ構造間の学習と変換の必要なく、ユーザーが代替ワークフローをより容易に試すことができるようにします。
注釈データパッケージは、多様なゲノム注釈の自己完結型データベース(例:遺伝子識別子、生物学的経路)を提供します。
さまざまなコレクションの注釈パッケージはBioconductorプロジェクトに存在します。
それらは、それぞれの命名パターンや含まれている情報によって識別できます。
たとえば、いわゆるOrgDbパッケージ(例としてorg.Hs.eg.dbパッケージ)は、異なるタイプの遺伝子識別子および経路データベースをマッピングする情報を提供します。
いわゆるEnsDb(例としてEnsDb.Hsapiens.v86)パッケージは、BioconductorパッケージのEnsembl注釈の個別バージョンをカプセル化します。いわゆるTxDbパッケージ(例としてTxDb.Hsapiens.UCSC.hg38.knownGene)は、個別バージョンのUCSC遺伝子注釈テーブルをカプセル化します。
実験データパッケージは、ソフトウェアパッケージの開発者が、パッケージのビネットにおいて自分のパッケージの使用を示すためによく使用される自己完結型データセットを提供します。
最後に、ワークフローパッケージは、他のパッケージの結合使用を示すビネットのコレクションを提供しますが、新しいソースコードや機能は提供しません。
チャレンジ: Bioconductorウェブサイト
Bioconductorウェブサイトはhttps://bioconductor.org/からアクセス可能です。
ウェブサイトをブラウズして、次の質問に答える情報を見つけてください:
- 現在のBioconductorプロジェクトのリリースにはいくつのパッケージが含まれていますか?
- この数には各タイプのパッケージがいくつ含まれていますか?
以下の解決策には、執筆時点で有効だった数字(Bioconductorリリース3.13)が含まれています; Bioconductorプロジェクトの将来のリリースでは、数字が異なることは避けられません。
- https://bioconductor.org/のページで、「インストール」というセクションを読むことができます。
Bioconductorリリース3.13で利用可能な2042ソフトウェアパッケージを発見する。
-
https://bioconductor.org/の「ニュース」セクションで、「Bioconductor
Bioc
X.Yリリース」というリンクをクリックしてください(X.Yは、この演習を通じて行くときの現在のBioconductorリリースのバージョン)。 リンクされたページでは、以下の内容を読むことができます:
Bioconductor 3.13を発表できることを嬉しく思います。これは2042ソフトウェアパッケージ、406の実験データパッケージ、965の注釈パッケージ、29のワークフローから構成されています。
新しいソフトウェアパッケージは133、新しいデータ実験パッケージは22、新しい注釈パッケージは7、新しいワークフローは1、新しい書籍はなく、多くの更新と既存パッケージの改善があります。Bioconductor 3.13はR 4.1.0と互換性があり、Linux、32ビットおよび64ビットのWindows、macOS 10.14.6 Mojave以上でサポートされています。 このリリースには更新されたBioconductor Dockerコンテナが含まれます。
biocViewsを使用したパッケージ分類
Bioconductorプロジェクトは、Bioconductorパッケージを分類し、Bioconductorウェブサイトでのテーマ別検索を介してその発見を容易にするために、制御語彙からの用語の集合であるbiocViewsを使用します。
各Bioconductorパッケージは、パッケージのタイプと機能を説明するために選ばれた制御語彙からの小さな用語セットでタグ付けされます。 用語は最初にパッケージの著者によって選択され、次にパッケージレビューや制御語彙の更新中に精査されます。
チャレンジ
BioconductorのすべてのパッケージのリストをBioconductor biocViewsのウェブページで訪れてください。 左上の「Autocomplete biocViews search」ボックスを使用して、カテゴリ別にパッケージをフィルタリングし、個々の用語を展開および収縮させてソフトウェアパッケージのグラフを探ります。
さらに進む
BioconductorパッケージbiocViewsは、制御語彙のインフラをサポートおよび管理するために使用されます。 また、用語のリストをプログラム的に検査し、グラフとしてその関係によってサブセットを取得するためにも使用されます。
さらに、BiocPkgToolsパッケージを使用して、異なるbiocViewsのもとにあるパッケージをブラウズすることができます (Su, Carey, Shepherd, Ritchie, Morgan, and Davis, 2019)。
パッケージの相互運用性
Bioconductorの哲学の中心には、相互運用性の概念があります。 つまり、パッケージが同じデータ構造で動作する能力です。 重要なのは、相互運用性はユーザーと開発者の両方に利益をもたらすことです。
ユーザーは、複数のパッケージを組み合わせた任意の複雑なワークフローを書くことがより簡単になります。 同じデータ構造に基づくパッケージがあるため、ユーザーはワークフローの実際のステップに最大限注意を払い、各パッケージに特有の異なるデータ構造間で行う複雑かつエラーが発生しやすい変換に費やす時間を最小限に抑えることができます。 同様に、新しいパッケージの開発者は、情報の保存、検証、およびインデックスを行うために堅牢で信頼されるインフラを提供する既存のデータ構造を借用し、革新的な機能の実装に焦点を当てることができます。
最終的に、下図は、さまざまなBioconductorパッケージとベースRパッケージがどのように組み合わされて、シーケンシングデータをRセッションにインポートし、データと結果の注釈、統合、視覚化を実行するさまざまな分析を実行できるかを示しています。
シーケンシングエコシステム 主要データ処理ステップ(青)と関連ソフトウェアパッケージ(ピンク)が、さまざまなタイプのゲノム分析の典型的なワークフローの文脈で一覧表示されます。
ワークフローのステップとソフトウェアパッケージの連続的な関係は、完全なエンドツーエンドのワークフローを構成するためのソフトウェアパッケージ間の相互運用性の重要性を示しています。 ワークフローのステップとソフトウェアパッケージの逐次関係は、完全なエンドツーエンドのワークフローを構成するために、ソフトウェアパッケージ間の相互運用性の重要性を示しています。
会議、コース、ワークショップ
Bioconductorコミュニティは、年中世界中の数多くのイベントを定期的に開催しています。 例えば:
- 北アメリカでの年次BioCサマー会議
- 冬の地域会議(例:BioC Europe、BioC Asia)
- サマー学校(例:CSAMA)
- すべてのコミュニティメンバーが参加できるオンラインミーティング(例:Bioconductor Developers Forum)
コース教材は、これらのイベントの各後に定期的にBioconductorウェブサイトにアップロードされます。 特に、オンライン書籍はコミュニティメンバーによって開発および維持されています。
BioconductorのYouTubeチャンネルは、会議の発表のビデオ録画(講演やワークショップを含む)を公開するために使用され、通常のBioconductor開発者フォーラムの版(リンクが必要)も含まれています。
貢献しよう!
年を通じての会議の典型的なサイクルを示すことができれば素晴らしいでしょう。例えば、
- 7月末頃の北アメリカでのBioC会議
- 12月頃のヨーロッパでのEuroBioC会議
- 11月頃のアジアでのBioCAsia会議
オンラインコミュニケーションチャネル
サポートサイト
Bioconductorのサポートサイトは、ユーザーと開発者が自由にコミュニケーションをとるためのプラットフォームを提供し(Bioconductorの行動規範に従って)、パッケージに関する問題やベストプラクティスに関する概念的な質問について議論します。
Slackワークスペース
BioconductorのSlackワークスペースは、すべてのコミュニティメンバーが参加(無料)して、迅速な相互作用を行うことができるオープンスペースです。 現在、「Pro」プランの価格設定は、コア資金によって支援されています。
- 無制限のメッセージアーカイブ
- 無制限のアプリ
- 画面共有付きのグループビデオ通話
- Slack Connectを使用して他の組織と安全に作業する
さまざまなトピックについて議論するために多くのチャンネルが作成されており、コミュニティメンバーはそのチャンネルに自由に参加することができ、また新しいチャンネルを作成して新しいトピックについて議論することもできます。
重要な発表は#generalチャンネルに投稿されます。
注意
ユーザーは、Bioconductorのサポートサイトを使用して、より広いコミュニティに関連する問題を提起することを奨励されています。
Slack
ワークスペースは、ライブディスカッションに最も便利で、広く参加しているチャンネル(例:#general)は適度に使用する必要があります。
開発者メーリングリスト
bioc-devel@r-project.org メーリングリストは、パッケージ開発者間のコミュニケーションや Bioconductor コアチームからの発表に使用されます。
科学技術コミュニティ
- 科学諮問委員会 (SAB) 年に1度の会合、プロジェクトアドバイザーとして活動する外部および内部のリーダー。 任期の制限なし。
- 技術技術委員会 (TAB)。 月に一度、プロジェクトのコアインフラストラクチャと科学的方向性の技術的側面を検討します。 メンバーは 15 人、任期は 3 年。 年に一度の公開選挙でメンバーを交代します。 現在の役員は、ビンス・ケアリー(議長)、リーヴァイ・ウォルドロン(副議長)、シャーロット・ソネソン(書記)。
- コミュニティ諮問委員会 (CAB) 月に一度、地域社会への outreach、イベント、教育、トレーニングを考慮します。 メンバーは 15 人、任期は 3 年。 年に一度の公開選挙でメンバーを交代します。 現在の役員は、エイディン・カルハン(議長)、マット・リッチー(共同議長)、ローリ・カーン(書記)。
- 行動規範委員会
注意
TAB/CABの少なくとも1名のメンバーが両方に参加し、ボードのコミュニケーションを確保するための連絡役を果たします。
参考文献
[1] R. A. Amezquita, A. T. L. Lun, E. Becht, et al. “Orchestrating single-cell analysis with Bioconductor”. In: Nat Methods 17.2 (2020), pp. 137-145. ISSN: 1548-7105 (Electronic) 1548-7091 (Linking). DOI: 10.1038/s41592-019-0654-x. https://www.ncbi.nlm.nih.gov/pubmed/31792435.
[2] R. C. Gentleman, V. J. Carey, D. M. Bates, et al. “Bioconductor: open software development for computational biology and bioinformatics”. In: Genome Biol 5.10 (2004), p. R80. ISSN: 1474-760X (Electronic) 1474-7596 (Linking). DOI: 10.1186/gb-2004-5-10-r80. https://www.ncbi.nlm.nih.gov/pubmed/15461798.
[3] W. Huber, V. J. Carey, R. Gentleman, et al. “Orchestrating high-throughput genomic analysis with Bioconductor”. In: Nat Methods 12.2 (2015), pp. 115-21. ISSN: 1548-7105 (Electronic) 1548-7091 (Linking). DOI: 10.1038/nmeth.3252. https://www.ncbi.nlm.nih.gov/pubmed/25633503.
[4] S. Su, V. Carey, L. Shepherd, et al. “BiocPkgTools: Toolkit for mining the Bioconductor package ecosystem [version 1; peer review: 2 approved, 1 approved with reservations]”. In: F1000Research 8.752 (2019). DOI: 10.12688/f1000research.19410.1.
- R パッケージは Bioconductor プロジェクトの一側面に過ぎません。
- Bioconductor プロジェクトは CRAN リポジトリを拡張し、補完します。
- 異なる種類のパッケージは、ソフトウェアだけでなく、アノテーション、実験データも提供し、統合されたワークフローで複数のパッケージの使用方法を示します。
- Bioconductor パッケージ間の相互運用性は、統合ワークフローの作成を容易にし、ユーザーの認知的負担を最小限に抑えます。
- コースや会議の教育資料はアーカイブされ、Bioconductor ウェブサイトと YouTube チャンネルでアクセス可能です。
- 異なるコミュニケーションのチャネルを使用して、コミュニティのメンバーが会話し、ユーザーとパッケージ開発者の両方として互いに助け合います。
- Bioconductor プロジェクトは、科学、技術、諮問の各委員会および行動規範委員会によって運営されています。
Content from Bioconductor パッケージのインストール
Last updated on 2025-09-23 | Edit this page
Overview
Questions
- Bioconductor パッケージをどのようにインストールしますか?
- インストールしたパッケージの新しいバージョンが利用可能かどうかを確認するにはどうすればよいですか?
- Bioconductor パッケージをどのように更新しますか?
- Bioconductor リポジトリから利用可能なパッケージの名前をどのように調べますか?
Objectives
- BiocManager をインストールします。
- Bioconductor パッケージをインストールします。
BiocManager
BiocManager パッケージは Bioconductor パッケージ リポジトリへのエントリ ポイントです。 技術的には、これは CRAN リポジトリで配布される唯一の Bioconductor パッケージです。
Bioconductor パッケージを安全にインストールし、利用可能な更新を確認するための関数を提供します。
パッケージがインストールされると、BiocManager::install()
関数を使用して Bioconductor
リポジトリからパッケージをインストールできます。
この関数は、Bioconductor
リポジトリ内にパッケージが見つからない場合、他のリポジトリ (例: CRAN)
からパッケージをインストールすることも可能です。
BiocManager パッケージは CRAN
リポジトリから利用可能で、Bioconductor
リポジトリからパッケージをインストールするために使用されます。
基本 R パッケージ utils の install.packages()
関数を使用して、CRAN リポジトリで配布される BiocManager
パッケージをインストールできます。
その後、BiocManager::install() 関数を使用して、Bioconductor
リポジトリで利用可能なパッケージをインストールできます。
特に、BiocManager::install() 関数は、Bioconductor
リポジトリでパッケージが見つからない場合、CRAN
リポジトリにフォールバックします。
以下のコードを使用してパッケージをインストールします。
R
install.packages("BiocManager")
さらに進む
基本 R インストールの一部ではない多くのパッケージも、さまざまなリポジトリからパッケージをインストールするための関数を提供します。 たとえば:
devtools::install()remotes::install_bioc()remotes::install_bitbucket()remotes::install_cran()remotes::install_dev()remotes::install_github()remotes::install_gitlab()remotes::install_git()remotes::install_local()remotes::install_svn()remotes::install_url()renv::install()
これらの関数は、このレッスンの範囲を超えており、特定の動作について充分な知識を持って注意して使用する必要があります。
一般的な推奨事項は、Bioconductor
パッケージの正しいバージョン管理を確保するために、他のインストールメカニズムの上に
BiocManager::install() を使用することです。
Bioconductor のリリースと現在のバージョン
BiocManager
パッケージがインストールされると、BiocManager::version()
関数は現在の R セッションでアクティブな Bioconductor
プロジェクトのバージョン (すなわちリリース) を表示します。
R
BiocManager::version()
OUTPUT
[1] '3.19'R と Bioconductor パッケージの正しいバージョンを使用することは、再現性の重要な側面です。 BiocManager パッケージは、現在のセッションで実行されている R のバージョンを使用して、現在の R ライブラリにインストールできる Bioconductor パッケージのバージョンを決定します。
Bioconductor プロジェクトは、毎年 4 月と 10 月の 2 回のリリースを行います。 Bioconductor の 4 月のリリースは、R の年次リリースと一致します。 Bioconductor の 10 月のリリースは、次のリリース(4 月)まで、その年次サイクルのために同じバージョンの R を引き続き使用します。
選択された Bioconductor と R のバージョンのリリース日程。 タイムラインの上部には R プロジェクトのバージョンとおおよそのリリース日が示されています。 タイムラインの下部には Bioconductor プロジェクトのバージョンとリリース日が示されています。 出典: Bioconductor。
各 6 か月のパッケージ開発サイクル中に、Bioconductor は次のリリースサイクルで利用可能になる R のバージョンとの互換性をチェックします。 その後、新しい Bioconductor リリースが生成されるたびに、Bioconductor リポジトリ内のすべてのパッケージのバージョンがインクリメントされます。これには Bioconductor プロジェクトのバージョンを決定する BiocVersion パッケージも含まれます。
R
packageVersion("BiocVersion")
OUTPUT
[1] '3.19.1'これは、前のリリース以降まったく更新されていないパッケージにも当てはまります。 各パッケージの新しいバージョンは、対応する R のバージョン用に指定されています。 言い換えれば、そのパッケージのバージョンは、正しいバージョンの R を使用する R セッションでのみインストールおよびアクセスできます。 このバージョンのインクリメントは、Bioconductor パッケージの各バージョンを Bioconductor プロジェクトのユニークなリリースに関連付けるために不可欠です。
4 月のリリースに続いて、ユーザーは新しい Bioconductor パッケージのリリースを利用するために新しいバージョンの R をインストールする必要があります。
一方、10 月には、ユーザーは新しい Bioconductor
パッケージのリリースを利用するために同じバージョンの R
を使用し続けることができます。 ただし、4 月の Bioconductor リリースから
10 月の Bioconductor リリースに R
ライブラリを更新するには、BiocManager::install()
関数を呼び出し、version オプションとして正しい Bioconductor
のバージョンを指定する必要があります。たとえば:
R
BiocManager::install(version = "3.14")
これは 1 回だけ実行する必要があります。BiocVersion パッケージは対応するバージョンに更新され、現在の R ライブラリで使用している Bioconductor のバージョンを示します。
更新を確認する
BiocManager::valid()
関数は、ユーザーライブラリに現在インストールされているパッケージのバージョンを検査し、いずれかのパッケージの新しいバージョンが
Bioconductor リポジトリで利用可能かどうかを確認します。
すべてが最新の場合、関数は単に TRUE を返します。
R
BiocManager::valid()
WARNING
Warning: 15 packages out-of-date; 0 packages too newOUTPUT
* sessionInfo()
R version 4.4.3 (2025-02-28)
Platform: x86_64-pc-linux-gnu
Running under: Ubuntu 22.04.5 LTS
Matrix products: default
BLAS: /usr/lib/x86_64-linux-gnu/openblas-pthread/libblas.so.3
LAPACK: /usr/lib/x86_64-linux-gnu/openblas-pthread/libopenblasp-r0.3.20.so; LAPACK version 3.10.0
locale:
[1] LC_CTYPE=C.UTF-8 LC_NUMERIC=C LC_TIME=C.UTF-8
[4] LC_COLLATE=C.UTF-8 LC_MONETARY=C.UTF-8 LC_MESSAGES=C.UTF-8
[7] LC_PAPER=C.UTF-8 LC_NAME=C LC_ADDRESS=C
[10] LC_TELEPHONE=C LC_MEASUREMENT=C.UTF-8 LC_IDENTIFICATION=C
time zone: UTC
tzcode source: system (glibc)
attached base packages:
[1] stats graphics grDevices utils datasets methods base
other attached packages:
[1] BiocStyle_2.32.1
loaded via a namespace (and not attached):
[1] digest_0.6.37 assertthat_0.2.1 R6_2.6.1
[4] fastmap_1.2.0 xfun_0.52 magrittr_2.0.4
[7] glue_1.8.0 knitr_1.50 sandpaper_0.17.2.9000
[10] htmltools_0.5.8.1 rmarkdown_2.29 lifecycle_1.0.4
[13] xml2_1.4.0 ps_1.9.1 cli_3.6.5
[16] processx_3.8.6 callr_3.7.6 vctrs_0.6.5
[19] renv_1.1.5 withr_3.0.2 compiler_4.4.3
[22] purrr_1.1.0 tools_4.4.3 tinkr_0.3.0
[25] evaluate_1.0.4 yaml_2.3.10 BiocManager_1.30.26
[28] pegboard_0.7.9 rlang_1.1.6
Bioconductor version '3.19'
* 15 packages out-of-date
* 0 packages too new
create a valid installation with
BiocManager::install(c(
"bookdown", "curl", "dbplyr", "evaluate", "httr2", "magrittr", "pillar",
"purrr", "Rcpp", "renv", "RSQLite", "stringr", "xfun", "XML", "xml2"
), update = TRUE, ask = FALSE, force = TRUE)
more details: BiocManager::valid()$too_new, BiocManager::valid()$out_of_date便利なことに、更新できるパッケージがある場合、関数はそれらのパッケージを更新するために必要なコマンドを生成して表示します。 ユーザーは単にそのコマンドをコピーして R コンソールで実行する必要があります。
古くなったパッケージライブラリの例
以下の例では、BiocManager::valid() 関数は
TRUE を返しませんでした。
その代わりに、アクティブなユーザー
セッションに関する情報が含まれ、ユーザーが古いライブラリ内のすべての古いパッケージを最新バージョンに置き換えるために実行すべき
BiocManager::install()
への正確な呼び出しが表示されます。
> BiocManager::valid()
* sessionInfo()
R バージョン 4.1.0 (2021-05-18)
プラットフォーム: x86_64-apple-darwin17.0 (64-bit)
実行環境: macOS Big Sur 11.6
行列積: デフォルト
LAPACK: /Library/Frameworks/R.framework/Versions/4.1/Resources/lib/libRlapack.dylib
ロケール:
[1] en_GB.UTF-8/en_GB.UTF-8/en_GB.UTF-8/C/en_GB.UTF-8/en_GB.UTF-8
添付された基本パッケージ:
[1] stats graphics grDevices datasets utils methods base
名前空間を介して読み込まれたパッケージ (添付されていない):
[1] BiocManager_1.30.16 compiler_4.1.0 tools_4.1.0 renv_0.14.0
Bioconductor バージョン '3.13'
* 18 パッケージが古くなっています
* 0 パッケージが新しすぎます
以下のコマンドを実行して、有効なインストールを作成します。
BiocManager::install(c(
"cpp11", "data.table", "digest", "hms", "knitr", "lifecycle", "matrixStats", "mime", "pillar", "RCurl",
"readr", "remotes", "S4Vectors", "shiny", "shinyWidgets", "tidyr", "tinytex", "XML"
), update = TRUE, ask = FALSE)
詳細: BiocManager::valid()$too_new, BiocManager::valid()$out_of_date
警告メッセージ:
18 パッケージが古くなっています; 0 パッケージが新しすぎます 具体的には、この例では、メッセージがユーザーに次のコマンドを実行してインストールを最新の状態にするように指示しています。
BiocManager::install(c(
"cpp11", "data.table", "digest", "hms", "knitr", "lifecycle", "matrixStats", "mime", "pillar", "RCurl",
"readr", "remotes", "S4Vectors", "shiny", "shinyWidgets", "tidyr", "tinytex", "XML"
), update = TRUE, ask = FALSE)パッケージ リポジトリを探る
Bioconductor biocViews は、以前のエピソード Introduction to Bioconductor で示されたように、テーマに沿って Bioconductor パッケージの階層的分類をブラウズすることで新しいパッケージを発見する優れた方法です。
さらに、BiocManager::available() 関数は、Bioconductor
および CRAN
リポジトリからインストールできるパッケージ名の完全なリストを返します。
たとえば、BiocManager
を使用してインストールできるパッケージの総数
R
length(BiocManager::available())
OUTPUT
[1] 26347具体的には、現在の Bioconductor リポジトリと検索パス上の他のリポジトリの和集合を次のように表示できます。
R
BiocManager::repositories()
OUTPUT
BioCsoft
"https://bioconductor.org/packages/3.19/bioc"
BioCann
"https://bioconductor.org/packages/3.19/data/annotation"
BioCexp
"https://bioconductor.org/packages/3.19/data/experiment"
BioCworkflows
"https://bioconductor.org/packages/3.19/workflows"
BioCbooks
"https://bioconductor.org/packages/3.19/books"
carpentries
"https://carpentries.r-universe.dev"
carpentries_archive
"https://carpentries.github.io/drat"
CRAN
"https://cloud.r-project.org" 各リポジトリの URL は Web ブラウザでアクセスでき、そのリポジトリから利用可能なパッケージの完全なリストが表示されます。 たとえば、https://bioconductor.org/packages/3.14/bioc に移動します。
さらに進む
BiocManager::repositories() 関数は、基本関数
available.packages()
と組み合わせて、特定のパッケージリポジトリから利用できるパッケージを照会できます。たとえば、Bioconductor
ソフトウェア パッケージ
リポジトリです。
> db = available.packages(repos = BiocManager::repositories()["BioCsoft"])
> dim(db)
[1] 1948 17
> head(rownames(db))
[1] "a4" "a4Base" "a4Classif" "a4Core" "a4Preproc"
[6] "a4Reporting"便利なことに、BiocManager::available() には
pattern=
引数が含まれ、特に注釈リソースをナビゲートするために便利です
(元のユースケースがその理由です)。
たとえば、マウスモデル生物に対して利用可能なさまざまな 注釈データパッケージ
を次のようにリストできます。
R
BiocManager::available(pattern = "*Mmusculus")
OUTPUT
[1] "BSgenome.Mmusculus.UCSC.mm10" "BSgenome.Mmusculus.UCSC.mm10.masked"
[3] "BSgenome.Mmusculus.UCSC.mm39" "BSgenome.Mmusculus.UCSC.mm8"
[5] "BSgenome.Mmusculus.UCSC.mm8.masked" "BSgenome.Mmusculus.UCSC.mm9"
[7] "BSgenome.Mmusculus.UCSC.mm9.masked" "EnsDb.Mmusculus.v75"
[9] "EnsDb.Mmusculus.v79" "PWMEnrich.Mmusculus.background"
[11] "TxDb.Mmusculus.UCSC.mm10.ensGene" "TxDb.Mmusculus.UCSC.mm10.knownGene"
[13] "TxDb.Mmusculus.UCSC.mm39.knownGene" "TxDb.Mmusculus.UCSC.mm39.refGene"
[15] "TxDb.Mmusculus.UCSC.mm9.knownGene" パッケージのインストール
BiocManager::install()
関数を使用して、パッケージをインストールまたは更新します。
この関数は、パッケージ名の文字列ベクトルを受け取り、Bioconductor リポジトリからこれらをインストールしようとします。
R
BiocManager::install(c("S4Vectors", "BiocGenerics"))
ただし、Bioconductor
リポジトリでパッケージが見つからない場合、関数はグローバルオプション
repos
にリストされているリポジトリでこれらのパッケージも検索します。
貢献する!
BiocManager を使用してインストールできる非 Bioconductor パッケージの例を追加します。 できれば、このレッスンで後で使用されるパッケージです。
パッケージのアンインストール
Bioconductor パッケージは、基本 R 関数 remove.packages()
を使用して、他の R パッケージと同様に R ライブラリから削除できます。
本質的に、この関数は単にインストールされたパッケージを削除し、必要に応じてインデックス情報を更新します。
その結果、そのパッケージをセッションに添付したり、そのパッケージのドキュメントを参照したりすることはできなくなります。
R
remove.packages("S4Vectors")
- BiocManager パッケージは CRAN リポジトリから利用可能です。
-
BiocManager::install()は、Bioconductor パッケージ (CRAN および GitHub からも) のインストールと更新に使用されます。 -
BiocManager::valid()は、利用可能なパッケージの更新をチェックするために使用されます。 -
BiocManager::version()は、現在インストールされている Bioconductor のバージョンを報告します。 -
BiocManager::install()は、特定の Bioconductor のバージョンに対して R ライブラリ全体を更新するためにも使用できます。
Content from ヘルプを取得する
Last updated on 2025-09-23 | Edit this page
Overview
Questions
- どこでオンラインの助けを見つけることができますか?
- パッケージの開発者や他のユーザーに質問するにはどこに行けばよいですか?
- 特定のパッケージのドキュメントはどこで見つけることができますか?
- 複数のパッケージを組み合わせて、一貫したワークフローを学ぶにはどこに行けばよいですか?
Objectives
- オンラインリソースを特定する手助けをします。
- パッケージのドキュメントにアクセスする。
Bioconductorパッケージに関するヘルプ
Bioconductorパッケージやベストプラクティスに関するヘルプは、いくつかの場所で入手可能です。 多くの場合、最良の助けの供給源は状況によって異なります。
- 特定のタスクに対して最適なパッケージを特定しようとしていますか?
- パッケージを初めて使用しようとしていますか?
- 特定の種類の分析において、複数のパッケージや関数を合理的なワークフローで使用および組み合わせるためのベストプラクティスについて不明な点はありますか?
- データに適用したときに関数がエラーを出していますか?
- 特定のパッケージや関数に実装された理論や方法論について質問がありますか?
次のセクションでは、Bioconductorユーザーが利用できるさまざまなヘルプの供給源と、それぞれが最も有用な状況について説明します。
Bioconductorウェブサイト
メインのBioconductorウェブサイトは、RやBioconductorパッケージをインストールする必要なしに、すべてのリソースを無料で提供します。
特に、biocViewsページは、パッケージのコレクションをテーマに沿って探索し、特定の機能を提供するBioconductorパッケージを特定する素晴らしい方法です。
さらに、ウェブサイトはコースや会議からの資料も収集しており、プレゼンテーション、ビデオ録画、教育資料が含まれています。
自然に、個々のプレゼンテーションやトレーニング資料は、特定のBioconductorパッケージのバージョンに結びついていることがよくあります。 そのため、教えられた資料において示されたパッケージのバージョンが、ユーザーのRライブラリのバージョンと一致しているかを確認することが重要です。 したがって、使用される教えられた資料において異なるバージョンのパッケージが参照されている場合には、期待される結果と実際の結果の間の不一致を注意深く解釈することが重要です。 パッケージのランディングページ
パッケージランディングページ
Bioconductorプロジェクトに受け入れられた各パッケージには、メインのBioconductorウェブサイトにランディングページが与えられます(例:S4Vectors)。
パッケージランディングページには、パッケージ自体をインストールする必要なしに参照できる便利な情報が含まれています。 特定のタスクに適したパッケージを探してBioconductorリポジトリを閲覧している場合、これは特に便利です。
ランディングページでは、見込みユーザーがパッケージ機能の短い説明や、パッケージビネットへのリンクを見つけることができます。 Bioconductorウェブサイトで入手可能なパッケージビネットは、開発者によって記述されており、パッケージで利用可能な関数がどのように使用され、完全なワークフローに組み合わされるべきかを示しています。 しばしば、ビネットでは、標準的なデータセットが使用され、公開リポジトリから無償で入手可能です(ExperimentDataパッケージやBioconductorのExperimentHub)。
ランディングページの「詳細」セクションでは、多くのパッケージが「BugReports」とラベル付けされたフィールドを提供します。 そのフィールドには、ユーザーがパッケージ開発者にバグを報告するために訪れることができるURLが提供されます。
注意
実際のソフトウェアバグと、パッケージに十分に慣れていないユーザーによる意図しないミスを区別するのが難しいことがあります。 このエピソードの後半では、バグを報告するためのアドバイスや、迅速かつ最も有用な返答を受け取るために必要な情報を含める方法を提供します。
疑わしい場合、Bioconductorサポートサイトは、個々の経験やパッケージおよびベストプラクティスに関する知識を共有できる素晴らしい場所でもあります。
さらに、ランディングページでは、パッケージが、ユーザーのシステムでパッケージ自体をインストールして使用できる前にインストールする必要のある他のBioconductorパッケージの数を示すソフトウェアの依存関係から、パッケージがすべてのテストに合格したかどうかを示す日次ビルドレポートまで、多くの重要な情報を提供しています。
さらに進む
各パッケージには、パッケージがリポジトリに追加されて以来、各Bioconductorのリリースに対してランディングページがあります、例:
- https://www.bioconductor.org/packages/3.14/bioc/html/BiocVersion.html
- https://www.bioconductor.org/packages/3.13/bioc/html/BiocVersion.html
- https://www.bioconductor.org/packages/3.12/bioc/html/BiocVersion.html
パッケージランディングページのURLのバージョン番号は、「release」または「devel」という単語に置き換えることができ、最新の安定版リリースまたは開発版のランディングページにアクセスできます。
パッケージビネット
各Bioconductorパッケージには少なくとも1つのビネットを含めることが求められています。 多くのパッケージには1つ以上のビネットがあり、コア機能を特定の使用例から分離しています。
このエピソードの前半で説明したように、ビネットはBioconductorウェブサイトのパッケージランディングページから入手可能です。 ただし、ランディングページは、各Bioconductorのバージョンの最も最近のバージョンのパッケージドキュメントにしかリンクされていません。 これは、ユーザーのRライブラリにインストールされ、Rセッションで使用されるパッケージのバージョンとは異なる可能性があります。
ユーザーのRライブラリにBioconductorパッケージがインストールされると、そのパッケージの特定のバージョンに関連付けられたビネットもユーザーのコンピュータにインストールされます。
それらのローカルにインストールされたビネットは、現在RライブラリにインストールされてRセッションで使用されるパッケージのバージョンのゴールドスタンダードリファレンスです。
それらはbrowseVignettes()関数を使用してアクセスできます。例えば:
R
browseVignettes("BiocManager")
具体的には、browseVignettes()関数は、ユーザーのデフォルトのウェブブラウザで開かれ、リクエストされたパッケージに利用可能なすべてのビネットをリストします。
各ビネットは3つのフォーマットで提供されます:
- コンパイル済みのPDFまたはHTML形式
- ソース、SweaveまたはR Markdown形式
- Rスクリプトとして
プリコンパイル形式は、ドキュメントの内容を統合されたビューでプレビューできるため、通常は最も快適に読むことができます。 これには、プレーンテキストの説明と、コードおよびそれらの出力(図やコンソールメッセージの両方)が含まれます。
パッケージヘルプページ
Bioconductorは、すべてのユーザー向けパッケージ機能がパッケージヘルプページ(通常は「manページ」と呼ばれ、パッケージのサブディレクトリの名前に由来します)で文書化されることを要求します。
help()関数や疑問符シンボル?を使用してヘルプページにアクセスできます。
R
help(topic = "install", package = "BiocManager")
?BiocManager::install
Bioconductorパッケージのヘルプページは、フォーマットや基本的な内容に関して、一般的にCRANパッケージと同じルールに従います。 ただし、Bioconductorは、エクスポートされたオブジェクトを文書化するためのほとんどのmanページには、実行可能な例が必要となることも要求します。 実行可能な例は、ユーザーが即座に利用可能な小規模データセットに対して、個別の関数の使用を示すのに特に役立ちます - 人工的にシミュレーションされたデータや、公共のデータリポジトリからプログラムでインポートされたデータのいずれかです。
特に、実行可能な例は、理想的なケースで関数の使用を示し、関数への入力がどのようにフォーマットされるべきか、また関数の出力にユーザーがどのような情報を得られるかを示します。 これらの例を実行し、例の入力と出力をユーザー自身のデータと比較することで、関数をユーザー自身のデータに適用する前に必要な変換について重要な洞察を提供します。関数の出力がどのように解釈され、相互作用できるかについてもです。
Bioconductorサポートサイト
Bioconductorサポートサイトは、ユーザーと開発者のコミュニティが質問をし、Bioconductorパッケージや分析ワークフローに関する疑問や課題を通じてお互いをサポートするためのプラットフォームを提供します。
サポートサイトは、アカウントなしで自由にブラウズでき、すでに質問され、回答された多くの質問を検索して読むことができます。 ただし、新しい質問を投稿するには、プラットフォーム上のアカウントが必要です。 プラットフォームへのサインアップは、信頼されているいくつかのプロバイダーからのメールアドレスやオープンオーソリゼーションを使用して簡単に行うことができます。
アップボート制度により、最も人気のある回答が各ページの上部に表示されやすくなります。 さらに、オリジナルの投稿者は、自分の問題を解決した回答を1つ選ぶ権利を保持しています。
さらに、回答を提供することでオリジナルの投稿者によって人気または受け入れられたユーザーに付与されたポイント制度は、プラットフォーム上で最も活発で信頼されている貢献者を際立たせます。
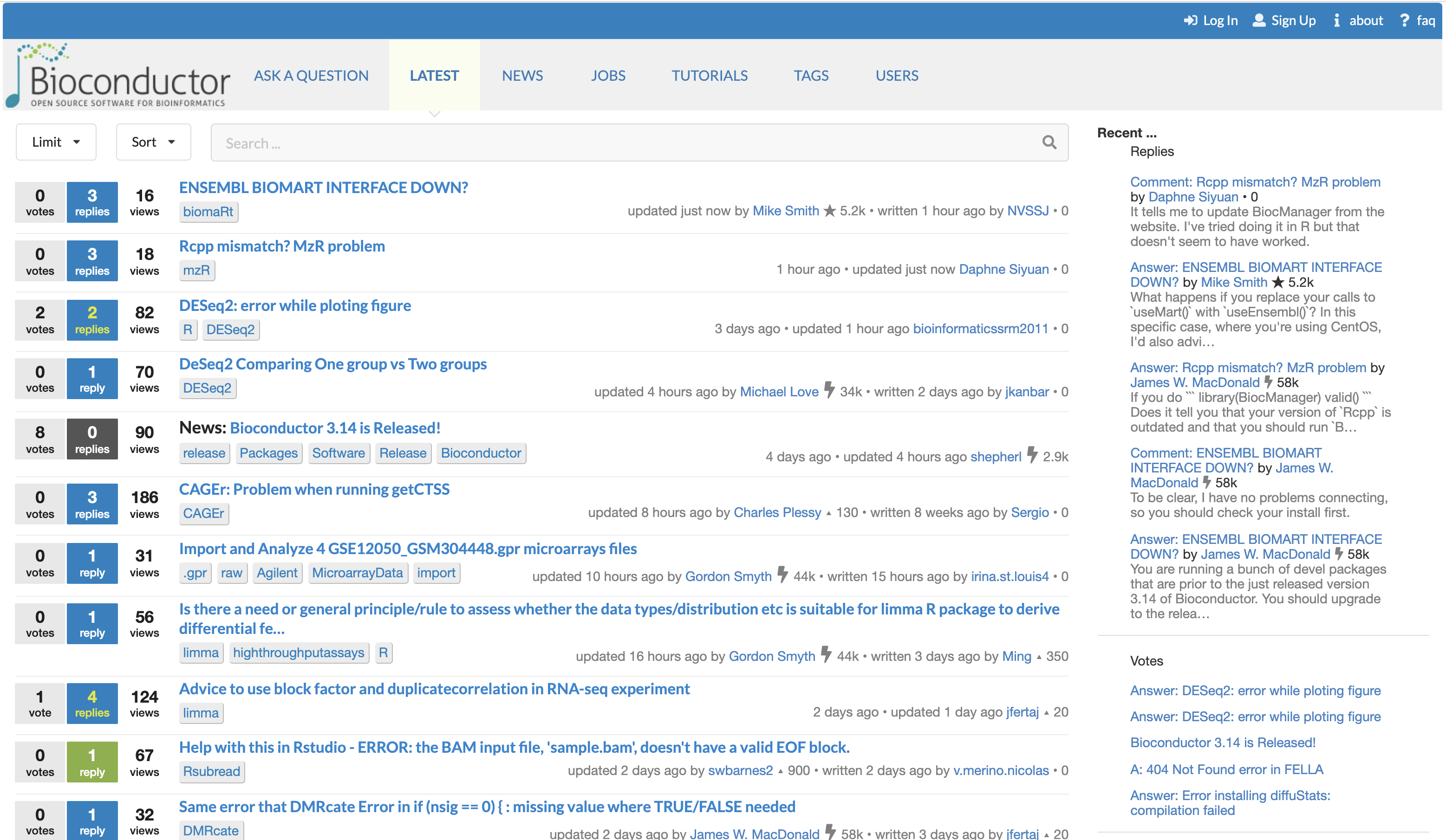
Bioconductorサポートサイト。 Bioconductorサポートサイトは、登録ユーザーによって投稿された質問と回答を追跡します。 プラットフォームは、未登録のユーザーによって自由にブラウズおよび検索が可能です。
ワークフローパッケージ
Bioconductorワークフローパッケージは、独自の追加コードや機能なしでビネットのみを含むことが期待される点で特別です。 代わりに、ワークフローパッケージのビネットは他のパッケージから機能を独占的にインポートし、ユーザーが日常の作業中に直面する可能性のある関数をどのように組み合わせて統合されたワークフローを実現するかを示します。
通常のビネットのように、データは一般に公開されているソースから取得され、BioconductorのExperimentDataパッケージやBioconductorのExperimentHubを含みます。 これらの無料で入手可能な標準データセットにより、ユーザーはビネットを読みながら、出力を対話的に再現することができます。
ワークフローパッケージは、biocViewsページの専用セクションで閲覧できます。
Slackワークスペース
Bioconductor Slackワークスペースは、2016年に設立されました。
このHerokuアプリを使用して、個々のメールアドレスの招待を生成することで、自由に参加できます。
Bioconductor Slackワークスペースは、公式発表(例:Bioconductorリリース、会議&イベント)、および特定のトピックに登録されたユーザーグループ間の非公式な議論、そしてコミュニティメンバー間の直接メッセージのための活発なオンラインプラットフォームです。
このワークスペースには、コミュニティ内の特定のトピックや関心分野に特化した多数のチャネルがあります。 これらのチャネルは、活発な研究分野(例:単一細胞ゲノミクス)から、期間限定のイベント(例:会議)、さらには地域社会の Outreach(例:多様性と代表性)まで一般的です。
プライベートチャネルも、ガバナンス(例:イベントの組織、諮問委員会)に存在します。
注意
Slackワークスペースは、チャネルや直接メッセージを通じて仲間のコミュニティメンバーと迅速な日常的コミュニケーションや議論を行うことを可能にします。
ただし、人気のあるチャネルには、さまざまなタイムゾーンにいる数百人のユーザーが集まる可能性があり、節度と配慮を持って使用する必要があります。 対照的に、直接メッセージやプライベートチャネルは、議論に招待されたユーザーに制限され、コミュニティにとって関連する結果は公のチャネルに再投稿する必要があります。
その結果、Bioconductorサポートサイトは、コミュニティにとって興味のある質問を公にするための好ましい方法として残ります。これは、質問、議論、回答が簡単に検索可能であり、主要な検索エンジンによってインデックスされるためです。
ヘルプを効率的に求めるには
コミュニティメンバーのほとんどは、ボランティアとして、自己の余暇の時間で、無報酬で助けを提供しています。 したがって、問題の原因を迅速に特定するのを手助けするために、関連情報を提供して、できるだけ明確に質問を行うことが重要です。そうすれば、助ける側と元々の投稿者の両方の時間を節約できます。
質問の内容によっては、いくつかの重要な情報が含まれます:
- オペレーティングシステム
- Rのバージョン
- Bioconductorのバージョン
- Rライブラリにインストールされている個別のパッケージのバージョン
- 現在のセッションにアタッチされている個別のパッケージのバージョン
- ユーザーのシステムにインストールされているサードパーティ製ソフトウェア、ライブラリ、およびコンパイラ
- Rライブラリにインストールされたパッケージのソース(例:Bioconductor、CRAN、GitHub)
- 問題に至るまでにRセッションで実行されたコード
- セッションで有効なグローバルオプション(
options()を使用してアクセス可能)
問題が実行されるコードに関連していて、想定外の出力を生成する場合、他の人がコンピュータ上で問題を再現できるように十分な情報を含めることが重要です。 実際、多くの問題には、問題の原因を適切に調査するため、修正をテストするため、または回避策やアドバイスを提供するためには、ライブのRセッションが必要です。
重要なのは、投稿の一部としてコードを提供する際には、読者が実行可能なコードであり、コードによって処理されたデータも含まれていることが重要です。
しばしば、コード自体は正しく見えますが、問題はコードと特定のデータセットとの相互作用に関連しています。
機密データを共有することができない場合、問題は再計画されて、インターネット上で公開されているデータセットを使用して提示されるか、再現可能な方法でランダムに生成されたシミュレーションデータを生成するためのコードを含む必要があります(例:set.seed())。
1つの選択肢は、reprexパッケージを使用して、問題を説明するコードおよび出力を整理して、Bioconductorサポートサイトなどの多くのオンラインフォーラムで投稿しやすい形式のテキストにします。
最後に、Bioconductorサポートサイトは、Bioconductorパッケージに関連する質問を投稿するための好ましいプラットフォームです。 それは、質問がコミュニティ全体に表示され、定期的にその質問に回答する経験豊富なBioconductorユーザーや、問題に直面するまでにすでに投稿されて解決済みの質問に対する回答を見つけることができる他のユーザーを含むからです。
-
browseVignettes()関数は、各パッケージにインストールされているビネットにアクセスするために推奨されています。 - ビネットはBioconductorウェブサイトでもアクセスできますが、パッケージバージョン間の違いには注意してください!
- Bioconductorメインウェブサイトには一般情報、パッケージドキュメント、コース資料が含まれています。
- Bioconductorサポートサイトは、開発者に連絡し、質問をすべて行うために推奨される場所です。
Content from BioconductorにおけるS4クラス
Last updated on 2025-09-23 | Edit this page
Overview
Questions
- S4クラスシステムとは?
- BioconductorはS4クラスをどのように使用しますか?
- Bioconductorの
DataFrameは、基本のdata.frameとどう違うのですか?
Objectives
- S4クラス、ジェネリックおよびメソッドとは何かを説明してください。
- Bioconductorパッケージインフラストラクチャの中心にあるS4クラスを特定します。
- さまざまなS4オブジェクトを作成し、関連するS4メソッドを適用します。
パッケージのインストール
以下のセクションに進む前に、必要なBioconductorパッケージをいくつかインストールします。
最初に、BiocManagerパッケージがインストールされているかを確認し、それを使用しようとする前にインストールします。
次に、BiocManager::install()関数を使用して必要なパッケージをインストールします。
R
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("S4Vectors")
講師のために
このエピソードの最初の部分は、少し理論的に見えるかもしれません。
S4クラスシステムの内部動作に関する詳細に深入りしないでください(例えば、new()関数に言及する必要はありませんし、クラスを作成する具体的なコード例を示す必要もありません)。
代わりに、最初の図のキャプションでは、技術用語を単純な文で導入しながら、図を通じて段階的に説明し、S4クラスシステムの中心となるメソッドディスパッチの概念に至り、初心者ユーザーにとっての混乱の原因となることを示しています。
S4クラスとメソッド
メソッドパッケージ
S4クラスシステムは、基本パッケージmethodsに実装されています。 そのため、この概念はBioconductorプロジェクトに特有なものではなく、さまざまな独立したパッケージにも存在します。 この主題は、Hadley Wickhamによるオンライン書籍Advanced Rで詳しく文書化されています。 ほとんどのBioconductorユーザーは、S4クラスシステムの詳細を過度に熟知する必要がないでしょう。 むしろ、Bioconductorプロジェクトにおけるパッケージの効率的な使用の鍵は、S4クラスシステムを使用する重要な動機と、ユーザー向けの機能に関するベストプラクティス(クラス、ジェネリック、メソッドを含む)を十分に理解することにあります。 このエピソードの次のセクションでは、BioconductorプロジェクトにおけるS4クラスとメソッドの基本的な機能性とユーザーエクスペリエンスに焦点を当てます。
一方で、S4クラスは、Rセッション内の変数名に割り当てることができる計算オブジェクトにおいて、任意に複雑な情報を格納できるデータ構造を提供します。 もう一方で、S4ジェネリックおよびメソッドは、それらのオブジェクトを処理するために適用される関数を定義します。
これまでの数年で、BioconductorプロジェクトはS4クラスシステムを使用して、ほとんどの生物学的アッセイ(生アッセイデータや処理済みアッセイデータ、個別の特徴やサンプルに関する実験メタデータ、関連する他のアッセイ特有の情報を含む)のデータを格納および処理できるいくつかのクラスとメソッドを開発してきました。 Bioconductorパッケージ全体で一般的に使用される標準S4クラスに慣れることは、分析ワークフローを開発したいユーザーがベストプラクティスに従う自信を高めるための重要なステップです。
S4クラス、ジェネリック、およびメソッド。
左側には、S4Class1とS4Class2という2つの例のクラスが、継承の概念を示しています。
クラスS4Class1には、データを格納するためのSlotName1とSlotName2という2つのスロットがあります。
これらの2つのスロットは、それぞれSlotType1およびSlotType2型のオブジェクトを格納するように制限されています。
このクラスは、オブジェクトが更新されるたびにデータの整合性を確認する有効性ルールも定義しています。
クラスS4Class2は、S4Class1からすべてのスロットと有効性ルールを継承し、新しいスロットSlotName3と新しい有効性ルールを定義します。
例のコードは、それぞれのクラスのオブジェクトが通常、対応するクラスにちなんで命名されたコンストラクタ関数を使用してどのように作成されるかを示しています。
右側には、1つのジェネリック関数と2つのメソッドが多様性の概念とS4メソッドディスパッチのプロセスを示しています。
ジェネリック関数S4Generic1()は、関数の名前とその引数を定義します。
ただし、その関数の実装は提供されていません。
代わりに、2つのメソッドが定義され、それぞれ特定のクラスの入力に対して単純な実装を提供します。
具体的には、最初のメソッドは、S4Class1のオブジェクトが引数xとして与えられた場合のS4Generic1()の実装を定義し、2番目のメソッドは、S4Class2のオブジェクトが引数xとして与えられた場合のS4Generic1()の別の実装を提供します。
ジェネリック関数S4Generic1()が呼び出されると、メソッドディスパッチと呼ばれるプロセスが発生し、渡されたオブジェクトのクラスに応じてS4Generic1()メソッドの適切な実装が呼び出されます。
スロットと有効性
S3クラスシステムは基本Rに直接用意されているのに対し(このレッスンでは説明されていません)、S4クラスシステムは、Rにおけるオブジェクト指向プログラミング(OOP)のためのクラスとメソッドのより厳格な定義を提供します。
OOPモデルを実装している多くのプログラミング言語と同様に、S4クラスは実世界のエンティティを、情報を*slots*と呼ばれる1つ以上の内部コンポーネントに格納する計算オブジェクトとして表現するために使用されます。
クラスの定義は、各スロットに格納される可能性のあるデータのタイプを明示します。不適切なデータを格納しようとした場合にはエラーが発生します。
さらに、クラスの定義には、型を超えてオブジェクトに格納されるデータの有効性を確認するコードを含めることもできます。
例えば、numeric型のスロットを使用して人の年齢を格納することはできますが、有効性メソッドは格納される値が実際に正であることを確認できます。
継承
OOPモデルの中核的な柱の1つは、既存のクラスの機能を継承して拡張できる新しいクラスを開発する可能性です。 S4クラスシステムはこのパラダイムを実装しています。
新しいS4クラスの定義は、継承する他のクラスの名前を宣言します。 新しいクラスには、親クラスのすべてのスロットが含まれ、新しいクラスの定義に追加される新しいスロットが含まれます。
新しいクラスの定義では、新しい有効性チェックも定義でき、親クラスの各クラスに実装された各有効性チェックに追加されます。
ジェネリックおよびメソッド
クラスが情報を格納するデータ構造を定義する一方で、ジェネリックおよびメソッドは、それらのクラスからインスタンス化されたオブジェクトに適用される関数を定義します。
S4ジェネリック関数は、いくつかの重要な引数に依存して、異なる振る舞いが期待される関数の名前を宣言するために使用されます。 その代わりに、S4メソッドは、各特定の入力の組み合わせに対してジェネリック関数の異なる実装を定義するために使用されます。
ジェネリック関数が呼び出され、S4オブジェクトが渡されると、メソッドディスパッチと呼ばれるプロセスが発生し、オブジェクトのクラスが実行すべき適切なメソッドを決定するために使用されます。
S4Vectorsパッケージ
S4Vectorsパッケージは、VectorおよびListの仮想クラスと、Rの通常のベクトルおよびリストのセマンティクスを拡張する一連のジェネリック関数を定義しています。
S4クラスシステムを使用することで、パッケージ開発者はVectorやListの具体的なサブクラスとして、ベクトルやリストに似たオブジェクトを簡単に実装できます。
仮想クラス(VectorとListなど)は、オブジェクト自体としてインスタンス化することはできません。
むしろ、これらの仮想クラスは、それらから派生したすべての具体的なクラスが継承する基本機能を提供します。
代わりに、一般的に興味のあるいくつかの低レベルの具体的なサブクラス(例えば、DataFrame、Rle、Hits)は、S4Vectorsパッケージ自体に実装されています。また、Bioconductorプロジェクト全体の他のパッケージ(例えば、IRanges)にも、多くの他のパッケージが実装されています。
次のように、パッケージを現在のRセッションにアタッチします。
R
library(S4Vectors)
注意
パッケージをセッションにアタッチした場合、コンソールに印刷されるパッケージ起動メッセージには、S4Vectorsパッケージが、セッションにアタッチされると、baseパッケージから数機能をマスクすることが記載されています。 これは、S4Vectorsパッケージが、これらの関数の実装を含んでいることを意味し、Rセッションにアタッチされた最新のパッケージであるため、その関数の独自の実装がR検索パス上で最初に見つかり、baseパッケージの元の実装ではなく、使用されます。
多くの場合、マスクされた関数は、特に問題なく以前のように使用できます。
時折、パッケージ名に加えて関数名を使ってマスクされた関数を呼び出す必要があるかもしれません。例:base::anyDuplicated()。
DataFrameクラス
長方形データの概念への拡張
S4Vectorsパッケージで実装されたDataFrameクラスは、基本Rのdata.frameクラスやtidyverseのtibbleに慣れているユーザーに馴染みのある長方形データの概念を拡張しています。
具体的には、DataFrameは、カラムとしてあらゆるタイプのオブジェクト(lengthおよび[メソッドを持つ)を格納することをサポートしています。
全体として、DataFrameクラスは、構築、部分的選択、分割、結合などの観点から、非常に似た形で振る舞うS4クラスの正式な定義を提供します。
新しいオブジェクトを作成するには、DataFrame()コンストラクタ関数を使用する必要があります。これは、基本Rのdata.frame()に相当します。
その関数のヘルプページは、?DataFrameでアクセスできますので、詳細情報を参照できます。
R
DF1 <- DataFrame(
Integers = c(1L, 2L, 3L),
Letters = c("A", "B", "C"),
Floats = c(1.2, 2.3, 3.4)
)
DF1
OUTPUT
DataFrame with 3 rows and 3 columns
Integers Letters Floats
<integer> <character> <numeric>
1 1 A 1.2
2 2 B 2.3
3 3 C 3.4実際、DataFrameオブジェクトは、同等のdata.frameオブジェクトに簡単に変換できます。
R
df1 <- as.data.frame(DF1)
df1
OUTPUT
Integers Letters Floats
1 1 A 1.2
2 2 B 2.3
3 3 C 3.4逆に、as()関数を使用してdata.frameオブジェクトをDataFrameに変換することもできます。
R
as(df1, "DataFrame")
OUTPUT
DataFrame with 3 rows and 3 columns
Integers Letters Floats
<integer> <character> <numeric>
1 1 A 1.2
2 2 B 2.3
3 3 C 3.4基本のdata.frameとの違い
最も注目すべき例外は、行名の処理に関するものです。
まず、行名はオプションです。
これは、行名がない場合、rownames(x)がNULLを返すことを意味します。
R
rownames(DF1)
OUTPUT
NULLこれは、data.frameとは異なり、rownames(x)がas.character(seq_len(nrow(x)))の等価物を返すことになります。
R
rownames(df1)
OUTPUT
[1] "1" "2" "3"ただし、NULLを返すことは、例えば、組み合わせ関数に対して、行名が不要であることを通知します(大規模なデータを扱うときには、しばしば贅沢です)。
さらに、DataFrameオブジェクトの行名は一意である必要はありません。これは、基本Rのdata.frameとは対照的です。
行名は、長方形データで観測結果を一意に特定してインデックス付けするために使用されるため、しばしば議論の原因となります。
設定されている場合、行名は[演算子を使用して長方形データを部分選択するために使用できます。
非一意の行名はその目的を妨げ、選択された各行名の最初の出現だけが抽出されるため、予期しない結果をもたらす可能性があります。
その代わりに、tidyverseのtibbleは、行名を完全に設定する機能を排除し、ユーザーに特定のカラムに情報を明示的にストアさせるように強制しています。また、行を効率的にフィルタリングするための関数を提供し、[演算子を使用する必要はありません。
R
DF2 <- DataFrame(
Integers = c(1L, 2L, 3L),
Letters = c("A", "B", "C"),
Floats = c(1.2, 2.3, 3.4),
row.names = c("name1", "name1", "name2")
)
DF2
OUTPUT
DataFrame with 3 rows and 3 columns
Integers Letters Floats
<integer> <character> <numeric>
name1 1 A 1.2
name1 2 B 2.3
name2 3 C 3.4チャレンジ
上記の例を使用して、DF2["name1", ]は何を返しますか?
なぜですか?
> DF2["name1", ]
DataFrame with 1 row and 3 columns
Integers Letters Floats
<integer> <character> <numeric>
name1 1 A 1.2行名name1に一致した行の最初の出現のみが返されます。
この場合、行名には特別な意味がなく、それらの必要性を正当化することが難しいです。
代わりに、ユーザーは、DF2[rownames(DF2) == "name1", ]のように、行名name1に一致するすべての行をより明示的に抽出することができます。
ユーザーは、特定の状況で行名を使用する動機、何を表し、分析中にどのように使用されるべきかを意識するべきです。
最後に、DataFrameの行名は、部分一致による選択をサポートしません。これは、基本のdata.frameとは対照的です。
DataFrameのより厳格な動作により、予期しない結果が生じることは、意図しないユーザーによって防止されます。
R
DF3 <- DataFrame(
Integers = c(1L, 2L, 3L),
Letters = c("A", "B", "C"),
Floats = c(1.2, 2.3, 3.4),
row.names = c("alpha", "beta", "gamma")
)
df3 <- as.data.frame(DF3)
チャレンジ
上記の例を使用して、DF3["a", ]とdf3["a", ]の出力は何ですか?
なぜ違うのですか?
> DF3["a", ]
DataFrame with 1 row and 3 columns
Integers Letters Floats
<integer> <character> <numeric>
<NA> NA NA NA
> df3["a", ]
Integers Letters Floats
alpha 1 A 1.2DataFrameオブジェクトは、部分一致による行名のマッチングを実行せず、そのため、行がマッチせず、NA値のいっぱいのDataFrameを返しました。
その代わりに、data.frameオブジェクトは部分一致による行名マッチングを実行し、要求された"a"を"alpha"行名に一致させ、新しいdata.frameオブジェクトとして該当行を返しました。
インデックス付け
通常のdata.frameと同様に、カラムは$、[、および[[を使用してアクセスできます。
各演算子には異なる目的があり、最も適切なものは、達成しようとしていることによく依存します。
例えば、ドル演算子$は、名前で単一列を抽出するために使用できます。
それはしばしばベクターであることが多いですが、それはその列内のデータの性質によって異なる場合もあります。
この演算子は、インタラクティブなRセッションで非常に便利で、利用可能な列名の自動補完を提供します。
R
DF3$Integers
OUTPUT
[1] 1 2 3同様に、ダブルブラケット演算子[[も単一の列を抽出するために使用できます。
これは$よりも柔軟性があり、文字列名と整数インデックスの両方を処理できます。
R
DF3[["Letters"]]
OUTPUT
[1] "A" "B" "C"R
DF3[[2]]
OUTPUT
[1] "A" "B" "C"演算子[は、行と列の同時選択や、単一列の選択がDataFrameまたはvectorとして返されるかどうかを制御する際に最も便利です。
R
DF3[2:3, "Letters", drop=FALSE]
OUTPUT
DataFrame with 2 rows and 1 column
Letters
<character>
beta B
gamma Cメタデータ列
DataFrameにおける最も注目すべき新機能は、メタデータを別のDataFrameでカラムに保持できる能力です。
メタデータ列。
メタデータ列は、DataFrameオブジェクトの文脈で示されます。
左側には、AとBという名前の列を持つDataFrameオブジェクトDFが作成されます。
右側には、DFのメタデータ列をmcols(DF)を使ってアクセスします。
この例では、名前meta1とmeta2の2つのメタデータ列が作成されます。
メタデータ列は、親DataFrameの各列に対して1行を含むDataFrameとして格納されます。
メタデータ列は、関数mcols()を使用してアクセスされます。
メタデータ列が定義されていない場合、mcols()は単にNULLを返します。
R
DF4 <- DataFrame(
Integers = c(1L, 2L, 3L),
Letters = c("A", "B", "C"),
Floats = c(1.2, 2.3, 3.4),
row.names = c("alpha", "beta", "gamma")
)
mcols(DF4)
OUTPUT
NULL関数mcols()は、メタデータ列を追加、編集、または削除するためにも使用できます。
例えば、2つの列を持つDataFrameとして、メタデータ列を初期化できます。
- 対応する列に格納されている値のタイプを示す1つの列
- 対応する列で観察された異なる値の数を示す1つの列
R
mcols(DF4) <- DataFrame(
Type = sapply(DF4, typeof),
Distinct = sapply(DF4, function(x) { length(unique(x)) } )
)
mcols(DF4)
OUTPUT
DataFrame with 3 rows and 2 columns
Type Distinct
<character> <integer>
Integers integer 3
Letters character 3
Floats double 3注意
メタデータ列の行名は、親DataFrameの列名と一致するように自動的に設定され、列とメタデータのペアを明確に示します。
実行長エンコーディング (RLE)
ベクターの概念への拡張
S4Vectorsで実装されたDataFrameクラスと同様に、Rleクラスは基本パッケージのrle()関数にS4の拡張を提供します。
具体的には、Rleクラスは、原子ベクターを実行長エンコーディング形式で保存することをサポートしています。
実行長エンコーディング。 実行長エンコーディングの概念は、核酸の配列の例を使用してここに示されています。 エンコードの前に、配列の各位置にある各ヌクレオチドはメモリに明示的に保存されます。 エンコーディング中に、同一のヌクレオチドの連続したランは、ヌクレオチドの同一性とランの長さの2ビットの情報に圧縮されます。
実行長エンコーディングは、同一の情報の頻繁な連続が含まれるベクターのメモリ使用量を劇的に削減できます。
たとえば、実行長エンコーディングの有力な応用は、シーケンシング実験におけるゲノムカバレッジの表現です。ここで、大きなゲノム領域にマッピングされたリードが存在しない場合、長い0の値の連続が発生します。
各個別の値を保存することは、メモリ使用量の観点から非常に非効率的です。
その代わりに、実行長エンコーディングプロセスは、冗長な情報のそのような連続を、同一の情報の任意の長い連続から2つの値、すなわち繰り返される値自体と、それが繰り返される回数に圧縮します。
R
v1 <- c(0, 0, 0, 0, 0, 0, 0, 1, 2, 3, 2, 1, 0, 0, 0, 0, 0)
rle1 <- Rle(v1)
rle1
OUTPUT
numeric-Rle of length 17 with 7 runs
Lengths: 7 1 1 1 1 1 5
Values : 0 1 2 3 2 1 0インデクシング
通常のvectorと同様に、Rleオブジェクトは[を使用してインデックス付けすることができます。
R
rle1[2:4]
OUTPUT
numeric-Rle of length 3 with 1 run
Lengths: 3
Values : 0使用法
ベクターのようなオブジェクトとして、RleオブジェクトもDataFrameオブジェクトの列として保存でき、他のベクター状のオブジェクトとともに格納できます。
R
v2 <- c(rep(1, 5), rep(2, 5))
rle2 <- Rle(v2)
DF5 <- DataFrame(
vector = v2,
rle = rle2,
equal = v2 == rle2
)
DF5
OUTPUT
DataFrame with 10 rows and 3 columns
vector rle equal
<numeric> <Rle> <Rle>
1 1 1 TRUE
2 1 1 TRUE
3 1 1 TRUE
4 1 1 TRUE
5 1 1 TRUE
6 2 2 TRUE
7 2 2 TRUE
8 2 2 TRUE
9 2 2 TRUE
10 2 2 TRUEさらに進む
Rleオブジェクトに関する多くの標準操作は、Rleクラスのヘルプページに文書化されており、?Rleとしてアクセス可能で、S4Vectorsパッケージのビネットにおいても、browseVignettes("S4Vectors")を使ってアクセスできます。
- S4クラスはスロットに情報を保存し、オブジェクトが更新されるたびに情報の有効性を確認します。
- S4オブジェクトの継続的な整合性を確保するために、ユーザーはスロットに直接アクセスせず、専用の関数を使用する必要があります。
- S4ジェネリックは、与えられたオブジェクトのクラスに応じてメソッドの異なる実装を呼び出します。
- S4クラス
DataFrameは、data.frameの基本機能を拡張しており、メタデータ列の各列に関する情報を保持できる能力を持っています。 - S4クラス
Rleは、基本のvectorの機能を拡張しており、メモリ効率の良い形式で繰り返しベクターをエンコードする能力を持っています。
Content from 生物学的配列の取り扱い
Last updated on 2025-09-23 | Edit this page
Overview
Questions
- Bioconductor で生物学的配列を表現するための推奨される方法は何ですか?
- 生物学的配列を効率的に処理するためのメソッドを提供する Bioconductor パッケージはどれですか?
Objectives
- Bioconductor プロジェクトにおいて生物学的配列がどのように表現されているかを説明してください。
- 生物学的配列を処理するために利用可能な Bioconductor パッケージとメソッドを特定してください。
パッケージのインストール
次のセクションに進む前に、必要な Bioconductor
パッケージをインストールします。 まず、BiocManager
パッケージがインストールされているか確認します;
そうでなければ、インストールします。
次に、BiocManager::install()
関数を使用して必要なパッケージをインストールします。
R
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("Biostrings")
Biostrings パッケージとクラス
なぜ生物学的配列のためのクラスが必要なのですか?
生物学的配列は、計算上、最も単純な生物エンティティの一部であると主張できます。 例として、核酸配列(例: DNA, RNA)やアミノ酸から構成されるタンパク質配列があります。
それは、アルファベットが個々のモノマーをキャラクターシンボルを使用して表現するために設計され、同意されているからです。
たとえば、アミノ酸のアルファベットを使用すると、アクチン、α骨格筋タンパク質配列 の参照タンパク質配列は次のように表されます。
OUTPUT
[1] "MCDEDETTALVCDNGSGLVKAGFAGDDAPRAVFPSIVGRPRHQGVMVGMGQKDSYVGDEAQSKRGILTLKYPIEHGIITNWDDMEKIWHHTFYNELRVAPEEHPTLLTEAPLNPKANREKMTQIMFETFNVPAMYVAIQAVLSLYASGRTTGIVLDSGDGVTHNVPIYEGYALPHAIMRLDLAGRDLTDYLMKILTERGYSFVTTAEREIVRDIKEKLCYVALDFENEMATAASSSSLEKSYELPDGQVITIGNERFRCPETLFQPSFIGMESAGIHETTYNSIMKCDIDIRKDLYANNVMSGGTTMYPGIADRMQKEITALAPSTMKIKIIAPPERKYSVWIGGSILASLSTFQQMWITKQEYDEAGPSIVHRKCF"しかし、通常の文字ベクトルの大きな制限は、それらが含む配列の妥当性をチェックしないことです。 実際には、公式アルファベットに含まれない記号を含む無意味な記号列を文字列に格納することが可能です。 その場合、配列の妥当性をチェックする負担はそれらを処理するプログラムにかかるか、配列内の無効な記号が予期せず遭遇したときにエラーを引き起こすことになります。
代わりに、S4クラス – 以前のエピソードで示されているthe S4 class system – は、生物学的配列の「DNA」、「RNA」、または「タンパク質」という異なる種類のオブジェクトをラベル付けする方法を提供します。 このラベルは、配列に期待できるキャラクターシンボルのセットや、それらの配列に適用できる計算操作の範囲をプログラムに通知するための非常に強力な方法です。 たとえば、核酸配列を対応するアミノ酸配列に変換するために設計された関数は、核酸を表す配列に対してのみ実行されるべきです。
挑戦
この文字列が有効な DNA 配列であるかどうかを教えてくれますか?
AATTGGCCRGGCCAATTはい、これは IUPAC
表記で定義されたノンアンビギュリティのコードを使用した有効な DNA
配列です。
この場合、A、T、C、および
G は、4 つの標準的な核酸を表し、R
シンボルは、A または G
の2つのプリン核酸塩基のどちらかを表す正規表現として機能します。
Biostrings パッケージ
概要
Bioconductor プロジェクトにおいて、Biostrings
パッケージは、生物学的配列を S4 オブジェクトとして表現するための S4
クラスを実装しています。たとえば、脱ox DNA ポリマーの核酸の配列に対して
DNAString
があり、タンパク質ポリマーにおけるアミノ酸の配列に対しては
AAString があります。 これらの S4
クラスは、文字列のメモリ効率の良いコンテナ、自動的な妥当性チェック機能、生物学的分子の各クラスに対するさまざまな文字列一致アルゴリズムおよび他のユーティリティのメソッドを実装して、大規模な生物学的配列または配列のセットの迅速な操作と処理を可能にします。
Biostrings
パッケージで定義された基本クラスの簡単なプレゼンテーションは、vignette("Biostrings2Classes")
からアクセスできるパッケージのビネットの1つにあります。より詳細な情報は、browseVignettes("Biostrings")
からアクセスできる他のパッケージビネットに提供されます。
最初のステップ
開始するには、パッケージを読み込みます。
R
library(Biostrings)
パッケージが読み込まれ、セッションに追加されると、パッケージ関数にアクセスできます。
これには、パッケージで定義されたクラスの新しいオブジェクトを作成するための関数が含まれます。
たとえば、DNAString() コンストラクタ関数を使用して DNA
配列を表すオブジェクトを作成できます。
出力をオブジェクトに割り当てずに、結果として得られたオブジェクトをコンソールに出力させます。
R
DNAString("ATCG")
OUTPUT
4-letter DNAString object
seq: ATCG注意すべきは、DNA配列は、4つのDNA 核酸塩基を表すために
A、T、C、および G
のシンボルを含むことができ、未知または未指定の塩基に対しては
N
のシンボルを使用し、公式に定義された特別な意味を持つ一連の追加記号を制限していることです。
コンストラクタ関数は、無効な文字を含むオブジェクトを作成することを許可しないことに注意してください。たとえば、Z
は無効です。
R
DNAString("ATCGZ")
ERROR
Error in .Call2("new_XString_from_CHARACTER", class(x0), string, start, : key 90 (char 'Z') not in lookup table具体的に言えば、IUPAC
拡張遺伝子アルファベット
は、一種の核酸を表現するセットを表すアンビギュリティコードを定義しています。
IUPAC_CODE_MAP という名前のキャラクター ベクトルは、IUPAC
核酸のアンビギュリティ
コードからそれらの意味へのマッピングを含みます。
R
IUPAC_CODE_MAP
OUTPUT
A C G T M R W S Y K V
"A" "C" "G" "T" "AC" "AG" "AT" "CG" "CT" "GT" "ACG"
H D B N
"ACT" "AGT" "CGT" "ACGT" これらの核酸コードは、DNAString
オブジェクトの配列に含めることができます。 たとえば、シンボル
M は、核酸配列の特定の位置において、A もしくは
C の2つの核酸のどちらかを表します。
R
特に、`r BiocStyle::Biocpkg("Biostrings")` パッケージで実装されているパターンマッチングメソッドは、各生物学的配列のアンビギュリティコードの意味を認識し、ユーザーが照会したモチーフに効率的に一致させることができ、複雑な正規表現を設計する必要がありません。ERROR
Error in parse(text = input): <text>:1:3: unexpected invalid token
1: 特に、
^特に、Biostrings
パッケージで実装されたパターンマッチング方法は、各種生物学的配列のあいまいさコードの意味を認識し、ユーザーがクエリしたモチーフを効率的に一致させることができるため、煩雑な正規表現を設計する必要がありません。
たとえば、matchPattern() メソッドは、pattern=
と subject= 引数を取り、subject
配列の任意の位置で pattern 式の一致を報告および表示する
Views オブジェクトを返します。
デフォルトオプション fixed = TRUE
は、メソッドにクエリを正確に一致させるよう指示します。すなわち、アンビギュリティコードを無視し、この場合正確な一致は報告されません。
R
dna1 <- DNAString("ATCGCTTTGA")
matchPattern("GM", dna1, fixed = TRUE)
OUTPUT
Views on a 10-letter DNAString subject
subject: ATCGCTTTGA
views: NONEその代わりに、パターンにアンビギュリティコードが含まれていることを示すには、引数
fixed を FALSE に設定する必要があります。
R
matchPattern("GM", dna1, fixed = FALSE)
OUTPUT
Views on a 10-letter DNAString subject
subject: ATCGCTTTGA
views:
start end width
[1] 4 5 2 [GC]
[2] 9 10 2 [GA]この特定の例では、2つのビューがサブジェクトシーケンスのパターンの一致を説明します。
具体的には、GM
のパターンは最初にサブジェクトシーケンスの位置 4 から 5 にまたがる
GC のシーケンスと一致し、次に位置 9 から 10 のシーケンス
GA でも一致しました。
matchPattern()
メソッドと同様に、countPattern()
メソッドは、subject 配列内の pattern
の一致数を単純にカウントするために適用できます。
また、fixed
オプションは、アンビギュリティコードを尊重するか、正確に一致させるかを制御します。
挑戦
次のコードは何回ヒットしますか? なぜですか?
dna2 <- DNAString("TGATTGCTTGGTTGMTT")
countPattern("GM", dna2, fixed = FALSE)countPattern() メソッドは 3
回のヒットを報告します。なぜなら、オプション fixed = FALSE
が GM パターンが GA、GC、および
GM
の配列と一致させることを可能にするからです。これは、pattern
にアンビギュリティコード M を使用しているためです。
ファイルから生物学的文字列をインポートする
実際には、ユーザーは、自分で生物学的配列を表す文字列をタイピングすることはめったにありません。 ほとんどの時間は、生物学的文字列は、公的なレポジトリからダウンロードされたファイルや、バイオインフォマティクスプログラムを使用してローカルで生成されたファイルからインポートされます。
たとえば、レッスンのセットアップ時にダウンロードしたファイルから、TruSeq™
DNA PCR-Free Whole Genome Sequencing Library Preparation
キットのアダプタシーケンスのセットを読み込みます。
アダプタシーケンスは核酸配列であるため、readDNAStringSet()
関数を使用する必要があります。
R
truseq_adapters <- readDNAStringSet(filepath = "data/TruSeq3-PE-2.fa")
truseq_adapters
OUTPUT
DNAStringSet object of length 6:
width seq names
[1] 34 TACACTCTTTCCCTACACGACGCTCTTCCGATCT PrefixPE/1
[2] 34 GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT PrefixPE/2
[3] 34 TACACTCTTTCCCTACACGACGCTCTTCCGATCT PE1
[4] 34 AGATCGGAAGAGCGTCGTGTAGGGAAAGAGTGTA PE1_rc
[5] 34 GTGACTGGAGTTCAGACGTGTGCTCTTCCGATCT PE2
[6] 34 AGATCGGAAGAGCACACGTCTGAACTCCAGTCAC PE2_rcさらなる進展
readDNAStringSet()
関数のヘルプページは、関連する生物学的配列タイプをインポートするために設計された関連関数を文書化しています。たとえば、readRNAStringSet()、readAAStringSet()
などがあります。
生物学的文字列に対する操作
記号の頻度を計算する
Biostrings
パッケージは、生物学的文字列のクラスを処理および操作するためのいくつかの関数を提供します。
たとえば、このエピソードの前に matchPattern() と
countPattern() に出会いました。
生物学的文字列に適用できるメソッドの別の例は、letterFrequency()
で、生物学的配列内の文字の頻度を計算します。
R
letterFrequency(truseq_adapters, letters = DNA_ALPHABET)
OUTPUT
A C G T M R W S Y K V H D B N - + .
[1,] 6 14 3 11 0 0 0 0 0 0 0 0 0 0 0 0 0 0
[2,] 5 8 10 11 0 0 0 0 0 0 0 0 0 0 0 0 0 0
[3,] 6 14 3 11 0 0 0 0 0 0 0 0 0 0 0 0 0 0
[4,] 11 3 14 6 0 0 0 0 0 0 0 0 0 0 0 0 0 0
[5,] 5 8 10 11 0 0 0 0 0 0 0 0 0 0 0 0 0 0
[6,] 11 10 8 5 0 0 0 0 0 0 0 0 0 0 0 0 0 0出力は、DNAStringSet オブジェクト内の各配列の 1
行と、デオキシリボ核酸のアルファベットに対して 1
列を持つ行列です。これは、Biostrings
パッケージから提供される、DNA_ALPHABET
という名前のビルトインオブジェクトによって得られます。
アミノ酸配列
DNAString と DNAStringSet
クラスと同様に、AAString と AAStringSet
クラスは、長いアミノ酸配列またはそれらのセットを効率的に保存および操作することを可能にします。
DNA
アルファベットのビルトインオブジェクトに似て、ビルトインオブジェクト
AA_ALPHABET、AA_STANDARD、AA_PROTEINOGENIC
は、アミノ酸配列の有効なシンボルのアルファベットの異なるサブセットを説明しています。
たとえば、AA_ALPHABET
オブジェクトは、完全なアミノ酸アルファベットのシンボルセットを説明しています。
R
AA_ALPHABET
OUTPUT
[1] "A" "R" "N" "D" "C" "Q" "E" "G" "H" "I" "L" "K" "M" "F" "P" "S" "T" "W" "Y"
[20] "V" "U" "O" "B" "J" "Z" "X" "*" "-" "+" "."挑戦
AA_PROTEINOGENIC
アルファベットオブジェクトに存在し、AA_STANDARD
アルファベットオブジェクトには存在しない2つのシンボルを特定するために、基本的な
R コードを使用してみましょう。
それらの2つのシンボルは何を表していますか?
> setdiff(AA_PROTEINOGENIC, AA_STANDARD)
[1] "U" "O"シンボル U と O
は、それぞれセレノシステインとピロリジンを表します。
これら2つのアミノ酸は、いくつかの種で通常はストップコドンとして解釈されるコドンによってコードされています。
したがって、彼らは「標準」アミノ酸のアルファベットには含まれておらず、これらのアミノ酸の特別な生物学を認めるために「タンパク質製生成的」アミノ酸のアルファベットが定義されました。
これらのアルファベットのいずれかは、アミノ酸配列の妥当性を決定するために使用できます。生物学的性質に応じて。
核酸配列の翻訳
S4クラス とオブジェクト指向プログラミング (OOP) モデルを使用する主な動機の1つは、S4 ジェネリックとメソッドのインフラストラクチャに依存しています。 前のエピソードで説明しましたが、The S4 class system において、ジェネリックは、関数呼び出しに提供された入力オブジェクトの性質に応じて、同じジェネリック関数名の異なる実装を定義して適用するためのメカニズムを提供します。
たとえば、Biostrings
パッケージは、DNA または RNA 配列をアミノ酸配列に変換するための
translate() というジェネリックの複数の実装を提供します。
ジェネリック translate()
でサポートされている入力オブジェクトのセットは、CRAN パッケージ methods
の関数 showMethods() を使用してリスト可能です。
R
showMethods("translate")
OUTPUT
Function: translate (package Biostrings)
x="DNAString"
x="DNAStringSet"
x="MaskedDNAString"
x="MaskedRNAString"
x="RNAString"
x="RNAStringSet"上記の出力では、ジェネリック関数 translate()
は、DNAString と RNAString クラスにおける DNA
および RNA
シーケンスを表すオブジェクトを処理する能力のあるメソッドを含むことがわかります。さらに、DNAStringSet
と RNAStringSet クラスのオブジェクトにおける DNA および RNA
のシーケンスのリスト、ならびに DNA および RNA
シーケンスを保存する能力のある他のクラスもリストされています。
translate() メソッドの使用を示すために、最初に、NIH Open Reading Frame
Finderにより特定されたオープンリーディングフレーム (ORF)
のセットを読み込みます。これは、Homo sapiens アクチン β (ACTB)
mRNA (RefSeq: NM\_001101) の標準的な遺伝コードを使用し、最小の ORF
長さは 75 核酸で、ATG スタートコドンのみで始めます。
R
actb_orf_nih <- readDNAStringSet("data/actb_orfs.fasta")
actb_orf_nih
OUTPUT
DNAStringSet object of length 13:
width seq names
[1] 222 ATGCCCACCATCACGCCCTGGTG...CGGGGCGGACGCGGTCTCGGCG gi|1519311456|ref...
[2] 1128 ATGGATGATGATATCGCCGCGCT...CGTCCACCGCAAATGCTTCTAG gi|1519311456|ref...
[3] 126 ATGATGATATCGCCGCGCTCGTC...CGCCCCAGGCACCAGGGCGTGA gi|1519311456|ref...
[4] 90 ATGTCGTCCCAGTTGGTGACGAT...CTGGGCCTCGTCGCCCACATAG gi|1519311456|ref...
[5] 225 ATGGGCACAGTGTGGGTGACCCC...AGCCACACGCAGCTCATTGTAG gi|1519311456|ref...
... ... ...
[9] 342 ATGAGATTGGCATGGCTTTATTT...ATGTAATGCAAAATTTTTTTAA gi|1519311456|ref...
[10] 168 ATGGCTTTATTTGTTTTTTTTGT...TTGCACATTGTTGTTTTTTTAA gi|1519311456|ref...
[11] 111 ATGACTATTAAAAAAACAACAAT...CCTTCACCGTTCCAGTTTTTAA gi|1519311456|ref...
[12] 105 ATGCAAAATTTTTTTAATCTTCG...CCTTTTTTGTCCCCCAACTTGA gi|1519311456|ref...
[13] 135 ATGATGAGCCTTCGTGCCCCCCC...TGACTTGAGACCAGTTGAATAA gi|1519311456|ref...核酸列を DNAStringSet
オブジェクトとしてインポートした後、そのオブジェクトに対して
translate()
メソッドを適用し、各核酸配列の翻訳プロセスによって生成されるアミノ酸配列を生成することができます。
R
actb_aa <- translate(actb_orf_nih)
actb_aa
OUTPUT
AAStringSet object of length 13:
width seq names
[1] 74 MPTITPWCLGRPTMEGKTARGAS...VWTGGGSAKARLCARGADAVSA gi|1519311456|ref...
[2] 376 MDDDIAALVVDNGSGMCKAGFAG...MWISKQEYDESGPSIVHRKCF* gi|1519311456|ref...
[3] 42 MMISPRSSSTTAPACARPASRATMPPGPSSPPSWGAPGTRA* gi|1519311456|ref...
[4] 30 MSSQLVTMPCSMGYFRVRMPLLLWASSPT* gi|1519311456|ref...
[5] 75 MGTVWVTPSPESITMPVVRPEAY...GFRGASVSSTGCSSGATRSSL* gi|1519311456|ref...
... ... ...
[9] 114 MRLAWLYLFFLFCFGFFFFFGLT...QVHTGEVIALLSCKLCNAKFF* gi|1519311456|ref...
[10] 56 MALFVFFVLFWFFFFFWLDSGFK...ERASPKVHNVAEDFDCTLLFF* gi|1519311456|ref...
[11] 37 MTIKKTTMCNQSPRPHCELWGMLAPTDCCHLHRSSF* gi|1519311456|ref...
[12] 35 MQNFFNLRLNTFLFCFILNDEPSCPPFPLFCPPT* gi|1519311456|ref...
[13] 45 MMSLRAPPSPFFVPQLEMYEGFWSPWEWVEAARAYLYTDLRPVE* gi|1519311456|ref...上記の例では、すべてのアミノ酸配列は、ATG
スタートコドンによってコードされる典型的なメチオニンアミノ酸から始まっています。
また、目に見えるアミノ酸配列のすべては一つを除いて、すべてが
*
シンボルで終わっており、これは翻訳プロセスがストップコドンで終了したことを示しています。
対照的に、上記の最初のオープンリーディングフレームは、ストップコドンに遭遇せずに核酸配列の終わりに到達しました。
便利なことに、各シーケンス内のアミノ酸の数は、width
ヘッダーの下に示されています。
挑戦
上記の各アミノ酸配列の長さを整数ベクトルとして抽出してください。 これらのオープンリーディングフレームから翻訳された最も長いアミノ酸配列の長さは何ですか?
あなたの結果を、ACTBの UniPro ページのシーケンス情報と比較してください。(https://www.uniprot.org/uniprot/P60709#sequences)
width(actb_aa)
# または
max(width(actb_aa))翻訳された最も長い配列には、376 のアミノ酸が含まれます。
UniProt ページでは、375 のアミノ酸の配列が報告されています。
ただし、UniProt
のアミノ酸配列にはストップコドンを表すためのシンボルは含まれていません。
それ以外の違いとして、UniProt のアミノ酸配列は、translate()
メソッドによって生成された配列と同一です。
BSgenome パッケージ
概要
Bioconductor プロジェクトでは、BSgenome パッケージが、全ゲノムとその単核苷酸ポリモーフィズムの効率的な表現のためのソフトウェアインフラストラクチャを提供しています。
BSgenome パッケージ自体にはゲノム配列は含まれていませんが、次のセクションで示すように、他の Bioconductor パッケージで利用可能なゲノム配列へのアクセス機能を提供します。
最初のステップ
開始するには、パッケージを読み込みます。
R
library(BSgenome)
パッケージが読み込まれ、セッションに追加されると、パッケージ関数にアクセスできます。
特に、関数 BSgenome::available.genomes()
を利用することで、ゲノムシーケンスを含む Bioconductor
パッケージの名前を表示できます。
R
available.genomes()
OUTPUT
'getOption("repos")' replaces Bioconductor standard repositories, see
'help("repositories", package = "BiocManager")' for details.
Replacement repositories:
BioCsoft: https://bioconductor.org/packages/3.19/bioc
BioCann: https://bioconductor.org/packages/3.19/data/annotation
BioCexp: https://bioconductor.org/packages/3.19/data/experiment
BioCworkflows: https://bioconductor.org/packages/3.19/workflows
BioCbooks: https://bioconductor.org/packages/3.19/books
CRAN: https://cloud.r-project.orgOUTPUT
[1] "BSgenome.Alyrata.JGI.v1"
[2] "BSgenome.Amellifera.BeeBase.assembly4"
[3] "BSgenome.Amellifera.NCBI.AmelHAv3.1"
[4] "BSgenome.Amellifera.UCSC.apiMel2"
[5] "BSgenome.Amellifera.UCSC.apiMel2.masked"
[6] "BSgenome.Aofficinalis.NCBI.V1"
[7] "BSgenome.Athaliana.TAIR.04232008"
[8] "BSgenome.Athaliana.TAIR.TAIR9"
[9] "BSgenome.Btaurus.UCSC.bosTau3"
[10] "BSgenome.Btaurus.UCSC.bosTau3.masked"
[11] "BSgenome.Btaurus.UCSC.bosTau4"
[12] "BSgenome.Btaurus.UCSC.bosTau4.masked"
[13] "BSgenome.Btaurus.UCSC.bosTau6"
[14] "BSgenome.Btaurus.UCSC.bosTau6.masked"
[15] "BSgenome.Btaurus.UCSC.bosTau8"
[16] "BSgenome.Btaurus.UCSC.bosTau9"
[17] "BSgenome.Btaurus.UCSC.bosTau9.masked"
[18] "BSgenome.Carietinum.NCBI.v1"
[19] "BSgenome.Celegans.UCSC.ce10"
[20] "BSgenome.Celegans.UCSC.ce11"
[21] "BSgenome.Celegans.UCSC.ce2"
[22] "BSgenome.Celegans.UCSC.ce6"
[23] "BSgenome.Cfamiliaris.UCSC.canFam2"
[24] "BSgenome.Cfamiliaris.UCSC.canFam2.masked"
[25] "BSgenome.Cfamiliaris.UCSC.canFam3"
[26] "BSgenome.Cfamiliaris.UCSC.canFam3.masked"
[27] "BSgenome.Cjacchus.UCSC.calJac3"
[28] "BSgenome.Cjacchus.UCSC.calJac4"
[29] "BSgenome.CneoformansVarGrubiiKN99.NCBI.ASM221672v1"
[30] "BSgenome.Creinhardtii.JGI.v5.6"
[31] "BSgenome.Dmelanogaster.UCSC.dm2"
[32] "BSgenome.Dmelanogaster.UCSC.dm2.masked"
[33] "BSgenome.Dmelanogaster.UCSC.dm3"
[34] "BSgenome.Dmelanogaster.UCSC.dm3.masked"
[35] "BSgenome.Dmelanogaster.UCSC.dm6"
[36] "BSgenome.Drerio.UCSC.danRer10"
[37] "BSgenome.Drerio.UCSC.danRer11"
[38] "BSgenome.Drerio.UCSC.danRer5"
[39] "BSgenome.Drerio.UCSC.danRer5.masked"
[40] "BSgenome.Drerio.UCSC.danRer6"
[41] "BSgenome.Drerio.UCSC.danRer6.masked"
[42] "BSgenome.Drerio.UCSC.danRer7"
[43] "BSgenome.Drerio.UCSC.danRer7.masked"
[44] "BSgenome.Dvirilis.Ensembl.dvircaf1"
[45] "BSgenome.Ecoli.NCBI.20080805"
[46] "BSgenome.Gaculeatus.UCSC.gasAcu1"
[47] "BSgenome.Gaculeatus.UCSC.gasAcu1.masked"
[48] "BSgenome.Ggallus.UCSC.galGal3"
[49] "BSgenome.Ggallus.UCSC.galGal3.masked"
[50] "BSgenome.Ggallus.UCSC.galGal4"
[51] "BSgenome.Ggallus.UCSC.galGal4.masked"
[52] "BSgenome.Ggallus.UCSC.galGal5"
[53] "BSgenome.Ggallus.UCSC.galGal6"
[54] "BSgenome.Gmax.NCBI.Gmv40"
[55] "BSgenome.Hsapiens.1000genomes.hs37d5"
[56] "BSgenome.Hsapiens.NCBI.GRCh38"
[57] "BSgenome.Hsapiens.NCBI.T2T.CHM13v2.0"
[58] "BSgenome.Hsapiens.UCSC.hg17"
[59] "BSgenome.Hsapiens.UCSC.hg17.masked"
[60] "BSgenome.Hsapiens.UCSC.hg18"
[61] "BSgenome.Hsapiens.UCSC.hg18.masked"
[62] "BSgenome.Hsapiens.UCSC.hg19"
[63] "BSgenome.Hsapiens.UCSC.hg19.masked"
[64] "BSgenome.Hsapiens.UCSC.hg38"
[65] "BSgenome.Hsapiens.UCSC.hg38.dbSNP151.major"
[66] "BSgenome.Hsapiens.UCSC.hg38.dbSNP151.minor"
[67] "BSgenome.Hsapiens.UCSC.hg38.masked"
[68] "BSgenome.Hsapiens.UCSC.hs1"
[69] "BSgenome.Mdomestica.UCSC.monDom5"
[70] "BSgenome.Mfascicularis.NCBI.5.0"
[71] "BSgenome.Mfascicularis.NCBI.6.0"
[72] "BSgenome.Mfuro.UCSC.musFur1"
[73] "BSgenome.Mmulatta.UCSC.rheMac10"
[74] "BSgenome.Mmulatta.UCSC.rheMac2"
[75] "BSgenome.Mmulatta.UCSC.rheMac2.masked"
[76] "BSgenome.Mmulatta.UCSC.rheMac3"
[77] "BSgenome.Mmulatta.UCSC.rheMac3.masked"
[78] "BSgenome.Mmulatta.UCSC.rheMac8"
[79] "BSgenome.Mmusculus.UCSC.mm10"
[80] "BSgenome.Mmusculus.UCSC.mm10.masked"
[81] "BSgenome.Mmusculus.UCSC.mm39"
[82] "BSgenome.Mmusculus.UCSC.mm8"
[83] "BSgenome.Mmusculus.UCSC.mm8.masked"
[84] "BSgenome.Mmusculus.UCSC.mm9"
[85] "BSgenome.Mmusculus.UCSC.mm9.masked"
[86] "BSgenome.Osativa.MSU.MSU7"
[87] "BSgenome.Ppaniscus.UCSC.panPan1"
[88] "BSgenome.Ppaniscus.UCSC.panPan2"
[89] "BSgenome.Ptroglodytes.UCSC.panTro2"
[90] "BSgenome.Ptroglodytes.UCSC.panTro2.masked"
[91] "BSgenome.Ptroglodytes.UCSC.panTro3"
[92] "BSgenome.Ptroglodytes.UCSC.panTro3.masked"
[93] "BSgenome.Ptroglodytes.UCSC.panTro5"
[94] "BSgenome.Ptroglodytes.UCSC.panTro6"
[95] "BSgenome.Rnorvegicus.UCSC.rn4"
[96] "BSgenome.Rnorvegicus.UCSC.rn4.masked"
[97] "BSgenome.Rnorvegicus.UCSC.rn5"
[98] "BSgenome.Rnorvegicus.UCSC.rn5.masked"
[99] "BSgenome.Rnorvegicus.UCSC.rn6"
[100] "BSgenome.Rnorvegicus.UCSC.rn7"
[101] "BSgenome.Scerevisiae.UCSC.sacCer1"
[102] "BSgenome.Scerevisiae.UCSC.sacCer2"
[103] "BSgenome.Scerevisiae.UCSC.sacCer3"
[104] "BSgenome.Sscrofa.UCSC.susScr11"
[105] "BSgenome.Sscrofa.UCSC.susScr3"
[106] "BSgenome.Sscrofa.UCSC.susScr3.masked"
[107] "BSgenome.Tgondii.ToxoDB.7.0"
[108] "BSgenome.Tguttata.UCSC.taeGut1"
[109] "BSgenome.Tguttata.UCSC.taeGut1.masked"
[110] "BSgenome.Tguttata.UCSC.taeGut2"
[111] "BSgenome.Vvinifera.URGI.IGGP12Xv0"
[112] "BSgenome.Vvinifera.URGI.IGGP12Xv2"
[113] "BSgenome.Vvinifera.URGI.IGGP8X" BSgenomeパッケージのインストール
利用可能なゲノムのいずれかを使用するには、まず対応するパッケージをインストールする必要があります。
たとえば、以下の例では、前に見た BiocManager::install()
関数を使用して、データパッケージ BSgenome.Hsapiens.UCSC.hg38.masked
をインストールする方法を示します。
R
BiocManager::install("BSgenome.Hsapiens.UCSC.hg38.masked")
BSgenomeパッケージの使用
一旦インストールされると、BSgenomeパッケージは他のRパッケージと同様に読み込むことができ、library()
関数を使用します。
R
library(BSgenome.Hsapiens.UCSC.hg38.masked)
各 BSgenome パッケージには、パッケージと同じ名前のオブジェクトが含まれ、そのオブジェクトにはゲノム配列が含まれています。
上記のパッケージ BSgenome.Hsapiens.UCSC.hg38.masked をロードしたので、BSgenome オブジェクトを次のように表示できます。
R
BSgenome.Hsapiens.UCSC.hg38.masked
OUTPUT
| BSgenome object for Human
| - organism: Homo sapiens
| - provider: UCSC
| - genome: hg38
| - release date: 2023/01/31
| - 711 sequence(s):
| chr1 chr2 chr3
| chr4 chr5 chr6
| chr7 chr8 chr9
| chr10 chr11 chr12
| chr13 chr14 chr15
| ... ... ...
| chr19_KV575256v1_alt chr19_KV575257v1_alt chr19_KV575258v1_alt
| chr19_KV575259v1_alt chr19_KV575260v1_alt chr19_MU273387v1_alt
| chr22_KN196485v1_alt chr22_KN196486v1_alt chr22_KQ458387v1_alt
| chr22_KQ458388v1_alt chr22_KQ759761v1_alt chrX_KV766199v1_alt
| chrX_MU273395v1_alt chrX_MU273396v1_alt chrX_MU273397v1_alt
|
| Tips: call 'seqnames()' on the object to get all the sequence names, call
| 'seqinfo()' to get the full sequence info, use the '$' or '[[' operator to
| access a given sequence, see '?BSgenome' for more information.オブジェクト名の長さと複雑さを考慮すると、BSgenome
オブジェクトのコピーを単に genome
という名前の新しいオブジェクトに割り当てるのが一般的な慣行です。
R
genome <- BSgenome.Hsapiens.UCSC.hg38.masked
BSgenome オブジェクトの使用
コンソールに BSgenome オブジェクトを印刷すると(上記参照)、オブジェクト自体の下に、オブジェクト内の情報にアクセスするためによく使用される関数のヒントが表示されます。
たとえば、関数 seqnames()
を使用して、オブジェクトに存在する配列名(すなわち、染色体とコンティグ)のリストを取得できます。
R
seqnames(genome)
OUTPUT
[1] "chr1" "chr2"
[3] "chr3" "chr4"
[5] "chr5" "chr6"
[7] "chr7" "chr8"
[9] "chr9" "chr10"
[11] "chr11" "chr12"
[13] "chr13" "chr14"
[15] "chr15" "chr16"
[17] "chr17" "chr18"
[19] "chr19" "chr20"
[21] "chr21" "chr22"
[23] "chrX" "chrY"
[25] "chrM" "chr1_GL383518v1_alt"
[27] "chr1_GL383519v1_alt" "chr1_GL383520v2_alt"
[29] "chr1_KI270759v1_alt" "chr1_KI270760v1_alt"
[31] "chr1_KI270761v1_alt" "chr1_KI270762v1_alt"
[33] "chr1_KI270763v1_alt" "chr1_KI270764v1_alt"
[35] "chr1_KI270765v1_alt" "chr1_KI270766v1_alt"
[37] "chr1_KI270892v1_alt" "chr2_GL383521v1_alt"
[39] "chr2_GL383522v1_alt" "chr2_GL582966v2_alt"
[41] "chr2_KI270767v1_alt" "chr2_KI270768v1_alt"
[43] "chr2_KI270769v1_alt" "chr2_KI270770v1_alt"
[45] "chr2_KI270771v1_alt" "chr2_KI270772v1_alt"
[47] "chr2_KI270773v1_alt" "chr2_KI270774v1_alt"
[49] "chr2_KI270775v1_alt" "chr2_KI270776v1_alt"
[51] "chr2_KI270893v1_alt" "chr2_KI270894v1_alt"
[53] "chr3_GL383526v1_alt" "chr3_JH636055v2_alt"
[55] "chr3_KI270777v1_alt" "chr3_KI270778v1_alt"
[57] "chr3_KI270779v1_alt" "chr3_KI270780v1_alt"
[59] "chr3_KI270781v1_alt" "chr3_KI270782v1_alt"
[61] "chr3_KI270783v1_alt" "chr3_KI270784v1_alt"
[63] "chr3_KI270895v1_alt" "chr3_KI270924v1_alt"
[65] "chr3_KI270934v1_alt" "chr3_KI270935v1_alt"
[67] "chr3_KI270936v1_alt" "chr3_KI270937v1_alt"
[69] "chr4_GL000257v2_alt" "chr4_GL383527v1_alt"
[71] "chr4_GL383528v1_alt" "chr4_KI270785v1_alt"
[73] "chr4_KI270786v1_alt" "chr4_KI270787v1_alt"
[75] "chr4_KI270788v1_alt" "chr4_KI270789v1_alt"
[77] "chr4_KI270790v1_alt" "chr4_KI270896v1_alt"
[79] "chr4_KI270925v1_alt" "chr5_GL339449v2_alt"
[81] "chr5_GL383530v1_alt" "chr5_GL383531v1_alt"
[83] "chr5_GL383532v1_alt" "chr5_GL949742v1_alt"
[85] "chr5_KI270791v1_alt" "chr5_KI270792v1_alt"
[87] "chr5_KI270793v1_alt" "chr5_KI270794v1_alt"
[89] "chr5_KI270795v1_alt" "chr5_KI270796v1_alt"
[91] "chr5_KI270897v1_alt" "chr5_KI270898v1_alt"
[93] "chr6_GL000250v2_alt" "chr6_GL000251v2_alt"
[95] "chr6_GL000252v2_alt" "chr6_GL000253v2_alt"
[97] "chr6_GL000254v2_alt" "chr6_GL000255v2_alt"
[99] "chr6_GL000256v2_alt" "chr6_GL383533v1_alt"
[101] "chr6_KB021644v2_alt" "chr6_KI270758v1_alt"
[103] "chr6_KI270797v1_alt" "chr6_KI270798v1_alt"
[105] "chr6_KI270799v1_alt" "chr6_KI270800v1_alt"
[107] "chr6_KI270801v1_alt" "chr6_KI270802v1_alt"
[109] "chr7_GL383534v2_alt" "chr7_KI270803v1_alt"
[111] "chr7_KI270804v1_alt" "chr7_KI270805v1_alt"
[113] "chr7_KI270806v1_alt" "chr7_KI270807v1_alt"
[115] "chr7_KI270808v1_alt" "chr7_KI270809v1_alt"
[117] "chr7_KI270899v1_alt" "chr8_KI270810v1_alt"
[119] "chr8_KI270811v1_alt" "chr8_KI270812v1_alt"
[121] "chr8_KI270813v1_alt" "chr8_KI270814v1_alt"
[123] "chr8_KI270815v1_alt" "chr8_KI270816v1_alt"
[125] "chr8_KI270817v1_alt" "chr8_KI270818v1_alt"
[127] "chr8_KI270819v1_alt" "chr8_KI270820v1_alt"
[129] "chr8_KI270821v1_alt" "chr8_KI270822v1_alt"
[131] "chr8_KI270900v1_alt" "chr8_KI270901v1_alt"
[133] "chr8_KI270926v1_alt" "chr9_GL383539v1_alt"
[135] "chr9_GL383540v1_alt" "chr9_GL383541v1_alt"
[137] "chr9_GL383542v1_alt" "chr9_KI270823v1_alt"
[139] "chr10_GL383545v1_alt" "chr10_GL383546v1_alt"
[141] "chr10_KI270824v1_alt" "chr10_KI270825v1_alt"
[143] "chr11_GL383547v1_alt" "chr11_JH159136v1_alt"
[145] "chr11_JH159137v1_alt" "chr11_KI270826v1_alt"
[147] "chr11_KI270827v1_alt" "chr11_KI270829v1_alt"
[149] "chr11_KI270830v1_alt" "chr11_KI270831v1_alt"
[151] "chr11_KI270832v1_alt" "chr11_KI270902v1_alt"
[153] "chr11_KI270903v1_alt" "chr11_KI270927v1_alt"
[155] "chr12_GL383549v1_alt" "chr12_GL383550v2_alt"
[157] "chr12_GL383551v1_alt" "chr12_GL383552v1_alt"
[159] "chr12_GL383553v2_alt" "chr12_GL877875v1_alt"
[161] "chr12_GL877876v1_alt" "chr12_KI270833v1_alt"
[163] "chr12_KI270834v1_alt" "chr12_KI270835v1_alt"
[165] "chr12_KI270836v1_alt" "chr12_KI270837v1_alt"
[167] "chr12_KI270904v1_alt" "chr13_KI270838v1_alt"
[169] "chr13_KI270839v1_alt" "chr13_KI270840v1_alt"
[171] "chr13_KI270841v1_alt" "chr13_KI270842v1_alt"
[173] "chr13_KI270843v1_alt" "chr14_KI270844v1_alt"
[175] "chr14_KI270845v1_alt" "chr14_KI270846v1_alt"
[177] "chr14_KI270847v1_alt" "chr15_GL383554v1_alt"
[179] "chr15_GL383555v2_alt" "chr15_KI270848v1_alt"
[181] "chr15_KI270849v1_alt" "chr15_KI270850v1_alt"
[183] "chr15_KI270851v1_alt" "chr15_KI270852v1_alt"
[185] "chr15_KI270905v1_alt" "chr15_KI270906v1_alt"
[187] "chr16_GL383556v1_alt" "chr16_GL383557v1_alt"
[189] "chr16_KI270853v1_alt" "chr16_KI270854v1_alt"
[191] "chr16_KI270855v1_alt" "chr16_KI270856v1_alt"
[193] "chr17_GL000258v2_alt" "chr17_GL383563v3_alt"
[195] "chr17_GL383564v2_alt" "chr17_GL383565v1_alt"
[197] "chr17_GL383566v1_alt" "chr17_JH159146v1_alt"
[199] "chr17_JH159147v1_alt" "chr17_JH159148v1_alt"
[201] "chr17_KI270857v1_alt" "chr17_KI270858v1_alt"
[203] "chr17_KI270859v1_alt" "chr17_KI270860v1_alt"
[205] "chr17_KI270861v1_alt" "chr17_KI270862v1_alt"
[207] "chr17_KI270907v1_alt" "chr17_KI270908v1_alt"
[209] "chr17_KI270909v1_alt" "chr17_KI270910v1_alt"
[211] "chr18_GL383567v1_alt" "chr18_GL383568v1_alt"
[213] "chr18_GL383569v1_alt" "chr18_GL383570v1_alt"
[215] "chr18_GL383571v1_alt" "chr18_GL383572v1_alt"
[217] "chr18_KI270863v1_alt" "chr18_KI270864v1_alt"
[219] "chr18_KI270911v1_alt" "chr18_KI270912v1_alt"
[221] "chr19_GL000209v2_alt" "chr19_GL383573v1_alt"
[223] "chr19_GL383574v1_alt" "chr19_GL383575v2_alt"
[225] "chr19_GL383576v1_alt" "chr19_GL949746v1_alt"
[227] "chr19_GL949747v2_alt" "chr19_GL949748v2_alt"
[229] "chr19_GL949749v2_alt" "chr19_GL949750v2_alt"
[231] "chr19_GL949751v2_alt" "chr19_GL949752v1_alt"
[233] "chr19_GL949753v2_alt" "chr19_KI270865v1_alt"
[235] "chr19_KI270866v1_alt" "chr19_KI270867v1_alt"
[237] "chr19_KI270868v1_alt" "chr19_KI270882v1_alt"
[239] "chr19_KI270883v1_alt" "chr19_KI270884v1_alt"
[241] "chr19_KI270885v1_alt" "chr19_KI270886v1_alt"
[243] "chr19_KI270887v1_alt" "chr19_KI270888v1_alt"
[245] "chr19_KI270889v1_alt" "chr19_KI270890v1_alt"
[247] "chr19_KI270891v1_alt" "chr19_KI270914v1_alt"
[249] "chr19_KI270915v1_alt" "chr19_KI270916v1_alt"
[251] "chr19_KI270917v1_alt" "chr19_KI270918v1_alt"
[253] "chr19_KI270919v1_alt" "chr19_KI270920v1_alt"
[255] "chr19_KI270921v1_alt" "chr19_KI270922v1_alt"
[257] "chr19_KI270923v1_alt" "chr19_KI270929v1_alt"
[259] "chr19_KI270930v1_alt" "chr19_KI270931v1_alt"
[261] "chr19_KI270932v1_alt" "chr19_KI270933v1_alt"
[263] "chr19_KI270938v1_alt" "chr20_GL383577v2_alt"
[265] "chr20_KI270869v1_alt" "chr20_KI270870v1_alt"
[267] "chr20_KI270871v1_alt" "chr21_GL383578v2_alt"
[269] "chr21_GL383579v2_alt" "chr21_GL383580v2_alt"
[271] "chr21_GL383581v2_alt" "chr21_KI270872v1_alt"
[273] "chr21_KI270873v1_alt" "chr21_KI270874v1_alt"
[275] "chr22_GL383582v2_alt" "chr22_GL383583v2_alt"
[277] "chr22_KB663609v1_alt" "chr22_KI270875v1_alt"
[279] "chr22_KI270876v1_alt" "chr22_KI270877v1_alt"
[281] "chr22_KI270878v1_alt" "chr22_KI270879v1_alt"
[283] "chr22_KI270928v1_alt" "chrX_KI270880v1_alt"
[285] "chrX_KI270881v1_alt" "chrX_KI270913v1_alt"
[287] "chr1_KI270706v1_random" "chr1_KI270707v1_random"
[289] "chr1_KI270708v1_random" "chr1_KI270709v1_random"
[291] "chr1_KI270710v1_random" "chr1_KI270711v1_random"
[293] "chr1_KI270712v1_random" "chr1_KI270713v1_random"
[295] "chr1_KI270714v1_random" "chr2_KI270715v1_random"
[297] "chr2_KI270716v1_random" "chr3_GL000221v1_random"
[299] "chr4_GL000008v2_random" "chr5_GL000208v1_random"
[301] "chr9_KI270717v1_random" "chr9_KI270718v1_random"
[303] "chr9_KI270719v1_random" "chr9_KI270720v1_random"
[305] "chr11_KI270721v1_random" "chr14_GL000009v2_random"
[307] "chr14_GL000194v1_random" "chr14_GL000225v1_random"
[309] "chr14_KI270722v1_random" "chr14_KI270723v1_random"
[311] "chr14_KI270724v1_random" "chr14_KI270725v1_random"
[313] "chr14_KI270726v1_random" "chr15_KI270727v1_random"
[315] "chr16_KI270728v1_random" "chr17_GL000205v2_random"
[317] "chr17_KI270729v1_random" "chr17_KI270730v1_random"
[319] "chr22_KI270731v1_random" "chr22_KI270732v1_random"
[321] "chr22_KI270733v1_random" "chr22_KI270734v1_random"
[323] "chr22_KI270735v1_random" "chr22_KI270736v1_random"
[325] "chr22_KI270737v1_random" "chr22_KI270738v1_random"
[327] "chr22_KI270739v1_random" "chrY_KI270740v1_random"
[329] "chrUn_GL000195v1" "chrUn_GL000213v1"
[331] "chrUn_GL000214v1" "chrUn_GL000216v2"
[333] "chrUn_GL000218v1" "chrUn_GL000219v1"
[335] "chrUn_GL000220v1" "chrUn_GL000224v1"
[337] "chrUn_GL000226v1" "chrUn_KI270302v1"
[339] "chrUn_KI270303v1" "chrUn_KI270304v1"
[341] "chrUn_KI270305v1" "chrUn_KI270310v1"
[343] "chrUn_KI270311v1" "chrUn_KI270312v1"
[345] "chrUn_KI270315v1" "chrUn_KI270316v1"
[347] "chrUn_KI270317v1" "chrUn_KI270320v1"
[349] "chrUn_KI270322v1" "chrUn_KI270329v1"
[351] "chrUn_KI270330v1" "chrUn_KI270333v1"
[353] "chrUn_KI270334v1" "chrUn_KI270335v1"
[355] "chrUn_KI270336v1" "chrUn_KI270337v1"
[357] "chrUn_KI270338v1" "chrUn_KI270340v1"
[359] "chrUn_KI270362v1" "chrUn_KI270363v1"
[361] "chrUn_KI270364v1" "chrUn_KI270366v1"
[363] "chrUn_KI270371v1" "chrUn_KI270372v1"
[365] "chrUn_KI270373v1" "chrUn_KI270374v1"
[367] "chrUn_KI270375v1" "chrUn_KI270376v1"
[369] "chrUn_KI270378v1" "chrUn_KI270379v1"
[371] "chrUn_KI270381v1" "chrUn_KI270382v1"
[373] "chrUn_KI270383v1" "chrUn_KI270384v1"
[375] "chrUn_KI270385v1" "chrUn_KI270386v1"
[377] "chrUn_KI270387v1" "chrUn_KI270388v1"
[379] "chrUn_KI270389v1" "chrUn_KI270390v1"
[381] "chrUn_KI270391v1" "chrUn_KI270392v1"
[383] "chrUn_KI270393v1" "chrUn_KI270394v1"
[385] "chrUn_KI270395v1" "chrUn_KI270396v1"
[387] "chrUn_KI270411v1" "chrUn_KI270412v1"
[389] "chrUn_KI270414v1" "chrUn_KI270417v1"
[391] "chrUn_KI270418v1" "chrUn_KI270419v1"
[393] "chrUn_KI270420v1" "chrUn_KI270422v1"
[395] "chrUn_KI270423v1" "chrUn_KI270424v1"
[397] "chrUn_KI270425v1" "chrUn_KI270429v1"
[399] "chrUn_KI270435v1" "chrUn_KI270438v1"
[401] "chrUn_KI270442v1" "chrUn_KI270448v1"
[403] "chrUn_KI270465v1" "chrUn_KI270466v1"
[405] "chrUn_KI270467v1" "chrUn_KI270468v1"
[407] "chrUn_KI270507v1" "chrUn_KI270508v1"
[409] "chrUn_KI270509v1" "chrUn_KI270510v1"
[411] "chrUn_KI270511v1" "chrUn_KI270512v1"
[413] "chrUn_KI270515v1" "chrUn_KI270516v1"
[415] "chrUn_KI270517v1" "chrUn_KI270518v1"
[417] "chrUn_KI270519v1" "chrUn_KI270521v1"
[419] "chrUn_KI270522v1" "chrUn_KI270528v1"
[421] "chrUn_KI270529v1" "chrUn_KI270530v1"
[423] "chrUn_KI270538v1" "chrUn_KI270539v1"
[425] "chrUn_KI270544v1" "chrUn_KI270548v1"
[427] "chrUn_KI270579v1" "chrUn_KI270580v1"
[429] "chrUn_KI270581v1" "chrUn_KI270582v1"
[431] "chrUn_KI270583v1" "chrUn_KI270584v1"
[433] "chrUn_KI270587v1" "chrUn_KI270588v1"
[435] "chrUn_KI270589v1" "chrUn_KI270590v1"
[437] "chrUn_KI270591v1" "chrUn_KI270593v1"
[439] "chrUn_KI270741v1" "chrUn_KI270742v1"
[441] "chrUn_KI270743v1" "chrUn_KI270744v1"
[443] "chrUn_KI270745v1" "chrUn_KI270746v1"
[445] "chrUn_KI270747v1" "chrUn_KI270748v1"
[447] "chrUn_KI270749v1" "chrUn_KI270750v1"
[449] "chrUn_KI270751v1" "chrUn_KI270752v1"
[451] "chrUn_KI270753v1" "chrUn_KI270754v1"
[453] "chrUn_KI270755v1" "chrUn_KI270756v1"
[455] "chrUn_KI270757v1" "chr1_KN196472v1_fix"
[457] "chr1_KN196473v1_fix" "chr1_KN196474v1_fix"
[459] "chr1_KN538360v1_fix" "chr1_KN538361v1_fix"
[461] "chr1_KQ031383v1_fix" "chr1_KZ208906v1_fix"
[463] "chr1_KZ559100v1_fix" "chr1_MU273333v1_fix"
[465] "chr1_MU273334v1_fix" "chr1_MU273335v1_fix"
[467] "chr1_MU273336v1_fix" "chr2_KN538362v1_fix"
[469] "chr2_KN538363v1_fix" "chr2_KQ031384v1_fix"
[471] "chr2_ML143341v1_fix" "chr2_ML143342v1_fix"
[473] "chr2_MU273341v1_fix" "chr2_MU273342v1_fix"
[475] "chr2_MU273343v1_fix" "chr2_MU273344v1_fix"
[477] "chr2_MU273345v1_fix" "chr3_KN196475v1_fix"
[479] "chr3_KN196476v1_fix" "chr3_KN538364v1_fix"
[481] "chr3_KQ031385v1_fix" "chr3_KQ031386v1_fix"
[483] "chr3_KV766192v1_fix" "chr3_KZ559104v1_fix"
[485] "chr3_MU273346v1_fix" "chr3_MU273347v1_fix"
[487] "chr3_MU273348v1_fix" "chr4_KQ983257v1_fix"
[489] "chr4_ML143344v1_fix" "chr4_ML143345v1_fix"
[491] "chr4_ML143346v1_fix" "chr4_ML143347v1_fix"
[493] "chr4_ML143348v1_fix" "chr4_ML143349v1_fix"
[495] "chr4_MU273350v1_fix" "chr4_MU273351v1_fix"
[497] "chr5_KV575244v1_fix" "chr5_ML143350v1_fix"
[499] "chr5_MU273352v1_fix" "chr5_MU273353v1_fix"
[501] "chr5_MU273354v1_fix" "chr5_MU273355v1_fix"
[503] "chr6_KN196478v1_fix" "chr6_KQ031387v1_fix"
[505] "chr6_KQ090016v1_fix" "chr6_KV766194v1_fix"
[507] "chr6_KZ208911v1_fix" "chr6_ML143351v1_fix"
[509] "chr7_KQ031388v1_fix" "chr7_KV880764v1_fix"
[511] "chr7_KV880765v1_fix" "chr7_KZ208912v1_fix"
[513] "chr7_ML143352v1_fix" "chr8_KV880766v1_fix"
[515] "chr8_KV880767v1_fix" "chr8_KZ208914v1_fix"
[517] "chr8_KZ208915v1_fix" "chr8_MU273359v1_fix"
[519] "chr8_MU273360v1_fix" "chr8_MU273361v1_fix"
[521] "chr8_MU273362v1_fix" "chr8_MU273363v1_fix"
[523] "chr9_KN196479v1_fix" "chr9_ML143353v1_fix"
[525] "chr9_MU273364v1_fix" "chr9_MU273365v1_fix"
[527] "chr9_MU273366v1_fix" "chr10_KN196480v1_fix"
[529] "chr10_KN538365v1_fix" "chr10_KN538366v1_fix"
[531] "chr10_KN538367v1_fix" "chr10_KQ090021v1_fix"
[533] "chr10_ML143354v1_fix" "chr10_ML143355v1_fix"
[535] "chr10_MU273367v1_fix" "chr11_KN196481v1_fix"
[537] "chr11_KQ090022v1_fix" "chr11_KQ759759v1_fix"
[539] "chr11_KQ759759v2_fix" "chr11_KV766195v1_fix"
[541] "chr11_KZ559108v1_fix" "chr11_KZ559109v1_fix"
[543] "chr11_ML143356v1_fix" "chr11_ML143357v1_fix"
[545] "chr11_ML143358v1_fix" "chr11_ML143359v1_fix"
[547] "chr11_ML143360v1_fix" "chr11_MU273369v1_fix"
[549] "chr11_MU273370v1_fix" "chr11_MU273371v1_fix"
[551] "chr12_KN196482v1_fix" "chr12_KN538369v1_fix"
[553] "chr12_KN538370v1_fix" "chr12_KQ759760v1_fix"
[555] "chr12_KZ208916v1_fix" "chr12_KZ208917v1_fix"
[557] "chr12_ML143361v1_fix" "chr12_ML143362v1_fix"
[559] "chr12_MU273372v1_fix" "chr13_KN196483v1_fix"
[561] "chr13_KN538371v1_fix" "chr13_KN538372v1_fix"
[563] "chr13_KN538373v1_fix" "chr13_ML143363v1_fix"
[565] "chr13_ML143364v1_fix" "chr13_ML143365v1_fix"
[567] "chr13_ML143366v1_fix" "chr14_KZ208920v1_fix"
[569] "chr14_ML143367v1_fix" "chr14_MU273373v1_fix"
[571] "chr15_KN538374v1_fix" "chr15_ML143369v1_fix"
[573] "chr15_ML143370v1_fix" "chr15_ML143371v1_fix"
[575] "chr15_ML143372v1_fix" "chr15_MU273374v1_fix"
[577] "chr16_KV880768v1_fix" "chr16_KZ559113v1_fix"
[579] "chr16_ML143373v1_fix" "chr16_MU273376v1_fix"
[581] "chr16_MU273377v1_fix" "chr17_KV575245v1_fix"
[583] "chr17_KV766196v1_fix" "chr17_ML143374v1_fix"
[585] "chr17_ML143375v1_fix" "chr17_MU273379v1_fix"
[587] "chr17_MU273380v1_fix" "chr17_MU273381v1_fix"
[589] "chr17_MU273382v1_fix" "chr17_MU273383v1_fix"
[591] "chr18_KQ090028v1_fix" "chr18_KZ208922v1_fix"
[593] "chr18_KZ559115v1_fix" "chr19_KN196484v1_fix"
[595] "chr19_KQ458386v1_fix" "chr19_ML143376v1_fix"
[597] "chr19_MU273384v1_fix" "chr19_MU273385v1_fix"
[599] "chr19_MU273386v1_fix" "chr20_MU273388v1_fix"
[601] "chr20_MU273389v1_fix" "chr21_ML143377v1_fix"
[603] "chr21_MU273390v1_fix" "chr21_MU273391v1_fix"
[605] "chr21_MU273392v1_fix" "chr22_KQ759762v1_fix"
[607] "chr22_KQ759762v2_fix" "chr22_ML143378v1_fix"
[609] "chr22_ML143379v1_fix" "chr22_ML143380v1_fix"
[611] "chrX_ML143381v1_fix" "chrX_ML143382v1_fix"
[613] "chrX_ML143383v1_fix" "chrX_ML143384v1_fix"
[615] "chrX_ML143385v1_fix" "chrX_MU273393v1_fix"
[617] "chrX_MU273394v1_fix" "chrY_KN196487v1_fix"
[619] "chrY_KZ208923v1_fix" "chrY_KZ208924v1_fix"
[621] "chrY_MU273398v1_fix" "chr1_KQ458382v1_alt"
[623] "chr1_KQ458383v1_alt" "chr1_KQ458384v1_alt"
[625] "chr1_KQ983255v1_alt" "chr1_KV880763v1_alt"
[627] "chr1_KZ208904v1_alt" "chr1_KZ208905v1_alt"
[629] "chr1_MU273330v1_alt" "chr1_MU273331v1_alt"
[631] "chr1_MU273332v1_alt" "chr2_KQ983256v1_alt"
[633] "chr2_KZ208907v1_alt" "chr2_KZ208908v1_alt"
[635] "chr2_MU273337v1_alt" "chr2_MU273338v1_alt"
[637] "chr2_MU273339v1_alt" "chr2_MU273340v1_alt"
[639] "chr3_KZ208909v1_alt" "chr3_KZ559101v1_alt"
[641] "chr3_KZ559102v1_alt" "chr3_KZ559103v1_alt"
[643] "chr3_KZ559105v1_alt" "chr3_ML143343v1_alt"
[645] "chr4_KQ090013v1_alt" "chr4_KQ090014v1_alt"
[647] "chr4_KQ090015v1_alt" "chr4_KQ983258v1_alt"
[649] "chr4_KV766193v1_alt" "chr4_MU273349v1_alt"
[651] "chr5_KN196477v1_alt" "chr5_KV575243v1_alt"
[653] "chr5_KZ208910v1_alt" "chr5_MU273356v1_alt"
[655] "chr6_KQ090017v1_alt" "chr6_MU273357v1_alt"
[657] "chr7_KZ208913v1_alt" "chr7_KZ559106v1_alt"
[659] "chr7_MU273358v1_alt" "chr8_KZ559107v1_alt"
[661] "chr9_KQ090018v1_alt" "chr9_KQ090019v1_alt"
[663] "chr10_KQ090020v1_alt" "chr11_KN538368v1_alt"
[665] "chr11_KZ559110v1_alt" "chr11_KZ559111v1_alt"
[667] "chr11_MU273368v1_alt" "chr12_KQ090023v1_alt"
[669] "chr12_KZ208918v1_alt" "chr12_KZ559112v1_alt"
[671] "chr13_KQ090024v1_alt" "chr13_KQ090025v1_alt"
[673] "chr14_KZ208919v1_alt" "chr14_ML143368v1_alt"
[675] "chr15_KQ031389v1_alt" "chr15_MU273375v1_alt"
[677] "chr16_KQ031390v1_alt" "chr16_KQ090026v1_alt"
[679] "chr16_KQ090027v1_alt" "chr16_KZ208921v1_alt"
[681] "chr17_KV766197v1_alt" "chr17_KV766198v1_alt"
[683] "chr17_KZ559114v1_alt" "chr17_MU273378v1_alt"
[685] "chr18_KQ458385v1_alt" "chr18_KZ559116v1_alt"
[687] "chr19_KV575246v1_alt" "chr19_KV575247v1_alt"
[689] "chr19_KV575248v1_alt" "chr19_KV575249v1_alt"
[691] "chr19_KV575250v1_alt" "chr19_KV575251v1_alt"
[693] "chr19_KV575252v1_alt" "chr19_KV575253v1_alt"
[695] "chr19_KV575254v1_alt" "chr19_KV575255v1_alt"
[697] "chr19_KV575256v1_alt" "chr19_KV575257v1_alt"
[699] "chr19_KV575258v1_alt" "chr19_KV575259v1_alt"
[701] "chr19_KV575260v1_alt" "chr19_MU273387v1_alt"
[703] "chr22_KN196485v1_alt" "chr22_KN196486v1_alt"
[705] "chr22_KQ458387v1_alt" "chr22_KQ458388v1_alt"
[707] "chr22_KQ759761v1_alt" "chrX_KV766199v1_alt"
[709] "chrX_MU273395v1_alt" "chrX_MU273396v1_alt"
[711] "chrX_MU273397v1_alt" 同様に、関数 seqinfo()
を使用して、オブジェクトに保存されている完全な配列情報を取得できます。
R
seqinfo(genome)
OUTPUT
Seqinfo object with 711 sequences (1 circular) from hg38 genome:
seqnames seqlengths isCircular genome
chr1 248956422 FALSE hg38
chr2 242193529 FALSE hg38
chr3 198295559 FALSE hg38
chr4 190214555 FALSE hg38
chr5 181538259 FALSE hg38
... ... ... ...
chr22_KQ759761v1_alt 145162 FALSE hg38
chrX_KV766199v1_alt 188004 FALSE hg38
chrX_MU273395v1_alt 619716 FALSE hg38
chrX_MU273396v1_alt 294119 FALSE hg38
chrX_MU273397v1_alt 330493 FALSE hg38最後に、BSgenome
オブジェクトの性質は配列のリストに似ているため、演算子 $
および [[]] を使用して、BSgenome
オブジェクトから個々の配列を抽出することができます。
R
genome$chr1
OUTPUT
248956422-letter MaskedDNAString object (# for masking)
seq: ####################################...####################################
masks:
maskedwidth maskedratio active names desc
1 18470101 7.419010e-02 TRUE AGAPS assembly gaps
2 5309 2.132502e-05 TRUE AMB intra-contig ambiguities
3 119060341 4.782377e-01 FALSE RM RepeatMasker
4 1647959 6.619468e-03 FALSE TRF Tandem Repeats Finder [period<=12]
all masks together:
maskedwidth maskedratio
137685771 0.5530517
all active masks together:
maskedwidth maskedratio
18475410 0.07421142例えば、Y 染色体の配列を抽出して新しいオブジェクト chrY
に割り当てることができます。
R
chrY <- genome[["chrY"]]
ゲノム配列の使用
この時点から、ゲノム配列は以前に説明した生物学的文字列(例:DNAString)のように扱うことができます。
Biostrings
パッケージで。
たとえば、関数 countPattern()
を使用して、特定のゲノム配列内の特定のパターンの出現回数をカウントできます。
R
countPattern(pattern = "CANNTG", subject = chrY, fixed = FALSE)
OUTPUT
[1] 141609-
Biostringsパッケージは、ヌクレオチドおよびアミノ酸の配列を表すクラスを定義します。 -
Biostringsパッケージは、生物学的配列を効率的に処理するためのメソッドも定義します。 -
BSgenomeパッケージは、さまざまなモデル生物のゲノム配列を、Bioconductor オブジェクトとして即座に利用できるように提供します。
Content from Genomics ranges の取り扱い
Last updated on 2025-09-23 | Edit this page
Overview
Questions
- Bioconductorでゲノムスケールの座標を表現する推奨方法は何ですか?
- ゲノムの範囲を効率的に処理するためのメソッドを提供するBioconductorパッケージはどれですか?
- さまざまなゲノムファイル形式から/へのゲノム座標のセットをインポート/エクスポートする方法は?
Objectives
- ゲノム座標と区間がBioconductorプロジェクトでどのように表現されるかを説明します。
- ゲノム座標の範囲を処理するために利用可能なBioconductorパッケージおよびメソッドを特定します。
パッケージのインストール
次のセクションに進む前に、必要となるいくつかのBioconductorパッケージをインストールします。
まず、BiocManagerパッケージがインストールされているか確認し、ない場合はインストールします。
次に、BiocManager::install()関数を使用して必要なパッケージをインストールします。
R
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("GenomicRanges")
GenomicRangesパッケージとクラス
ゲノム範囲のためにクラスが必要な理由は?
ゲノミクスの時代に、多くの観察は遺伝子の座標の範囲、すなわちゲノムスケールの区間として報告されます。 アッセイの性質に応じて、これらのゲノム範囲は遺伝子、トランスクリプト、エクソン、一塩基多型(SNP)、転写因子結合部位、またはChIP-seqやATAC-seqなどの次世代シーケンシングアッセイからのピークを表す場合があります。
ゲノム範囲は、アッセイされた値(例:遺伝子発現)の観察をゲノムまたは生物内の物理的位置に結びつけます。 例えば、これらのゲノム範囲は、アッセイされた特徴と既知の調節領域のデータベースとの物理的近接性や重複を照会するために使用することができます。
最終的に報告される測定値に使用されるゲノム範囲は、既知のゲノム特徴のデータベース内のゲノム範囲のセットに対する組み合わせや操作の結果であることが多いです。 例えば、RNAシーケンシングにおいて、次世代シーケンシングリードはしばしば個々のエクソン内でカウントされ、それらのカウントは後に各遺伝子のすべてのエクソンで集約されます。 また、プロモーターは、既知の転写開始サイト(TSS)の上流および/または下流の任意の幅の領域として定義されることが多いです。
重要なことに、ゲノム範囲は必ずしも複数の座標を跨ぐ必要はありません。 範囲の概念は数学的な方法で意味されており、単一塩基のゲノム範囲(例:SNP)は、同じ座標で開閉するか(または右開区間の場合は次の座標で開閉する)と説明できます。
多くの生物において、遺伝物質は複数の別々の核酸分子(例:染色体、プラスミド)に分割されています。 そのため、ゲノム範囲は配列の名前とその配列上の座標の数値区間によって記述されます。
GenomicRanges代数の使用例。 Huber, Carey, Gentleman, Anders, Carlson, Carvalho, Bravo, Davis, Gatto, Girke, Gottardo, Hahne, Hansen, Irizarry, Lawrence, Love, MacDonald, Obenchain, Oles, Pages, Reyes, Shannon, Smyth, Tenenbaum, Waldron, and Morgan (2015)から適応。 この図は、2つのトランスクリプトからなる遺伝子モデルの例と、その遺伝子モデルに対するさまざまなゲノム範囲の定義を示しています。 例えば、特定の図示において、スプライスされていないトランスクリプトは、最初のエクソンの開始から最後のエクソンの終了までのすべての座標の範囲を要約します。一方、遺伝子領域は、少なくとも1つのトランスクリプトの1エクソンに含まれる座標のセットとして定義されます。
区間の簡単な紹介
区間は、連続した座標の軸上の開始位置と終了位置を使用して数学的に説明されます。 区間は、その二つの座標の間のすべての実数を含み、各区間の幅は開始位置と終了位置の座標の差から計算できます。
一般的に、開始と終了の位置は、任意の有理数、包括浮動小数点数になり得ます。 ただし、ゲノム学では、整数座標は通常、ポリマー(例:核酸、タンパク質)内のモノマー(例:ヌクレオチド、アミノ酸)の位置を表すために使用されます。
さまざまなルールを使用して区間を定義するパッケージ、データベース、プログラミング言語に出会うかもしれません。 Rでは、インデックスは1ベース(すなわちシーケンスの最初の位置は1)ですが、Pythonは0ベース(すなわちシーケンスの最初の位置のインデックスは0)です。 同様に、UCSCゲノムブラウザの参照ファイルは0ベースであり、Ensemblゲノムブラウザのものは1ベースです。
共有座標系における区間の定義は、二つの区間間の距離を計算したり、重複する区間を特定したりするなどの計算を可能にします。
区間の例。 A、B、およびCという名前の3つの区間が表されています。 区間Aは位置5から始まり、位置9で終わり、幅は4ユニットです。 区間Bは位置1から始まり、位置3で終わり、幅は2ユニットです。 区間Cは位置3から始まり、位置6で終わり、幅は3ユニットです。 区間AとCは、座標5から6の間で重複し、一方、区間BとCは座標3で会い、厳密には互いに重複していません。
ゲノム範囲の簡単な紹介
ゲノム範囲は、本質的に生物学的シーケンス(例:染色体)上の数学的な区間の概念を拡張します。 つまり、ゲノム範囲は、それが存在する生物学的配列の名前と、その配列内でゲノム範囲が跨ぐ整数座標の範囲を結合します。 これは、異なる生物学的配列上の重複する範囲のゲノム特徴を区別するための重要な要素です。
さらに、DNA配列の二重鎖特性も、ゲノム範囲に対してストランド性の概念を加えます。 もし知られている場合、ゲノム特徴のストランド情報は重要な情報であり、追跡されるべきで、後続の解析に使用されるかもしれません。 たとえば、同じDNA配列の反対のストランドで共通の座標範囲を跨ぐゲノム範囲は、重なっていると見なされない場合があります(例:ストランド特異的な次世代シーケンシングアッセイの目的のため)。
ゲノム範囲は_閉じた_区間です - 開始位置と終了位置は区間に含まれます。核酸の例では、開始位置は区間内の最初のヌクレオチドを示し、終了位置は区間内の最後のヌクレオチドを示します。
ゲノム範囲の例。 ゲノム範囲は、存在する生物学的配列の名前(ここでは「chr1」)と、その配列内での開始と終了の位置で定義されます。 ここで、数値位置は明示的に示されていませんが、核酸のシーケンスと座標が左から右に増加していることを示す矢印によって暗示されています。 この例では、ゲノム範囲を使用して個々のエクソンを記述でき、メタデータはそれらのエクソンをトランスクリプトと遺伝子にグループ化します。 さらに、エクソン、トランスクリプト、遺伝子のストランド性は、各ゲノム範囲の正確な位置を記述するための重要な情報です。
GenomicRangesパッケージ
概要
GenomicRangesパッケージは、ゲノム範囲をS4オブジェクトとして表現するためのS4クラスを実装しています。
具体的には、GRangesクラスは、機能が存在するシーケンスの名前と、そのシーケンス内で機能が跨ぐ整数座標の範囲を含む区間のセットを保存するために設計されています。
より一般的には、IRangesクラスは、シーケンス名の概念なしで、整数座標の範囲にわたる区間のセットを保存するために設計されています。
このように、GRangesオブジェクトは、IRangesオブジェクトとシーケンス名のベクターを組み合わせたものであるに過ぎません。
これらのS4クラスは、自動的な有効性チェック機能を提供し、整数区間およびゲノム範囲上の一般的な操作を実装するさまざまなメソッドを提供します。 区間間の距離の計算から重複するゲノム範囲の特定まで。
GenomicRangesパッケージで定義されている基本クラスの簡単な説明は、パッケージのヴィネットの1つに利用可能で、vignette("GenomicRangesIntroduction")としてアクセス可能。本より詳細な情報は、他のパッケージのヴィネットに提供され、browseVignettes("GenomicRanges")としてアクセス可能です。
IRangesクラス
多くの生物のゲノム空間は複数の配列(例:染色体)に分割されていますが、ゲノム範囲上での多くの操作は、整数位置のみが重要な個々の配列内で行われます。
IRangesクラスは、次の三つの情報のうち二つに基づいて定義される「単純な」範囲のためのコンテナを提供します:
- 範囲の開始位置
- 範囲の幅
- 範囲の終了位置
IRanges()コンストラクタ関数は、start=,
width=,
end=の引数でこれら三つの情報を受け入れます。
例えば、開始位置と幅から二つの整数範囲を作成します:
- ある範囲は位置10から始まり、幅は10です。
- 別の範囲は位置15から始まり、幅は5です。
R
demo_iranges <- IRanges(start = c(10, 15), width = c(10, 5))
demo_iranges
OUTPUT
IRanges object with 2 ranges and 0 metadata columns:
start end width
<integer> <integer> <integer>
[1] 10 19 10
[2] 15 19 5オブジェクトは、要求した_開始_と_幅_の情報だけでなく、他の二つの情報から自然に算出される_終了_位置も表示されることに注意します。
チャレンジ
IRanges()コンストラクタ関数のstart=およびend=の引数を使用して、上記と同じ二つの範囲を作成します。
R
IRanges(start = c(10, 15), end = c(19, 19))
OUTPUT
IRanges object with 2 ranges and 0 metadata columns:
start end width
<integer> <integer> <integer>
[1] 10 19 10
[2] 15 19 5すべての区間の開始および終了位置、ならびに各区間の幅は、start(),
end()およびwidth()関数を使用して数値ベクターとして抽出できます。
R
start(demo_iranges)
OUTPUT
[1] 10 15R
end(demo_iranges)
OUTPUT
[1] 19 19R
width(demo_iranges)
OUTPUT
[1] 10 5IRangesファミリーのオブジェクトはVectorクラスを拡張し、インデックスの観点から一次元ベクターとして扱われます。
そのため、個々の範囲は通常のベクターのように整数インデックスで抽出できます。
R
demo_iranges[1]
OUTPUT
IRanges object with 1 range and 0 metadata columns:
start end width
<integer> <integer> <integer>
[1] 10 19 10IRangesのメタデータ
IRangesクラスは、各範囲のメタデータ情報を収容でき、名前はnames=引数に渡され、さまざまなメタデータは名前付きベクターとして渡されます。
例えば、“A”と”B”という名前の二つの範囲を作成します。 さらに、例として文字値と数値を保存するメタデータフィールドを定義します。 この例では、メタデータフィールドの名前と値は完全に任意です。
R
demo_with_metadata <- IRanges(
start = c(10, 15),
end = c(19, 19),
names = c("A", "B"),
character_metadata = c("control", "target"),
numeric_metadata = c(100, 200)
)
demo_with_metadata
OUTPUT
IRanges object with 2 ranges and 2 metadata columns:
start end width | character_metadata numeric_metadata
<integer> <integer> <integer> | <character> <numeric>
A 10 19 10 | control 100
B 15 19 5 | target 200メタデータ列は、mcols()関数を使用してDataFrameとして抽出できます(“メタデータ列”の略)。
R
mcols(demo_with_metadata)
OUTPUT
DataFrame with 2 rows and 2 columns
character_metadata numeric_metadata
<character> <numeric>
A control 100
B target 200名前の文字ベクターは、names()関数を使用して抽出できます。
R
names(demo_with_metadata)
OUTPUT
[1] "A" "B"基本データ型の名前付きベクターに類似して、個々の範囲は名前で抽出できます。
R
demo_with_metadata["A"]
OUTPUT
IRanges object with 1 range and 2 metadata columns:
start end width | character_metadata numeric_metadata
<integer> <integer> <integer> | <character> <numeric>
A 10 19 10 | control 100IRangesでの操作
IRangesは、数値座標の範囲に対するほとんどの操作の基礎を提供します。
例えば、クエリセットとサブジェクトセットの二つの範囲が与えられた場合、findOVerlaps()関数を使用して、二つのセットの範囲のどのペアが互いに重なっているかを確認できます。
R
query_iranges <- IRanges(
start = c(8, 16),
end = c(14, 18)
)
overlaps_iranges <- findOverlaps(query = query_iranges, subject = demo_iranges)
overlaps_iranges
OUTPUT
Hits object with 3 hits and 0 metadata columns:
queryHits subjectHits
<integer> <integer>
[1] 1 1
[2] 2 1
[3] 2 2
-------
queryLength: 2 / subjectLength: 2結果は、まだ紹介していないHitsオブジェクトの形式で返されます。
Hitsオブジェクトは、queryHitsおよびsubjectHitsという二つの整数列からなるテーブルとして視覚化されます。
テーブルの各行は、クエリセット内の1つの範囲とサブジェクトセット内の1つの範囲の重複を報告し、各列の整数値は重複に関与する各セット内の範囲のインデックスを示します。
この例では、クエリセットの最初の範囲がサブジェクトセットの最初の範囲と重なっていることを確認し、クエリセットの二番目の範囲がサブジェクトセット内の両範囲と重なっています。
さらに深く
後処理用に、Hitsオブジェクトから二つのコンポーネントを、それぞれの名前を使用して抽出できます:
R
queryHits(overlaps_iranges)
OUTPUT
[1] 1 2 2R
subjectHits(overlaps_iranges)
OUTPUT
[1] 1 1 2テーブルとして表示される間、Hitsオブジェクトは実際にはベクターのように処理されます。
クエリ範囲とサブジェクト範囲の間の個々のヒットは、インデックスで抽出できます:
R
overlaps_iranges[1]
OUTPUT
Hits object with 1 hit and 0 metadata columns:
queryHits subjectHits
<integer> <integer>
[1] 1 1
-------
queryLength: 2 / subjectLength: 2GRangesクラス
整数範囲を定義した後、ゲノム範囲を定義するために必要な追加情報は、各範囲が位置するゲノム配列の名前です。
例えば、次のように二つのゲノム範囲を定義します。
- 1番染色体(略称”chr1”)の位置10から25の範囲
- 2番染色体(略称”chr2”)の位置20から35の範囲
それを行うために、GRanges()コンストラクタ関数を使用します。
シーケンス名をseqnames=引数に、開始位置と終了位置の両方をranges=引数に、IRangesオブジェクトとして提供します。
R
demo_granges <- GRanges(
seqnames = c("chr1", "chr2"),
ranges = IRanges(
start = c(10, 20),
end = c(25, 35))
)
demo_granges
OUTPUT
GRanges object with 2 ranges and 0 metadata columns:
seqnames ranges strand
<Rle> <IRanges> <Rle>
[1] chr1 10-25 *
[2] chr2 20-35 *
-------
seqinfo: 2 sequences from an unspecified genome; no seqlengthsコンソールで、オブジェクトはseqnamesコンポーネントにシーケンス名を表示し、rangesコンポーネントにstart-end形式で範囲を表示します。
さらに、上記の例は、GRangesオブジェクトがstrandというコンポーネントを持ち、シンボル*がストランドのないゲノム範囲を示すことも示しています。これは、その情報が提供されていないためです。
ストランド情報は、strand=引数に提供できます。例えば:
R
demo_granges2 <- GRanges(
seqnames = c("chr1", "chr2"),
ranges = IRanges(
start = c(10, 20),
end = c(25, 35)),
strand = c("+", "-")
)
demo_granges2
OUTPUT
GRanges object with 2 ranges and 0 metadata columns:
seqnames ranges strand
<Rle> <IRanges> <Rle>
[1] chr1 10-25 +
[2] chr2 20-35 -
-------
seqinfo: 2 sequences from an unspecified genome; no seqlengths最後に、上記の例は、GRangesオブジェクトにseqinfoというコンポーネントが含まれていることを示しています。これは、seqnamesコンポーネントに含まれる各シーケンスに関する情報を保存するために使用されます。
前の例では、シーケンスについての情報を提供していません。
そのため、seqinfoコンポーネントはオブジェクトを作成するために使用したシーケンスの名前で自動的に埋められ、残りの情報は指定されていないままとなっています。
R
seqinfo(demo_granges2)
OUTPUT
Seqinfo object with 2 sequences from an unspecified genome; no seqlengths:
seqnames seqlengths isCircular genome
chr1 NA NA <NA>
chr2 NA NA <NA>上記の例は、シーケンスに関する情報に、それぞれの名前と長さだけでなく、それらが環状ポリマー(例:プラスミド)を表すかどうか、またそれらが属するゲノムの名前が含まれていることも示しています。
この情報は、オブジェクトの作成時にコンストラクタに直接提供したり、既存のオブジェクトに対してseqinfo()アクセサとSeqinfo()コンストラクタを使用して編集することができます。
R
seqinfo(demo_granges2) <- Seqinfo(
seqnames = c("chr1", "chr2"),
seqlengths = c(1234, 5678),
isCircular = c(FALSE, TRUE),
genome = c("homo_sapiens", "homo_sapiens")
)
demo_granges2
OUTPUT
GRanges object with 2 ranges and 0 metadata columns:
seqnames ranges strand
<Rle> <IRanges> <Rle>
[1] chr1 10-25 +
[2] chr2 20-35 -
-------
seqinfo: 2 sequences (1 circular) from homo_sapiens genome各範囲の個々の開始および終了位置、ならびにすべての区間の幅は、start(),
end()およびwidth()関数を使用して数値ベクターとして抽出できます。
R
start(demo_granges2)
OUTPUT
[1] 10 20R
end(demo_granges2)
OUTPUT
[1] 25 35R
width(demo_granges2)
OUTPUT
[1] 16 16シーケンス名とストランド情報は、それぞれseqnames()およびstrand()の関数を使用して抽出できます。
R
seqnames(demo_granges2)
OUTPUT
factor-Rle of length 2 with 2 runs
Lengths: 1 1
Values : chr1 chr2
Levels(2): chr1 chr2R
strand(demo_granges2)
OUTPUT
factor-Rle of length 2 with 2 runs
Lengths: 1 1
Values : + -
Levels(3): + - *GRangesのメタデータ
IRangesと同様に、メタデータは直接GRangesコンストラクタ関数に渡すことができます。
例えば:
R
demo_granges3 <- GRanges(
seqnames = c("chr1", "chr2"),
ranges = IRanges(
start = c(10, 20),
end = c(25, 35)),
metadata1 = c("control", "target"),
metadata2 = c(1, 2)
)
demo_granges3
OUTPUT
GRanges object with 2 ranges and 2 metadata columns:
seqnames ranges strand | metadata1 metadata2
<Rle> <IRanges> <Rle> | <character> <numeric>
[1] chr1 10-25 * | control 1
[2] chr2 20-35 * | target 2
-------
seqinfo: 2 sequences from an unspecified genome; no seqlengthsファイルからのゲノム範囲のインポート
頻繁に、大規模なゲノム範囲のコレクションは、手動で記述されたコードではなく、ファイルからインポートされます。 特に、既知の遺伝子機能のゲノム全体の注釈は、Ensembl FTP や UCSC Genome Data のようなウェブサイトにファイルとして配布されています。
さまざまなファイル形式が、バイオインフォマティクスのワークフローでゲノム範囲を格納するために一般的に使用されています。 例えば、BED (Browser Extensible Data) 形式は、クロマチン免疫沈降シーケンシング (ChIP-Seq) で一般的に見られますが、GTF (Gene Transfer Format, GTF2.2) は、エクソン、転写物、遺伝子などのゲノム機能を記述するための_デファクト_標準ファイル形式です。
次の例では、私たちは小さなGTFファイルからアクチンベータ (ACTB) の遺伝子モデルを一連のゲノム範囲としてインポートします。 例示のファイルは、Ensembl FTP サイトからダウンロードされた_Homo sapiens_種のGTFファイルのサブセットを表しています。 元のファイルは300万行以上と22のメタデータフィールドを持っており、その中からサブセットが抽出されてこのレッスン用の小さなファイルになりました。
特に、私たちは import() ジェネリックを使用します。
BiocIO
パッケージで定義され、rtracklayer
パッケージで実装されたメソッドを用いて、一般的なファイル拡張子を認識し、それぞれの特定のファイル形式を解析するための適切なメソッドに関連付けることができる多目的関数です。
R
library(rtracklayer)
WARNING
Warning: replacing previous import 'S4Arrays::makeNindexFromArrayViewport' by
'DelayedArray::makeNindexFromArrayViewport' when loading 'SummarizedExperiment'R
actb_gtf_data <- rtracklayer::import("data/actb.gtf")
actb_gtf_data
OUTPUT
GRanges object with 267 ranges and 7 metadata columns:
seqnames ranges strand | source type score
<Rle> <IRanges> <Rle> | <factor> <factor> <numeric>
[1] 7 5526409-5563902 - | rtracklayer gene NA
[2] 7 5526409-5530601 - | rtracklayer transcript NA
[3] 7 5530542-5530601 - | rtracklayer exon NA
[4] 7 5529535-5529684 - | rtracklayer exon NA
[5] 7 5529535-5529657 - | rtracklayer CDS NA
... ... ... ... . ... ... ...
[263] 7 5540676-5540771 - | rtracklayer five_prime_utr NA
[264] 7 5529658-5529663 - | rtracklayer five_prime_utr NA
[265] 7 5561852-5562716 - | rtracklayer transcript NA
[266] 7 5562390-5562716 - | rtracklayer exon NA
[267] 7 5561852-5561949 - | rtracklayer exon NA
phase gene_id gene_name transcript_id
<integer> <character> <character> <character>
[1] <NA> ENSG00000075624 ACTB <NA>
[2] <NA> ENSG00000075624 ACTB ENST00000674681
[3] <NA> ENSG00000075624 ACTB ENST00000674681
[4] <NA> ENSG00000075624 ACTB ENST00000674681
[5] <NA> ENSG00000075624 ACTB ENST00000674681
... ... ... ... ...
[263] <NA> ENSG00000075624 ACTB ENST00000414620
[264] <NA> ENSG00000075624 ACTB ENST00000414620
[265] <NA> ENSG00000075624 ACTB ENST00000646584
[266] <NA> ENSG00000075624 ACTB ENST00000646584
[267] <NA> ENSG00000075624 ACTB ENST00000646584
-------
seqinfo: 1 sequence from an unspecified genome; no seqlengthsさらに進む
特定のファイル形式を解析するための個別のメソッドを直接呼び出すことができます。
例えば、この場合、GTFファイル形式はGFFバージョン2ファイル形式と同一であるため、私たちは
rtracklayer::import.gff2()
関数を直接呼び出すことができます。同じ効果を得ることができます。
すべてのメソッドのフルリストは、rtracklayer パッケージのドキュメントを参照してください。
上記の例では、GTFファイルの内容がGRangesオブジェクトにインポートされました。
ファイルの各エントリについて、配列名、開始位置と終了位置、ストランド情報がオブジェクトの専用コンポーネントをポピュレートするために使用され、他のすべての情報はメタデータの別のカラムとして保存されます。
ここから、このGRangesオブジェクトは、私たちがこのエピソードの初めに作成した他のGRangesオブジェクトのように操作できます。
GRangesおよびGRangesListクラスの操作
これまでに示したように、GRangesオブジェクトは手動で定義またはファイルからインポートできます。
それらは、興味のあるゲノム領域を表し、既知のゲノム機能のデータベースをそれぞれ表します。
いずれにせよ、生物情報学のワークフロー全体でGRangesオブジェクトに一般的に適用される多数の操作があります。
サブセット
例えば、subset()メソッドは、配列名、開始位置と終了位置、ストランド、または任意のメタデータフィールドの条件に一致するゲノム範囲のセットを抽出するのに非常に便利です。
以下の例では、位置5527147で開始するtranscript型のすべてのレコードを抽出します。
R
subset(actb_gtf_data, type == "transcript" & start == 5527147)
OUTPUT
GRanges object with 5 ranges and 7 metadata columns:
seqnames ranges strand | source type score
<Rle> <IRanges> <Rle> | <factor> <factor> <numeric>
[1] 7 5527147-5529949 - | rtracklayer transcript NA
[2] 7 5527147-5530581 - | rtracklayer transcript NA
[3] 7 5527147-5530604 - | rtracklayer transcript NA
[4] 7 5527147-5530604 - | rtracklayer transcript NA
[5] 7 5527147-5530604 - | rtracklayer transcript NA
phase gene_id gene_name transcript_id
<integer> <character> <character> <character>
[1] <NA> ENSG00000075624 ACTB ENST00000642480
[2] <NA> ENSG00000075624 ACTB ENST00000676397
[3] <NA> ENSG00000075624 ACTB ENST00000676319
[4] <NA> ENSG00000075624 ACTB ENST00000676189
[5] <NA> ENSG00000075624 ACTB ENST00000473257
-------
seqinfo: 1 sequence from an unspecified genome; no seqlengths分割
別々に、split()メソッドは、最初に単一のGRangesオブジェクトに格納されていたゲノム範囲のセットを、名前付きのGRangesオブジェクトのリストに分割するのに役立ちます。
便利なことに、GRangesListクラスは、GRangesオブジェクトのリストを効率的に表示および処理するためのコンテナを提供します。
以下の例では、最初にエクソンを表すエントリのサブセットを抽出し、次にそれらのエクソンを転写物識別子で分割し、結果をGRangesListオブジェクトとして得ます。
R
actb_exons <- subset(actb_gtf_data, type == "exon")
actb_exons_by_transcript <- split(actb_exons, actb_exons$transcript_id)
actb_exons_by_transcript
OUTPUT
GRangesList object of length 23:
$ENST00000414620
GRanges object with 4 ranges and 7 metadata columns:
seqnames ranges strand | source type score
<Rle> <IRanges> <Rle> | <factor> <factor> <numeric>
[1] 7 5562574-5562790 - | rtracklayer exon NA
[2] 7 5540676-5540771 - | rtracklayer exon NA
[3] 7 5529535-5529663 - | rtracklayer exon NA
[4] 7 5529282-5529400 - | rtracklayer exon NA
phase gene_id gene_name transcript_id
<integer> <character> <character> <character>
[1] <NA> ENSG00000075624 ACTB ENST00000414620
[2] <NA> ENSG00000075624 ACTB ENST00000414620
[3] <NA> ENSG00000075624 ACTB ENST00000414620
[4] <NA> ENSG00000075624 ACTB ENST00000414620
-------
seqinfo: 1 sequence from an unspecified genome; no seqlengths
$ENST00000417101
GRanges object with 3 ranges and 7 metadata columns:
seqnames ranges strand | source type score
<Rle> <IRanges> <Rle> | <factor> <factor> <numeric>
[1] 7 5529806-5529982 - | rtracklayer exon NA
[2] 7 5529535-5529663 - | rtracklayer exon NA
[3] 7 5529235-5529400 - | rtracklayer exon NA
phase gene_id gene_name transcript_id
<integer> <character> <character> <character>
[1] <NA> ENSG00000075624 ACTB ENST00000417101
[2] <NA> ENSG00000075624 ACTB ENST00000417101
[3] <NA> ENSG00000075624 ACTB ENST00000417101
-------
seqinfo: 1 sequence from an unspecified genome; no seqlengths
$ENST00000425660
GRanges object with 7 ranges and 7 metadata columns:
seqnames ranges strand | source type score
<Rle> <IRanges> <Rle> | <factor> <factor> <numeric>
[1] 7 5530524-5530601 - | rtracklayer exon NA
[2] 7 5529535-5529663 - | rtracklayer exon NA
[3] 7 5529161-5529400 - | rtracklayer exon NA
[4] 7 5529019-5529059 - | rtracklayer exon NA
[5] 7 5528281-5528719 - | rtracklayer exon NA
[6] 7 5528004-5528185 - | rtracklayer exon NA
[7] 7 5527156-5527891 - | rtracklayer exon NA
phase gene_id gene_name transcript_id
<integer> <character> <character> <character>
[1] <NA> ENSG00000075624 ACTB ENST00000425660
[2] <NA> ENSG00000075624 ACTB ENST00000425660
[3] <NA> ENSG00000075624 ACTB ENST00000425660
[4] <NA> ENSG00000075624 ACTB ENST00000425660
[5] <NA> ENSG00000075624 ACTB ENST00000425660
[6] <NA> ENSG00000075624 ACTB ENST00000425660
[7] <NA> ENSG00000075624 ACTB ENST00000425660
-------
seqinfo: 1 sequence from an unspecified genome; no seqlengths
...
<20 more elements>コンソールで上記のオブジェクトを印刷すると、最初の行はそのオブジェクトのクラスをGRrangesListとして確認し、そのリスト内の各名前付きGRangesがドル記号とそのアイテムの名前によって紹介され、通常のRの名前付きリストのように表示されます。
長さ
性質上、listオブジェクトに適用可能な多くのメソッドは、直接GRangesListオブジェクトに適用できます。
例えば、lengths()関数をGRangesListに使用して、リスト内の各GRangesオブジェクトの長さを整数ベクトルとして表示できます。
上記の最新の例では、各転写物内のエクソンの数を、GRangesList内の各GRangesオブジェクトの長さとして計算できます:
R
lengths(actb_exons_by_transcript)
OUTPUT
ENST00000414620 ENST00000417101 ENST00000425660 ENST00000432588 ENST00000443528
4 3 7 5 3
ENST00000462494 ENST00000464611 ENST00000473257 ENST00000477812 ENST00000480301
5 3 5 5 2
ENST00000484841 ENST00000493945 ENST00000642480 ENST00000645025 ENST00000645576
5 6 5 4 5
ENST00000646584 ENST00000646664 ENST00000647275 ENST00000674681 ENST00000675515
2 6 3 6 6
ENST00000676189 ENST00000676319 ENST00000676397
6 3 6 チャレンジ
重要なことに、上記の lengths() 関数 (最終的に
s が付いています) は、length() 関数 (
s が付いていないもの) とは異なります。
前者はリストオブジェクトに対して使用されることを目的としており、リスト内の各要素の長さを返すベクトルを返します。後者はリスト自体の長さを表す単一の数値スカラーを返します
(つまり、リスト内の要素数)。
length(actb_exons_by_transcript)
は何を返し、この数は生物学的に何を表しますか?
R
length(actb_exons_by_transcript)
OUTPUT
[1] 23このコードは、単一の整数値 23
を返します。それは、GRangesListオブジェクト内のGRangesの数であり、遺伝子ACTBの転写物の数でもあります。
オーバーラップによるサブセット
ゲノム範囲で作業するときの最も一般的な操作の一つは、特定のゲノム領域に位置するゲノム範囲の任意の大きなコレクションをサブセット化することです。たとえば、ゲノムブラウザに情報をトラックとして視覚化するときなどです。
示すために、私たちは興味のある領域を表す新しいGRangesを手動で定義し、その領域に重なる以前にインポートされたすべてのゲノム範囲をGTFファイルから抽出するために使用します。
R
region_of_interest <- GRanges(
seqnames = "7",
ranges = IRanges(start = 5525830, end = 5531239)
)
actb_in_region <- subsetByOverlaps(x = actb_gtf_data, ranges = region_of_interest)
actb_in_region
OUTPUT
GRanges object with 256 ranges and 7 metadata columns:
seqnames ranges strand | source type score
<Rle> <IRanges> <Rle> | <factor> <factor> <numeric>
[1] 7 5526409-5563902 - | rtracklayer gene NA
[2] 7 5526409-5530601 - | rtracklayer transcript NA
[3] 7 5530542-5530601 - | rtracklayer exon NA
[4] 7 5529535-5529684 - | rtracklayer exon NA
[5] 7 5529535-5529657 - | rtracklayer CDS NA
... ... ... ... . ... ... ...
[252] 7 5529535-5529657 - | rtracklayer CDS NA
[253] 7 5529655-5529657 - | rtracklayer start_codon NA
[254] 7 5529282-5529400 - | rtracklayer exon NA
[255] 7 5529282-5529400 - | rtracklayer CDS NA
[256] 7 5529658-5529663 - | rtracklayer five_prime_utr NA
phase gene_id gene_name transcript_id
<integer> <character> <character> <character>
[1] <NA> ENSG00000075624 ACTB <NA>
[2] <NA> ENSG00000075624 ACTB ENST00000674681
[3] <NA> ENSG00000075624 ACTB ENST00000674681
[4] <NA> ENSG00000075624 ACTB ENST00000674681
[5] <NA> ENSG00000075624 ACTB ENST00000674681
... ... ... ... ...
[252] <NA> ENSG00000075624 ACTB ENST00000414620
[253] <NA> ENSG00000075624 ACTB ENST00000414620
[254] <NA> ENSG00000075624 ACTB ENST00000414620
[255] <NA> ENSG00000075624 ACTB ENST00000414620
[256] <NA> ENSG00000075624 ACTB ENST00000414620
-------
seqinfo: 1 sequence from an unspecified genome; no seqlengthssubset()メソッドと同様に、subsetByOverlaps()メソッドは新しいGRangesオブジェクトを返します。
私たちは、新しいサブセットオブジェクト内の情報(新しいサブセットされたオブジェクト内に256の範囲があり、元のオブジェクトには267の範囲がある)を視覚的に比較することができ、あるいは新しいGRangesオブジェクトが元のGRangesオブジェクトよりも小さいかどうかを確認するために、プログラム的に2つのオブジェクトの長さを比較することもできます。
R
length(actb_in_region) - length(actb_gtf_data)
OUTPUT
[1] -11上記の例では、新しいGRangesオブジェクトは、元のGRangesオブジェクトよりも11レコード少ないことがわかります。
さらに進む
GRangesおよびGRangesListオブジェクトで動作するための方法が他にもたくさんありますが、ここで示すことができるのはそれだけです。
GenomicRangesパッケージで定義された関数の完全なリストは、パッケージのドキュメントのインデックスページで見つけることができ、help(package="GenomicRanges")を使用してアクセスできます。
GenomicRangesパッケージのビネットには、より多くの例やユースケースも見つけることができ、browseVignettes("GenomicRanges")を使用してアクセスできます。
-
GenomicRangesパッケージは、ゲノムスケールでの座標の範囲を表すクラスを定義します。 -
GenomicRangesパッケージは、ゲノム範囲を効率よく処理するためのメソッドも定義します。 -
rtracklayerパッケージは、一般的なゲノムファイル形式からゲノム範囲をインポートおよびエクスポートするための関数を提供します。
Content from アノテーションの取り扱い
Last updated on 2025-09-23 | Edit this page
Overview
Questions
- どのバイオコンダクターパッケージが、遺伝子アノテーションを効率的に取得および使用するためのメソッドを提供していますか?
- 異なる遺伝子識別子間を変換するために、遺伝子アノテーションパッケージをどのように使用できますか?
Objectives
- バイオコンダクタープロジェクトでの遺伝子アノテーションの管理方法を説明してください。
- 遺伝子アノテーションを取得および使用するためのバイオコンダクターパッケージとメソッドを特定します。
パッケージのインストール
次のセクションに進む前に、必要なバイオコンダクターパッケージをインストールします。
まず、BiocManagerパッケージがインストールされていることを確認します。そうでない場合は、それをインストールします。
次に、BiocManager::install()関数を使用して必要なパッケージをインストールします。
R
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install(c("biomaRt", "org.Hs.eg.db"))
概要
遺伝子アノテーションをクエリするために特化したパッケージは、それぞれの特性に基づいてバイオコンダクターの「ソフトウェア」と「アノテーション」カテゴリに存在します。
「ソフトウェア」セクションでは、実際に遺伝子アノテーションを含まないパッケージを見つけますが、それらはオンラインリソースから動的に取得します(例えば、Ensembl BioMart)。 そのようなバイオコンダクターパッケージの一つがbiomaRtです。
その代わりに、「アノテーション」セクションでは、アノテーションを含むパッケージを見つけます。 例として、org.Hs.eg.db、EnsDb.Hsapiens.v86、およびTxDb.Hsapiens.UCSC.hg38.knownGeneがあります。
このエピソードでは、2つのアプローチを示します:
- biomaRtパッケージを使用してEnsembl BioMart APIからアノテーションをクエリします。
- org.Hs.eg.dbアノテーションパッケージからアノテーションをクエリします。
オンラインリソースまたはバイオコンダクタアノテーションパッケージ?
最新情報へのアクセス
バイオコンダクターの6ヶ月のリリースサイクルは、最新の安定版リリースブランチから提供されるパッケージが6ヶ月間更新されないことを示唆しています(バグ修正のみが許可され、機能の更新はありません)。 その結果、アノテーションパッケージは最大で6ヶ月間の古い情報を含む可能性があります。
その代わりに、独立したオンラインリソースは、更新情報のリリースを促進する異なるポリシーを持ちます。 一部のデータベースは頻繁に更新されますが、他のデータベースは数年にわたって更新されていない場合があります。
最新の情報へのアクセスは、再現性とのバランスも考慮しなければなりません。 最新の情報を1回ダウンロードしただけでは、いつその情報がダウンロードされたかを記録していなければ意味がありません。
ストレージ要件
本質的に、バイオコンダクターアノテーションパッケージはソフトウェアパッケージよりも大きいです。 他のRパッケージと同様に、アノテーションパッケージは使用する前にユーザーのコンピューターにインストールする必要があります。 これにより、無視できない量のディスクスペースを使用する可能性があります。
逆に、オンラインリソースは一般にプログラムでアクセスされ、通常、ユーザーが再現可能な分析を行うためにコードを記録するだけで済みます。
インターネット接続
オンラインリソースを使用しているときは、オンラインリソースからダウンロードしたアノテーションをローカルファイルに書き込み、分析中にそのローカルファイルを参照するのが良いアイデアです。
オンラインリソースが何らかの理由で使用できなくなる(例えば、ダウンタイムやインターネット接続の喪失)と、ローカルファイルを使用する分析は続行できますが、オンラインリソースに依存する分析はできません。
対照的に、バイオコンダクターアノテーションパッケージは、インストール時にのみインターネット接続が必要です。 一度インストールされると、ローカルに保存された情報に依存するため、インターネット接続は必要ありません。
再現性
バイオコンダクターアノテーションパッケージは自然にバージョン管理されているため、ユーザーは分析で使用したパッケージのバージョンを自信を持って報告できます。 ソフトウェアパッケージと同様に、ユーザーはアノテーションパッケージをいつ、どのように更新するかを制御します。
オンラインリソースは再現可能な分析を促進するための異なるポリシーを持っています。 オンラインリソースの一部はアノテーションの過去のバージョンを保持しており、ユーザーは時間の経過とともに同じ情報に一貫してアクセスできます。 これがない場合、一つの時点でアノテーションのコピーをダウンロードし、そのコピーをプロジェクトの存続期間中保持する必要があるかもしれません。一貫したアノテーションセットを使用することを保証するために。
Ensembl BioMartからアノテーションをクエリする
Ensembl BioMart
Ensembl BioMartは、生物学的データの広範な配列へのアクセスを容易にするために設計された堅牢なデータマイニングツールです。
BioMart web interfaceは、研究者が複数の種にわたって遺伝子、タンパク質、およびその他のゲノムの特徴に関するデータを効率的にクエリし、取得できるようにします。 ユーザーに対して遺伝子ID、染色体位置、機能的アノテーションなどのさまざまな属性に基づいてデータをフィルタリング、並べ替え、エクスポートすることを許可します。
バイオコンダクターbiomaRtパッケージ
biomaRtは、Rセッションから直接Ensembl BioMartテーブルから大量のデータを取得できるバイオコンダクターパッケージです。
まずパッケージを読み込みます:
R
library(biomaRt)
利用可能なマートのリスト
Ensembl BioMartは、それぞれ「マート」または「バイオマート」とも呼ばれる4つのデータベースに多様な生物学的信息を整理します。 各マートは異なるタイプのデータに焦点を合わせています。
ユーザーは、関心のあるデータタイプに対応するマートを選択する必要があります。その後、情報をクエリできます。
関数listMarts()は、これらのマートの名前を表示するために使用できます。
これによりユーザーはマートの名前を記憶する必要がなくなり、また、関数はマートが名前変更、追加、または削除される場合にも更新された名前リストを返します。
R
listMarts()
OUTPUT
biomart version
1 ENSEMBL_MART_ENSEMBL Ensembl Genes 115
2 ENSEMBL_MART_MOUSE Mouse strains 115
3 ENSEMBL_MART_SNP Ensembl Variation 115
4 ENSEMBL_MART_FUNCGEN Ensembl Regulation 115このデモでは、Ensembl遺伝子セットを含むENSEMBL_MART_ENSEMBLというバイオマートを使用します。
特に、versionカラムはバイオマートのバージョンも示しています。
Ensembl BioMartは定期的に更新されます(年に複数回)。
デフォルトでは、biomaRt関数は各バイオマートの最新バージョンにアクセスします。
これは再現性には理想的ではありません。
幸いなことに、Ensembl BioMartは、過去のバージョンのアーカイブを行い、プログラムからでも、ウェブサイトからでもアクセスできます。
関数listEnsemblArchives()は、アクセス可能なすべてのEnsembl
BioMartのバージョンを表示するために使用できます。
R
listEnsemblArchives()
OUTPUT
name date url version
1 Ensembl GRCh37 Feb 2014 https://grch37.ensembl.org GRCh37
2 Ensembl 115 Sep 2025 https://sep2025.archive.ensembl.org 115
3 Ensembl 114 May 2025 https://may2025.archive.ensembl.org 114
4 Ensembl 113 Oct 2024 https://oct2024.archive.ensembl.org 113
5 Ensembl 112 May 2024 https://may2024.archive.ensembl.org 112
6 Ensembl 111 Jan 2024 https://jan2024.archive.ensembl.org 111
7 Ensembl 110 Jul 2023 https://jul2023.archive.ensembl.org 110
8 Ensembl 109 Feb 2023 https://feb2023.archive.ensembl.org 109
9 Ensembl 108 Oct 2022 https://oct2022.archive.ensembl.org 108
10 Ensembl 107 Jul 2022 https://jul2022.archive.ensembl.org 107
11 Ensembl 106 Apr 2022 https://apr2022.archive.ensembl.org 106
12 Ensembl 105 Dec 2021 https://dec2021.archive.ensembl.org 105
13 Ensembl 104 May 2021 https://may2021.archive.ensembl.org 104
14 Ensembl 103 Feb 2021 https://feb2021.archive.ensembl.org 103
15 Ensembl 102 Nov 2020 https://nov2020.archive.ensembl.org 102
16 Ensembl 101 Aug 2020 https://aug2020.archive.ensembl.org 101
17 Ensembl 100 Apr 2020 https://apr2020.archive.ensembl.org 100
18 Ensembl 80 May 2015 https://may2015.archive.ensembl.org 80
19 Ensembl 77 Oct 2014 https://oct2014.archive.ensembl.org 77
20 Ensembl 75 Feb 2014 https://feb2014.archive.ensembl.org 75
21 Ensembl 54 May 2009 https://may2009.archive.ensembl.org 54
current_release
1
2 *
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21 上記の出力での重要な情報は、urlカラムです。これは、biomaRt関数が対応するEnsembl
BioMartのスナップショットからデータにアクセスするために必要なURLを提供します。
執筆時点では、現在のリリースはEnsembl
112であるため、対応するurlhttps://may2024.archive.ensembl.orgを使用して、いつこのレッスンが配信されても再現可能な結果を確保します。
バイオマートへの接続
上記で収集した2つの情報、すなわちバイオマートの名前とスナップショットのURLが、バイオマートデータベースに再現可能に接続するために必要なすべてです。
関数useMart()を使用して接続を作成します。
接続は伝統的にmartというオブジェクトに保存され、次のステップでオンラインマートから情報をクエリするために再利用されます。
R
mart <- useMart(biomart = "ENSEMBL_MART_ENSEMBL", host = "https://may2024.archive.ensembl.org")
利用可能なデータセットのリスト
各バイオマートには複数のデータセットが含まれています。
関数listDatasets()は、これらのデータセットの情報を表示するために使用されます。
これにより、ユーザーはデータセットの名前を記憶する必要がなくなり、関数が返す情報には各データセットの簡単な説明とそのバージョンが含まれます。
以下の例では、出力テーブルを最初の数行に制限します。完全なテーブルは214行です。
R
head(listDatasets(mart))
OUTPUT
dataset description
1 abrachyrhynchus_gene_ensembl Pink-footed goose genes (ASM259213v1)
2 acalliptera_gene_ensembl Eastern happy genes (fAstCal1.3)
3 acarolinensis_gene_ensembl Green anole genes (AnoCar2.0v2)
4 acchrysaetos_gene_ensembl Golden eagle genes (bAquChr1.2)
5 acitrinellus_gene_ensembl Midas cichlid genes (Midas_v5)
6 amelanoleuca_gene_ensembl Giant panda genes (ASM200744v2)
version
1 ASM259213v1
2 fAstCal1.3
3 AnoCar2.0v2
4 bAquChr1.2
5 Midas_v5
6 ASM200744v2上記の出力での重要な情報は、datasetカラムです。これは、biomaRt関数が対応するバイオマートテーブルからデータにアクセスするために必要な識別子を提供します。
このデモでは、出力に表示されていないヒトのEnsembl遺伝子セットを使用します。
利用可能なデータセットの数を考えると、手動でテーブルを検索するのではなく、パターンマッチングを使用して情報のテーブルをプログラムでフィルタリングしてみましょう:
R
subset(listDatasets(mart), grepl("sapiens", dataset))
OUTPUT
dataset description version
80 hsapiens_gene_ensembl Human genes (GRCh38.p14) GRCh38.p14上記の出力から、欲しいデータセットの識別子がhsapiens_gene_ensemblであることが分かります。
データセットへの接続
使用したいデータセットを選択したら、再度関数useMart()を呼び出し、選択したデータセットを指定する必要があります。
一般的には、以前のuseMart()の呼び出しをコピーして必要に応じて編集することが多いです。
また、通常、martオブジェクトを新しい接続で置き換えることが一般的な慣行です。
R
mart <- useMart(
biomart = "ENSEMBL_MART_ENSEMBL",
dataset = "hsapiens_gene_ensembl",
host = "https://may2024.archive.ensembl.org")
データセット内の情報をリスト表示する
BioMartテーブルには、属性としても知られる多くの情報が含まれています。 実際には、それらは「ページ」として知られるカテゴリにグループ化されています。
関数listAttributes()は、それらの属性に関する情報を表示するために使用できます。
これにより、ユーザーはパッケージ内の利用可能な属性の名前を記憶する必要がなくなり、関数が返す情報には各属性の簡単な説明とそのページ分類が含まれます。
以下の例では、出力テーブルを最初の数行に制限します。完全なテーブルは3157行です。
R
head(listAttributes(mart))
OUTPUT
name description page
1 ensembl_gene_id Gene stable ID feature_page
2 ensembl_gene_id_version Gene stable ID version feature_page
3 ensembl_transcript_id Transcript stable ID feature_page
4 ensembl_transcript_id_version Transcript stable ID version feature_page
5 ensembl_peptide_id Protein stable ID feature_page
6 ensembl_peptide_id_version Protein stable ID version feature_page上記の出力での重要な情報は、nameカラムです。これは、biomaRt関数が対応するバイオマートデータセットからその情報をクエリするために必要な識別子を提供します。
クエリしたい属性の選択は、達成したいことによって決まります。
例えば、遺伝子識別子のセットがあり、それに対してクエリしたいことを想像しましょう:
- 遺伝子符号
- 遺伝子が位置する染色体の名前
- その染色体上の遺伝子の開始位置と終了位置
- 遺伝子がコーディングされているストランド
ユーザーは通常、含めたい属性を特定するために、属性テーブルを手動で調べます。 また、経験と直感に基づいて属性テーブルをプログラム的にフィルタリングして、検索を絞り込むことも可能です:
R
subset(listAttributes(mart), grepl("position", name) & grepl("feature", page))
OUTPUT
name description page
10 start_position Gene start (bp) feature_page
11 end_position Gene end (bp) feature_pageBioMartテーブルから情報をクエリする
これで、実際のクエリを実行するために必要な情報がすべて揃いました:
- BioMartデータセットへの接続
- そのデータセット内の利用可能な属性のリスト
関数getBM()は、主なbiomaRtクエリ関数です。
フィルターとそれに対応する値のセットを指定すると、接続されているBioMartデータセットからユーザーが要求した属性を取得します。
以下の例では、クエリ用の任意の遺伝子識別子のベクトルを手動で作成します。 実際には、クエリは先行する分析から始まることがよくあります(例えば、差次的な遺伝子発現)。
以下の例では、まだ紹介していない属性もクエリされます。
前のセクションで、listAttributes()から返される属性テーブルを検索して、クエリに含める属性を特定する方法を説明しました。
R
query_gene_ids <- c(
"ENSG00000133101",
"ENSG00000145386",
"ENSG00000134057",
"ENSG00000157456",
"ENSG00000147082"
)
getBM(
attributes = c(
"ensembl_gene_id",
"hgnc_symbol",
"chromosome_name",
"start_position",
"end_position",
"strand"
),
filters = "ensembl_gene_id",
values = query_gene_ids,
mart = mart
)
OUTPUT
ensembl_gene_id hgnc_symbol chromosome_name start_position end_position
1 ENSG00000133101 CCNA1 13 36431520 36442870
2 ENSG00000134057 CCNB1 5 69167135 69178245
3 ENSG00000145386 CCNA2 4 121816444 121823883
4 ENSG00000147082 CCNB3 X 50202713 50351914
5 ENSG00000157456 CCNB2 15 59105126 59125045
strand
1 1
2 1
3 -1
4 1
5 1フィルタリング属性ensembl_gene_idをデータセットから取得した属性に含めたことに注意してください。
これは、新しく取得された属性をクエリで使用される属性と確実に一致させるための鍵です。
アノテーションパッケージからアノテーションをクエリする
アノテーションパッケージのファミリー
包括的な情報を必要としつつ、適切なパッケージサイズを維持するために、バイオコンダクターアノテーションパッケージはリリース、データタイプ、種によって編成されています。
バイオコンダクターアノテーションパッケージの主要なファミリーは以下の通りです:
-
OrgDbパッケージは、さまざまなタイプの遺伝子識別子と経路情報の間のマッピングを提供します。 -
EnsDbパッケージは、Ensemblアノテーションの個々のリリースを提供します。 -
TxDbパッケージは、UCSCアノテーションの個々のリリースを提供します。
これらのアノテーションファミリーは、AnnotationDbiパッケージで定義されたAnnotationDb基底クラスから派生しています。
その結果、これらのアノテーションパッケージは、以下のセクションで示される同じ一連のR関数を使用してアクセスします。
OrgDbパッケージの使用
この例では、org.Hs.eg.dbパッケージを使用して、ヒト種の遺伝子アノテーションの使用方法を示します。
最初にパッケージを読み込みましょう:
R
library(org.Hs.eg.db)
各OrgDbパッケージには、パッケージ自体と同名のオブジェクトが含まれています。
そのオブジェクトには、パッケージが提供することを目的とするアノテーションが含まれています。
情報をクエリすることに加えて、オブジェクト全体を呼び出すことができ、含まれるアノテーションのスナップショットが作成された日付などの情報を印刷します。
R
org.Hs.eg.db
OUTPUT
OrgDb object:
| DBSCHEMAVERSION: 2.1
| Db type: OrgDb
| Supporting package: AnnotationDbi
| DBSCHEMA: HUMAN_DB
| ORGANISM: Homo sapiens
| SPECIES: Human
| EGSOURCEDATE: 2024-Mar12
| EGSOURCENAME: Entrez Gene
| EGSOURCEURL: ftp://ftp.ncbi.nlm.nih.gov/gene/DATA
| CENTRALID: EG
| TAXID: 9606
| GOSOURCENAME: Gene Ontology
| GOSOURCEURL: http://current.geneontology.org/ontology/go-basic.obo
| GOSOURCEDATE: 2024-01-17
| GOEGSOURCEDATE: 2024-Mar12
| GOEGSOURCENAME: Entrez Gene
| GOEGSOURCEURL: ftp://ftp.ncbi.nlm.nih.gov/gene/DATA
| KEGGSOURCENAME: KEGG GENOME
| KEGGSOURCEURL: ftp://ftp.genome.jp/pub/kegg/genomes
| KEGGSOURCEDATE: 2011-Mar15
| GPSOURCENAME: UCSC Genome Bioinformatics (Homo sapiens)
| GPSOURCEURL:
| GPSOURCEDATE: 2024-Feb29
| ENSOURCEDATE: 2023-Nov22
| ENSOURCENAME: Ensembl
| ENSOURCEURL: ftp://ftp.ensembl.org/pub/current_fasta
| UPSOURCENAME: Uniprot
| UPSOURCEURL: http://www.UniProt.org/
| UPSOURCEDATE: Thu Apr 18 21:39:39 2024OUTPUT
Please see: help('select') for usage informationそのオブジェクトは、クエリを実行し、アノテーションを取得するためのAnnotationDbi関数に供給する必要があるものです。
アノテーションパッケージ内の利用可能な情報をリスト表示する
関数columns()を使用してオブジェクト内で利用可能なアノテーションを表示できます。
ここで「カラム」という言葉は、データベース内の情報を格納するために使用されるテーブルのカラムを指し、BioMartの「属性」と同じ概念です。 言い換えれば、カラムはオブジェクトから取得可能なすべてのアノテーションのタイプを表します。
これにより、ユーザーはパッケージ内の利用可能なアノテーションのカラムの名前を記憶する必要がなくなります。
R
columns(org.Hs.eg.db)
OUTPUT
[1] "ACCNUM" "ALIAS" "ENSEMBL" "ENSEMBLPROT" "ENSEMBLTRANS"
[6] "ENTREZID" "ENZYME" "EVIDENCE" "EVIDENCEALL" "GENENAME"
[11] "GENETYPE" "GO" "GOALL" "IPI" "MAP"
[16] "OMIM" "ONTOLOGY" "ONTOLOGYALL" "PATH" "PFAM"
[21] "PMID" "PROSITE" "REFSEQ" "SYMBOL" "UCSCKG"
[26] "UNIPROT" キーとキータイプのリスト
データベース用語で言う_キー_は、データベーステーブルから情報をクエリするための値です。
カラムに整理されている情報のため、_キータイプ_はキー値が保存されているカラムの名前です。
データベーステーブル内のカラムの可変数を考慮すると、一部のテーブルでは複数のキーで情報をクエリできる場合があります。 その結果、クエリの一部としてキーとキーの型の両方を指定することが重要です。
関数 keytypes()
を使用すると、オブジェクトから情報をクエリするために使用できる列の名前を表示できます。
R
keytypes(org.Hs.eg.db)
OUTPUT
[1] "ACCNUM" "ALIAS" "ENSEMBL" "ENSEMBLPROT" "ENSEMBLTRANS"
[6] "ENTREZID" "ENZYME" "EVIDENCE" "EVIDENCEALL" "GENENAME"
[11] "GENETYPE" "GO" "GOALL" "IPI" "MAP"
[16] "OMIM" "ONTOLOGY" "ONTOLOGYALL" "PATH" "PFAM"
[21] "PMID" "PROSITE" "REFSEQ" "SYMBOL" "UCSCKG"
[26] "UNIPROT" 関数 keys()
を使用すると、特定のキータイプのすべての可能な値を表示できます。
一般的には、クエリされるキーの型を指定する方が良い慣行です(あいまいさを避けるため)。ただし、データベーステーブルには、ユーザーが自分で型を指定しない場合に使用される「主キー」が通常あります。
以下の例では、遺伝子記号のキーのリストを最初のいくつかの値に制限します。 完全なセットは 193279 値を含みます。
R
head(keys(org.Hs.eg.db, keytype = "SYMBOL"))
OUTPUT
[1] "A1BG" "A2M" "A2MP1" "NAT1" "NAT2" "NATP" アノテーションパッケージから情報をクエリする
関数 select() は、主な AnnotationDbi
クエリ関数です。 AnnotationDb
オブジェクト、キー値、および列(および必要に応じて、主キーでない場合は指定されたキーの型)を考えると、アノテーションオブジェクトからユーザーが要求した列を取得します。
以下の例では、いくつかのセクション上の BioMart 例で使用された任意の遺伝子識別子のベクターを再利用します。
columns()
関数の出力からわかるように、アノテーションオブジェクトには、BioMart
例でクエリした一部の属性が含まれていません。
この場合、クエリしてみましょう:
- 遺伝子記号
- 遺伝子名
- 遺伝子タイプ
R
select(
x = org.Hs.eg.db,
keys = query_gene_ids,
columns = c(
"SYMBOL",
"GENENAME",
"GENETYPE"
),
keytype = "ENSEMBL"
)
OUTPUT
'select()' returned 1:1 mapping between keys and columnsOUTPUT
ENSEMBL SYMBOL GENENAME GENETYPE
1 ENSG00000133101 CCNA1 cyclin A1 protein-coding
2 ENSG00000145386 CCNA2 cyclin A2 protein-coding
3 ENSG00000134057 CCNB1 cyclin B1 protein-coding
4 ENSG00000157456 CCNB2 cyclin B2 protein-coding
5 ENSG00000147082 CCNB3 cyclin B3 protein-codingbiomaRt
との小さな違いは、select()
の出力は、クエリで使用されたキータイプに対応する列を自動的に含むことです。
言い換えれば、取得する列にキータイプを再度指定する必要はありません。
ベクトル化された 1:1 マッピング
アノテーションが 1 対 多の関係を表示することがある場合があります。 例えば、個々の遺伝子には通常、ユニークな Ensembl 遺伝子識別子があり、複数の遺伝子名別名の下で知られることがあります。
前のセクションで示した select()
関数は、指定されたキーに対して要求された列の すべて
の値を自動的に返します。
これは、アノテーションが返される表形式であるため可能です。
行が追加され、関連するすべての値と同じ行に表示するために、必要に応じて値を繰り返します。
場合によっては、その動作は望ましくないことがあります。 その代わりに、ユーザーは入力した各キーに対して単一の値を取得したいと考える場合があります。 一般的なシナリオは、差次的遺伝子発現 (DGE) の際に発生し、遺伝子識別子が分析全体で遺伝子を一意に識別するために使用され、遺伝子記号が DGE 統計の最終表に追加され、読みやすい人間に優しい遺伝子識別子を提供します。 ただし、DGE 統計の行を複製することは望ましくないため、各遺伝子に注釈を付けるために単一の遺伝子記号が必要です。
この目的には mapIds() 関数を使用できます。 関数
mapIds() と select()
の主な違いは、それぞれの引数 column(単数)と
columns(複数)です。 関数 mapIds()
は単一の列名を受け入れ、名前付けされた文字ベクターを返します。ここで名前は入力クエリ値であり、値は要求された列内の対応する値です。
1 対 多の関係を処理するために、関数 mapIds() には
multiVals
という引数があり、関数が複数の値を処理する方法を指定できます。
デフォルトは最初の値を取得し、他の値は無視することです。
以下の例では、一連の Ensembl 遺伝子識別子の遺伝子記号をクエリします。
R
mapIds(
x = org.Hs.eg.db,
keys = query_gene_ids,
column = "SYMBOL",
keytype = "ENSEMBL"
)
OUTPUT
'select()' returned 1:1 mapping between keys and columnsOUTPUT
ENSG00000133101 ENSG00000145386 ENSG00000134057 ENSG00000157456 ENSG00000147082
"CCNA1" "CCNA2" "CCNB1" "CCNB2" "CCNB3" Challenge
パッケージ EnsDb.Hsapiens.v86 と TxDb.Hsapiens.UCSC.hg38.knownGene を読み込みます。 次に、それらのパッケージで利用可能なアノテーションの列を表示します。
R
library(EnsDb.Hsapiens.v86)
columns(EnsDb.Hsapiens.v86)
OUTPUT
[1] "ENTREZID" "EXONID" "EXONIDX"
[4] "EXONSEQEND" "EXONSEQSTART" "GENEBIOTYPE"
[7] "GENEID" "GENENAME" "GENESEQEND"
[10] "GENESEQSTART" "INTERPROACCESSION" "ISCIRCULAR"
[13] "PROTDOMEND" "PROTDOMSTART" "PROTEINDOMAINID"
[16] "PROTEINDOMAINSOURCE" "PROTEINID" "PROTEINSEQUENCE"
[19] "SEQCOORDSYSTEM" "SEQLENGTH" "SEQNAME"
[22] "SEQSTRAND" "SYMBOL" "TXBIOTYPE"
[25] "TXCDSSEQEND" "TXCDSSEQSTART" "TXID"
[28] "TXNAME" "TXSEQEND" "TXSEQSTART"
[31] "UNIPROTDB" "UNIPROTID" "UNIPROTMAPPINGTYPE" R
library(TxDb.Hsapiens.UCSC.hg38.knownGene)
columns(TxDb.Hsapiens.UCSC.hg38.knownGene)
OUTPUT
[1] "CDSCHROM" "CDSEND" "CDSID" "CDSNAME" "CDSPHASE"
[6] "CDSSTART" "CDSSTRAND" "EXONCHROM" "EXONEND" "EXONID"
[11] "EXONNAME" "EXONRANK" "EXONSTART" "EXONSTRAND" "GENEID"
[16] "TXCHROM" "TXEND" "TXID" "TXNAME" "TXSTART"
[21] "TXSTRAND" "TXTYPE" - Bioconductor は幅広いアノテーションパッケージを提供します。
- いくつかの Bioconductor ソフトウェアパッケージは、オンラインリソースにプログラム的にアクセスするために使用できます。
- ユーザーは、自分のニーズと期待に基づいてアノテーションのソースを慎重に選択するべきです。
Content from SummarizedExperimentクラス
Last updated on 2025-09-23 | Edit this page
Overview
Questions
- SummarizedExperimentオブジェクトの情報はどのように整理されていますか?
- その情報はどのように追加、編集、アクセスできますか?
Objectives
- 実験データとメタデータがどのように1つのオブジェクトに保存されるかを説明してください。
- データとメタデータを分析の過程全体で同期させることが重要な理由を説明してください。
パッケージのインストール
次のセクションに進む前に、必要なBioconductorのパッケージをいくつかインストールします。
最初に、BiocManagerパッケージがインストールされているか確認します。それがない場合はインストールします。
その後、BiocManager::install()関数を使用して必要なパッケージをインストールします。
R
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install(c("SummarizedExperiment"))
モチベーション
実験は多面的なデータセットで、通常、分析に必要な少なくとも2つの重要な情報を含んでいます:
- アッセイデータは、通常、サンプルのセット内の特徴のセットの測定を表す行列です (例:RNAシーケンシング)。
- サンプルメタデータは、通常、サンプルに関する情報を表す
data.frameです (例:治療群)。
これらすべての情報は、下流の分析で情報を正確に処理し、信頼できる結果を生成するために、 同じサンプルで同じ順序に保たれる必要があります。
分析ワークフローで、サンプルのサブセットを分析したり、より正確な下流の分析を可能にするために削除する必要のある外れ値を特定したりすることは非常に一般的です。 そのような場合、実験のすべての側面は同じサンプルのセットにサブセットされる必要があります -- 同じ順序で – データセットの一貫性と正確な結果を保つために。
SummarizedExperiment – SummarizedExperimentパッケージで実装されている
--
は、個々の実験の重要な側面を単一のオブジェクトに統合するコンテナを提供し、サブセットや再配置の操作中にデータとメタデータを調整します。
多くの生物学的データ型と包括的な機能セットを受け入れる柔軟性があり、
Bioconductor全体で再利用される人気のあるデータ構造です。
例えば、SummarizedExperimentに慣れていることは、 DESeq2パッケージや、シングルセル解析のためのSingleCellExperiment拡張クラスで作業するために必要です。
クラス構造
SummarizedExperimentは、行が対象の特徴(例:遺伝子、転写、エクソンなど)を表す行列のようなコンテナです。
列はサンプルを表します。
オブジェクトは1つ以上のアッセイを含むことができ、各アッセイは同じ次元の行列のようなオブジェクトで表されます。
特徴に関する情報は、SummarizedExperimentオブジェクト内にネストされたDataFrameオブジェクトに保存され、
関数rowData()を使用してアクセスできます。
DataFrameの各行は、SummarizedExperimentオブジェクトの対応する行の特徴に関する情報を提供します。
その情報には、実験とは無関係な注釈(例:遺伝子識別子)や、ワークフロー中にアッセイデータから計算された品質管理メトリックが含まれる場合があります。
同様に、サンプルに関する情報は、別のDataFrameオブジェクトに保存され、これもSummarizedExperimentオブジェクトにネストされ、
関数colData()を使用してアクセスできます。
次のグラフィックは、クラスのジオメトリを表示し、縦(列)および横(行)関係を強調します。 これは、SummarizedExperimentパッケージのビネットから得られました。
SummarizedExperimentオブジェクトの作成
はじめにパッケージをロードしましょう:
R
library(SummarizedExperiment)
それでは、レッスンセッティング中にダウンロードしたファイルからアッセイデータをインポートします。
そのファイルは単純なテキストファイルで、
最初の列には作られた特徴識別子が含まれ、
他のすべての列には作られたサンプルのシミュレーションデータが含まれています。
そのため、基本R関数read.csvを使用してファイルを解析し、
data.frameオブジェクトに変換できます。
以下の例では、行名は最初の列にあることを示しており、 関数は出力オブジェクト内ですぐに行名を設定します。 指定しなければ、関数は通常の列として解析し、デフォルトの整数インデックスで行名をそのままに 設定します。
R
count_data <- read.csv("data/counts.csv", row.names = 1)
count_data
OUTPUT
sample_1 sample_2 sample_3 sample_4
gene_1 109 84 91 105
gene_2 111 97 98 108
gene_3 89 121 105 99
gene_4 105 109 122 101
gene_5 82 97 112 83
gene_6 89 96 90 116
gene_7 121 95 88 106
gene_8 101 101 86 103
gene_9 91 119 89 87
gene_10 81 111 81 118
gene_11 93 118 93 99
gene_12 103 111 116 103
gene_13 89 126 103 100
gene_14 101 107 111 79
gene_15 96 91 103 108
gene_16 110 102 128 103
gene_17 95 106 118 100
gene_18 99 115 114 102
gene_19 114 105 94 118
gene_20 110 88 99 102
gene_21 116 95 94 105
gene_22 114 96 107 91
gene_23 97 120 93 90
gene_24 91 84 118 97
gene_25 99 106 97 1101つのアッセイデータ行列があれば、SummarizedExperimentオブジェクトを作成できますが、
サンプルメタデータがなければ、サンプルに関する情報を必要としない無監督分析のみが可能です。
以下の例では、カウントデータの行列を’counts’という名前で保存するSummarizedExperimentオブジェクトを作成します。
引数’Без’(複数形)は1つ以上のアッセイを受け入れることができ、 --
上記で説明されているように
–、そのため、唯一のアッセイ行列を名前付きのlistにカプセル化します。
アッセイに名前を付けることは、複数のアッセイを含むワークフローで、各アッセイを明確に識別し、取得するために重要です。
R
se <- SummarizedExperiment(
assays = list(counts = count_data)
)
se
OUTPUT
class: SummarizedExperiment
dim: 25 4
metadata(0):
assays(1): counts
rownames(25): gene_1 gene_2 ... gene_24 gene_25
rowData names(0):
colnames(4): sample_1 sample_2 sample_3 sample_4
colData names(0):上の出力では、オブジェクトの要約ビューがアッセイを確認でき、
そのため、全体的なSummarizedExperimentオブジェクトが4つのサンプル内の25の特徴に関する情報を含んでいることを思い出させます。
オブジェクトには、行メタデータや列メタデータは含まれていません。
より包括的なSummarizedExperimentオブジェクトを作成するために、他の2つのファイルから遺伝子メタデータとサンプルメタデータをインポートします。
ファイルはカウントデータと同様にフォーマットされているため、
再び基本R関数read.csv()を使用して、data.frameオブジェクトに解析します。
R
sample_metadata <- read.csv("data/sample_metadata.csv", row.names = 1)
sample_metadata
OUTPUT
condition batch
sample_1 A 1
sample_2 A 2
sample_3 B 1
sample_4 B 2R
gene_metadata <- read.csv("data/gene_metadata.csv", row.names = 1)
gene_metadata
OUTPUT
chromosome
gene_1 4
gene_2 4
gene_3 5
gene_4 4
gene_5 5
gene_6 1
gene_7 2
gene_8 1
gene_9 3
gene_10 1
gene_11 1
gene_12 5
gene_13 5
gene_14 1
gene_15 3
gene_16 4
gene_17 2
gene_18 5
gene_19 1
gene_20 3
gene_21 5
gene_22 5
gene_23 1
gene_24 4
gene_25 5これで、遺伝子とサンプルメタデータを含めたSummarizedExperimentオブジェクトを再作成できます:
R
se <- SummarizedExperiment(
assays = list(counts = count_data),
colData = sample_metadata,
rowData = gene_metadata
)
se
OUTPUT
class: SummarizedExperiment
dim: 25 4
metadata(0):
assays(1): counts
rownames(25): gene_1 gene_2 ... gene_24 gene_25
rowData names(1): chromosome
colnames(4): sample_1 sample_2 sample_3 sample_4
colData names(2): condition batchこの出力を前の’アッセイのみ’バージョンと比較すると、rowDataとcolData
のコンポーネントがそれぞれ1つと4つのメタデータを含むことがわかります。
情報にアクセスする
いくつかの関数がSummarizedExperimentオブジェクトのさまざまなコンポーネントにアクセスできます。
assays()関数は、オブジェクトに保存されたアッセイのリストを返します。
出力は常にListであり、オブジェクトに単一のアッセイが含まれている場合でもです。
R
assays(se)
OUTPUT
List of length 1
names(1): countsassayNames()関数は、アッセイ名の文字ベクトルを返します。
これは、オブジェクトに大量のアッセイが含まれる場合に最も便利です。
assays()関数(上記参照)は、すべてを表示しないかもしれません。
さまざまなアッセイの名前を知ることは、個々のアッセイにアクセスするための鍵です。
R
assayNames(se)
OUTPUT
[1] "counts"assay()関数を使用して、オブジェクトから単一のアッセイを取得できます。
これに対して、関数には目的のアッセイの名前または整数位置を指定する必要があります。
未指定の場合、関数は自動的にオブジェクト内の最初のアッセイを返します。
R
head(assay(se, "counts"))
OUTPUT
sample_1 sample_2 sample_3 sample_4
gene_1 109 84 91 105
gene_2 111 97 98 108
gene_3 89 121 105 99
gene_4 105 109 122 101
gene_5 82 97 112 83
gene_6 89 96 90 116colData()およびrowData()関数を使用して、サンプルメタデータおよび行メタデータを取得できます。
R
colData(se)
OUTPUT
DataFrame with 4 rows and 2 columns
condition batch
<character> <integer>
sample_1 A 1
sample_2 A 2
sample_3 B 1
sample_4 B 2R
rowData(se)
OUTPUT
DataFrame with 25 rows and 1 column
chromosome
<integer>
gene_1 4
gene_2 4
gene_3 5
gene_4 4
gene_5 5
... ...
gene_21 5
gene_22 5
gene_23 1
gene_24 4
gene_25 5また、
$演算子を使用して、サンプルメタデータの単一列にアクセスできます。
この演算子の便利な機能は、RStudioで自動的にトリガーされるオートコンプリートと、ターミナルアプリケーションでのタブキーを使用して自動的にトリガーされることです。
R
se$batch
OUTPUT
[1] 1 2 1 2特に、特徴メタデータの単一列にアクセスするための演算子はありません。
そのため、ユーザーはまずrowData()によって返される完全なDataFrameにアクセスし、
その後、標準の$または[[演算子を使用して列にアクセスする必要があります。
R
rowData(se)[["chromosome"]]
OUTPUT
[1] 4 4 5 4 5 1 2 1 3 1 1 5 5 1 3 4 2 5 1 3 5 5 1 4 5情報の追加と編集
情報は、SummarizedExperiment作成後に追加することができます。
実際、これは、正規化されたアッセイ値を計算し、
それをアッセイのリストに追加し、
特徴またはサンプルに対する品質管理メトリックを計算し、
それをrowDataとcolDataのコンポーネントに適切に追加し、
全体のオブジェクト内に保存された情報の量を徐々に増やすプロセスの基礎となります。
情報にアクセスするための多くの関数が、上記のセクションで説明されていますが、 新しい値を追加したり、既存の値を編集したりするための対応関数があります。 編集は、既に使用されている名前の下に値を追加するだけの結果であり、 既存の値を置き換える効果があります。
以下の例では、’counts’アッセイに対して1の擬似カウントを追加した後に、 その結果としてアッセイ名’logcounts’を追加します。
R
assay(se, "logcounts") <- log1p(assay(se, "counts"))
se
OUTPUT
class: SummarizedExperiment
dim: 25 4
metadata(0):
assays(2): counts logcounts
rownames(25): gene_1 gene_2 ... gene_24 gene_25
rowData names(1): chromosome
colnames(4): sample_1 sample_2 sample_3 sample_4
colData names(2): condition batch上の出力では、オブジェクトが現在2つのアッセイを持っていることがわかります: 最初に作成されたオブジェクトに含まれている’counts’アッセイと、 今追加した’logcounts’アッセイです。
同様に、colData()およびrowData()関数、
ならびに$演算子を使用して、対応するコンポーネントの値を追加および編集することができます。
以下の例では、各サンプルのカウントの合計を計算し、 結果を新しい名前’sum_counts’のサンプルメタデータテーブルに保存します。
R
colData(se)[["sum_counts"]] <- colSums(assay(se, "counts"))
colData(se)
OUTPUT
DataFrame with 4 rows and 3 columns
condition batch sum_counts
<character> <integer> <numeric>
sample_1 A 1 2506
sample_2 A 2 2600
sample_3 B 1 2550
sample_4 B 2 2533次の例では、各特徴の平均カウントを計算し、 新しい名前’mean_counts’の特徴メタデータテーブルに保存します。
R
rowData(se)[["mean_counts"]] <- rowSums(assay(se, "counts"))
rowData(se)
OUTPUT
DataFrame with 25 rows and 2 columns
chromosome mean_counts
<integer> <numeric>
gene_1 4 389
gene_2 4 414
gene_3 5 414
gene_4 4 437
gene_5 5 374
... ... ...
gene_21 5 410
gene_22 5 408
gene_23 1 400
gene_24 4 390
gene_25 5 412-
SummarizedExperimentクラスは、アッセイデータとメタデータの両方を保存するための単一のコンテナを提供します。 - アッセイデータとメタデータは、サブセット化と再配置の操作を通じて同期が保たれます。
-
SummarizedExperimentオブジェクトのさまざまなコンポーネントに保存された情報にアクセスしたり、追加したり、編集したりするための包括的な関数セットが利用可能です。
