RとRStudio
Last updated on 2025-11-11 | Edit this page
Overview
Questions
- RとRStudioとは何ですか?
Objectives
- RStudioの「スクリプト」「コンソール」「環境」「プロット」各ペインの機能と目的について説明する。
- Rプロジェクトとして一連の分析用ファイルとディレクトリを整理し、作業ディレクトリの役割を理解する。
- RStudioに標準搭載されているヘルプ機能を活用し、R関数に関する詳細情報を検索する方法を習得する。
- Rユーザーコミュニティに対して適切な情報を提供し、トラブルシューティングを効果的に行う方法を示す。
このエピソードは、Data Carpentriesの_Data Analysis and Visualisation in R for Ecologists_レッスンに基づいています。
Rとは何か? RStudioとは何か?
Rという用語は、以下の3つの概念を指すものとして用いられます: プログラミング言語、統計計算のための環境、そしてこの言語で記述されたスクリプトを解釈するソフトウェアです。
RStudioは現在、Rスクリプトの作成だけでなく、Rソフトウェアとの対話的な操作を行うための非常に人気の高いツールとなっています1。RStudioを正しく機能させるには、R本体のインストールが必須であり、両者をコンピュータにインストールする必要があります。
RStudio IDEチートシートにはここで紹介する内容以上の詳細な情報が記載されていますが、キーボードショートカットの習得や新機能の発見に役立つ有用な資料です。
Rを学ぶ理由
Rはマウス操作に頼らない設計――これはむしろ良いことです
他のソフトウェアに比べて学習曲線が急かもしれませんが、Rでは分析結果が一連のマウス操作の記憶に依存するのではなく、記述されたコマンドの系列によって得られる点が優れています。これはまさに良いことです! つまり、追加データを収集して分析をやり直す場合、どのボタンをどの順序でクリックしたかを覚える必要はなく、単にスクリプトを再実行すればよいのです。
スクリプトベースの作業は、分析プロセスの各ステップを明確に記録します。また、記述したコードは他者がレビュー可能であり、フィードバックを得たり誤りを発見したりするのに役立ちます。
スクリプトベースの作業は、自分が行っていることに対する深い理解を自然と促し、使用する手法の学習と理解を促進します。
Rコードは再現性に優れています
再現性とは、同じ分析コードを使用して同じデータセットを扱えば、他者(将来の自分自身を含む)が同じ結果を得られることを意味します。
Rは他のツールと連携することで、コードから論文やレポートを生成することが可能です。データをさらに収集した場合や、データセットに誤りを発見した場合でも、論文やレポート内の図表や統計テストは自動的に更新されます。
近年、多くの学術誌や資金提供機関が分析結果の再現性を求めています。Rを習得していれば、こうした要件に対応する上で有利になるでしょう。
Rは学際的で拡張性に優れています
CRAN(Comprehensive R Archive Network)で利用可能な1万以上のパッケージ2をインストールすることで、Rはその機能を拡張できるフレームワークを提供します。これにより、様々な科学分野の統計的アプローチを組み合わせ、データ解析に必要な分析フレームワークを最適に構築することが可能です。例えば、画像解析、GIS、時系列分析、集団遺伝学など、多岐にわたる分野に対応したパッケージが用意されています。
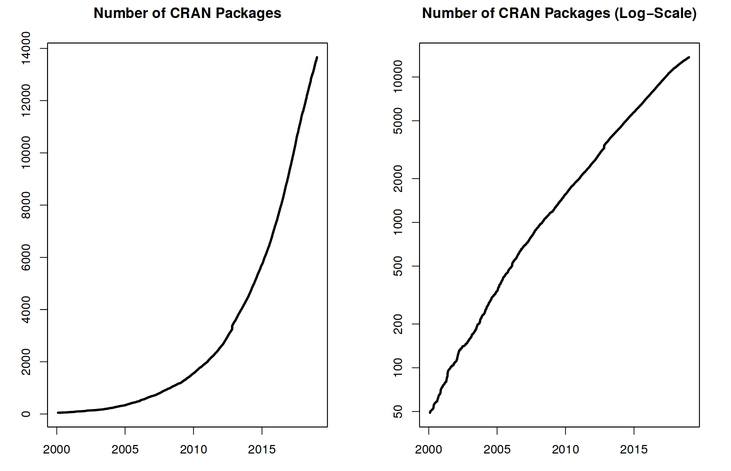
Rはあらゆる種類・規模のデータに対応します
Rで習得するスキルは、扱うデータセットの規模に容易に適応します。データセットの行数が数百行であろうと数百万行であろうと、作業効率にほとんど影響しません。
Rはデータ分析のために設計されています。欠損データの処理や統計的要因の考慮を容易にする特殊なデータ構造とデータ型を備えています。
Rはスプレッドシートやデータベース、その他の様々なデータ形式(コンピュータ上のものからウェブ上のものまで)に接続可能です。
Rには大規模で活発なコミュニティが存在する
何千人もの人々が日常的にRを利用しています。その中には、メーリングリストやStack Overflow、RStudioコミュニティなどのプラットフォームを通じて、積極的にサポートを提供してくれるユーザーが多数います。このような広範なユーザーコミュニティは、バイオインフォマティクスなどの専門分野にも及んでいます。Rコミュニティの代表的な専門組織の一つがBioconductorであり、「最新および新興の生物学的アッセイから得られるデータの解析と理解」を目的とした科学的プロジェクトです。このワークショップはBioconductorコミュニティのメンバーによって開発されました。Bioconductorに関する詳細情報は、関連ワークショップ『Bioconductorプロジェクト』をご覧ください。
RStudioの操作に慣れる
まずはRStudioについて学びましょう。これはRプログラミングのための統合開発環境(IDE)です。
RStudioのオープンソース製品は、Affero General Public License(AGPL)v3の下で無償で提供されています。また、Posit, Inc.が提供する商用ライセンス版では、優先的なメールサポートも利用可能です。
本ワークショップでは、RStudio IDEを使用してコードの記述、コンピュータ上のファイル操作、作成予定の変数の確認、生成するプロットの可視化を行います。なお、RStudioはバージョン管理、パッケージ開発、Shinyアプリの作成など、本ワークショップでは取り上げない他の用途にも使用可能です。
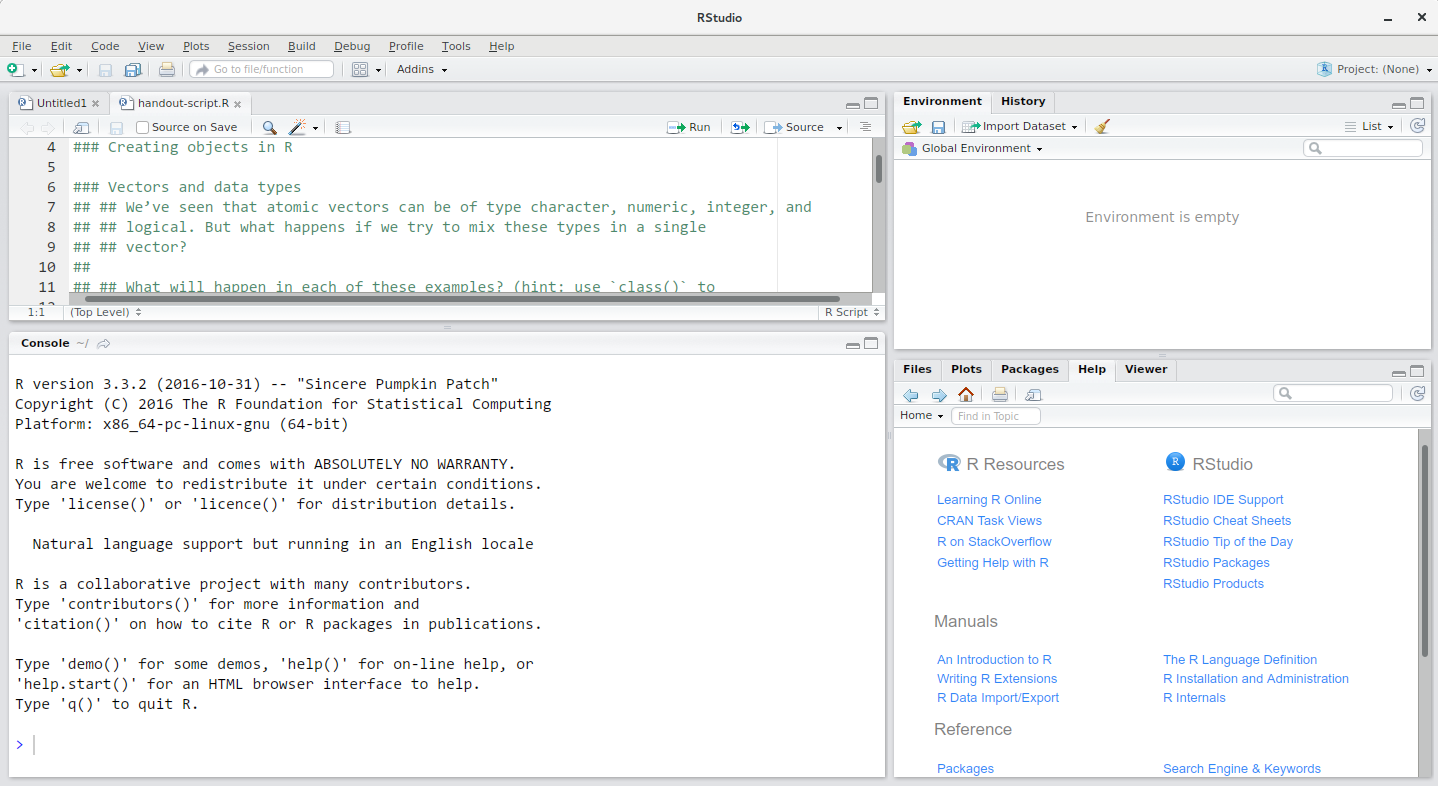
RStudioのウィンドウは4つの「パネル」に分割されています:
- ソースパネル:スクリプトやドキュメントの編集領域(デフォルトレイアウトでは左上)
- 環境/履歴パネル(右上)
- ファイル/プロット/パッケージ/ヘルプ/ビューアパネル(右下)
- Rコンソールパネル(左下)
これらのパネルの配置や内容はカスタマイズ可能です(メニューの「ツール」→「グローバルオプション」→「パネルレイアウト」から設定できます)。
RStudioを使用する利点の一つは、コード記述に必要なすべての情報が単一のウィンドウで確認できる点です。さらに、豊富なショートカットキー、自動補完機能、R開発時に頻繁に使用する主要ファイルタイプに対するハイライト表示などにより、タイピング作業がより容易になり、入力ミスも減少します。
作業環境のセットアップ方法
関連するデータ、分析結果、およびテキストファイルを単一のフォルダにまとめ、作業ディレクトリとして管理することは良い習慣です。このフォルダ内のすべてのスクリプトは、相対パスを使用してファイルを指定できます。相対パスとは、プロジェクト内のファイルの位置を示す方法です(絶対パスとは異なり、特定のコンピュータ上のファイルの正確な場所を指定するものです)。この方法を採用することで、プロジェクトをコンピュータ間で移動させたり、他者と共有したりする際にも、基礎となるスクリプトが正しく動作するかどうかを心配する必要がなくなります。
RStudioでは、「プロジェクト」インターフェースを通じて、この作業を容易にする便利なツールセットを提供しています。この機能は作業ディレクトリを作成するだけでなく、その場所を記憶し(簡単にアクセスできるようにします)、オプションでカスタム設定や開かれているファイルを保存することで、作業再開時の利便性を向上させます。以下の手順に従って、このチュートリアル用の「Rプロジェクト」を作成してください。
- RStudioを起動します。
-
ファイルメニューから新規プロジェクトをクリックします。次に新規ディレクトリを選択し、続けて新規プロジェクトを選びます。 - この新しいフォルダに名前を付けます(「ディレクトリ」とも呼ばれます)。また、作業しやすい場所を選択します。このフォルダがこのセッション(あるいはコース全体)の作業ディレクトリとなります(例:
bioc-intro)。 -
プロジェクトを作成をクリックします。 - (オプション) RStudioの設定で「ワークスペースを保存しない」を選択します。
RStudioのデフォルト設定は一般的に問題なく機能しますが、特に大規模なデータセットを扱う場合、ワークスペースを.RDataファイルに保存するのは煩雑になることがあります。この機能を無効にするには、ツールメニューからグローバルオプションを選択し、終了時にワークスペースを.RDataに保存するオプションを「しない」に設定してください。

Windowsと他のオペレーティングシステム間で発生する文字エンコーディングの問題を回避するため、デフォルトのテキストエンコーディングをUTF-8に設定します:
作業ディレクトリの整理方法
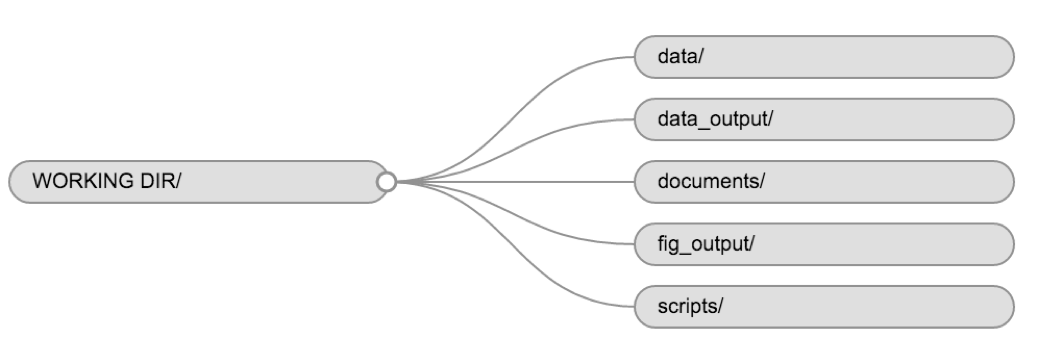
プロジェクト間で一貫したフォルダ構造を採用することで、作業環境を整理できるだけでなく、将来的にファイルを簡単に検索・管理できるようになります。特に複数のプロジェクトを扱う場合に有効です。一般的には、スクリプト用、データ用、ドキュメント用のディレクトリを作成するとよいでしょう。
-
data/このフォルダには、生データや特定の分析に必要な中間データセットを保存します。透明性とプロバンスを確保するため、常に生データのコピーをアクセス可能な状態で保持し、可能な限りデータのクリーニングや前処理はプログラム的に行う(手動ではなくスクリプトを使用する)ようにしてください。生データと処理済みデータを分離することも良い習慣です。例えば、data/raw/tree_survey.plot1.txtや...plot2.txtといったファイルは、scripts/01.preprocess.tree_survey.Rスクリプトによって生成されたdata/processed/tree.survey.csvファイルとは別に管理します。 -
documents/ここにはアウトライン、ドラフト、その他のテキストファイルを保存します。 -
scripts/(またはsrc) このフォルダには、各種分析やプロット作成用のRスクリプトを保存します。また、関数用の専用フォルダを設けることもできます(詳細は後述します)。
プロジェクトの要件に応じて追加のディレクトリやサブディレクトリが必要になる場合もありますが、これらが作業ディレクトリの基本構造となります。
本コースでは、生データを保存するためのdata/フォルダが必要です。また、CSVファイルとしてデータをエクスポートする方法を学ぶ際にはdata_output/フォルダを使用し、作成した図を保存するためのfig_output/フォルダも用意します。
課題: プロジェクト用ディレクトリ構造を作成する
画面右側の「ファイル」タブで、まず「新規フォルダ」をクリックし、
新たに作成した作業ディレクトリ内に「data」という名前のフォルダを作成します
(例:~/bioc-intro/data)。(別の方法として、Rコンソールでdir.create("data")と入力しても構いません)。
同様の操作を繰り返して、「data_output/」フォルダと「fig_output」フォルダを作成してください。
作業ディレクトリのルートディレクトリにスクリプトを保存することにします。 使用するファイルは1つだけであり、この方法を採用することで作業が より効率的になるからです。
作業ディレクトリの状態は以下のようになります:
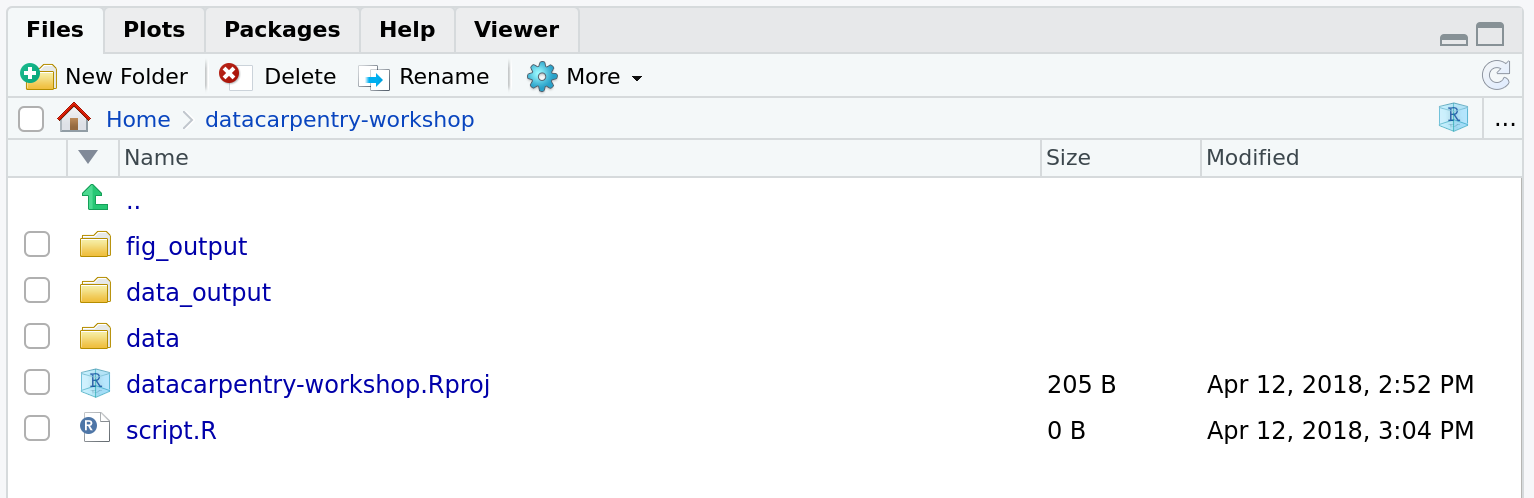
プロジェクト管理はバイオインフォマティクスプロジェクトにも 当然適用されます3。William Noble氏(@Noble:2009)は 以下のようなディレクトリ構造を提案しています:
ディレクトリ名は大文字で、ファイル名は小文字で表記しています。ここでは 一部のファイルのみを表示しています。日付は
<年>-<月>-<日>形式で 記載されており、時系列で並べ替えが可能です。ソースコードsrc/ms-analysis.cはコンパイルされてbin/ms-analysisとして実行可能になり、doc/ms-analysis.htmlでドキュメント化されています。データディレクトリ内のREADMEファイルには、 各データファイルが何月何日にどのURLからダウンロードされたかが記載されています。 ドライバスクリプトresults/2009-01-15/runallは自動的に3つのサブディレクトリ split1、split2、split3を生成します。これらは3つの交差検証分割に対応しています。bin/parse-sqt.pyスクリプトは、両方のrunallドライバスクリプトから呼び出されます。
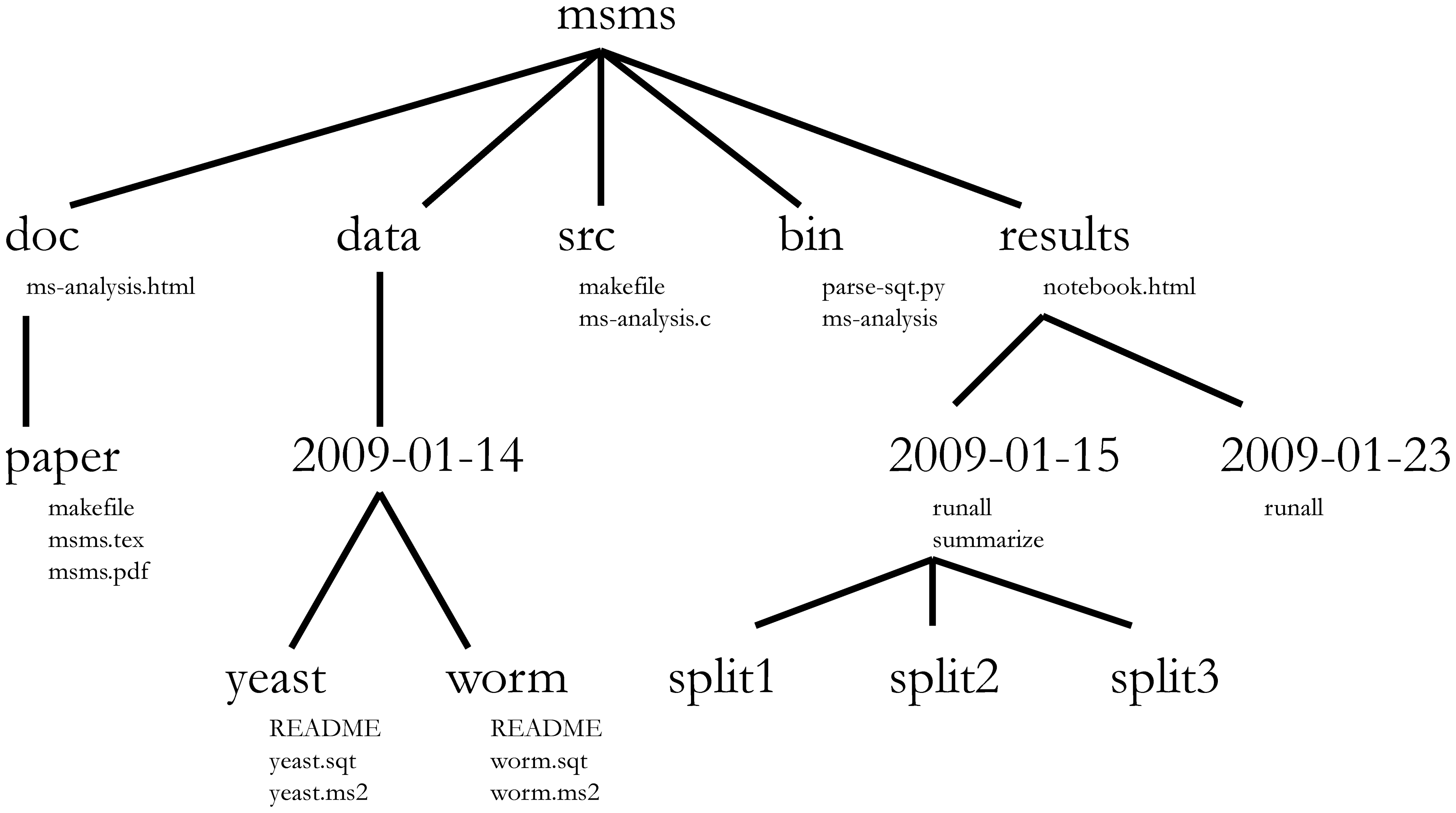
適切に定義され、十分に文書化されたプロジェクトディレクトリの最も重要な利点は、 プロジェクトに詳しくない人4が
プロジェクトの内容、利用可能なデータ、実施された分析、得られた結果を 理解できること、そして最も重要なのは
新しいデータを使用したり分析パラメータを変更したりして、 同じ分析を再現できることです。
作業ディレクトリ
作業ディレクトリは理解しておくべき重要な概念です。これはRがファイルの 検索と保存を行う基準となる場所です。プロジェクト用のコードを書く際には、 作業ディレクトリのルートを基準としてファイルを参照し、この構造内の ファイルのみを使用するようにしてください。
RStudioプロジェクトを使用すればこの作業が容易になり、作業ディレクトリが
適切に設定されることが保証されます。確認が必要な場合はgetwd()関数を使用できます。
何らかの理由で作業ディレクトリが想定通りでない場合は、RStudioのファイルブラウザで
作業ディレクトリが設定されるべき場所に移動し、青い歯車アイコンMoreを選択して
Set As Working Directoryを選択してください。別の方法として、setwd("/path/to/working/directory")
を使用して作業ディレクトリをリセットすることも可能です。ただし、この行をスクリプトに
含めると、他のコンピュータで実行した際にエラーが発生するため注意が必要です。
具体例
以下の図は、dataサブディレクトリとfig_outputサブディレクトリ、および
後者に2つのファイルを持つ作業ディレクトリbioc-introの構造を示しています:
bioc-intro/data/
/fig_output/fig1.pdf
/fig_output/fig2.png作業ディレクトリ内にいれば、fig1.pdfファイルには相対パスbioc-intro/fig_output/fig1.pdf
または絶対パス/home/user/bioc-intro/fig_output/fig1.pdfでアクセスできます。
dataディレクトリ内にいれば、相対パス../fig_output/fig1.pdfまたは同じ絶対パス
/home/user/bioc-intro/fig_output/fig1.pdfを使用してアクセスします。
Rとの対話操作
プログラミングの基本は、コンピュータが実行すべき指示を記述し、それを実際に実行させることにあります。私たちはR言語で指示を記述しますが、これはコンピュータと人間が共に理解できる共通言語だからです。これらの指示を「コマンド」と呼び、コンピュータに実行させるには「実行」(または「実行」とも呼ばれる)コマンドを発行します。
Rとの主な対話方法は2つあります:コンソールを使用する方法と、スクリプト(コードを記述したテキストファイル)を使用する方法です。RStudioでは、画面左下のコンソールパネルがR言語で記述したコマンドを入力し、即座にコンピュータに実行させることができる場所です。また、実行したコマンドの結果もこのコンソールに表示されます。コンソールに直接コマンドを入力してEnterキーを押せばコマンドを実行できますが、セッションを閉じると入力した内容は失われます。
コードと作業手順を再現可能にするため、コマンドはスクリプトエディタに入力して保存するのが理想的です。こうすることで、行った作業の完全な記録が残り、自分自身を含む誰もが簡単に同じ結果を再現できるようになります。ただし、スクリプトにコマンドを入力しただけでは自動的に実行されるわけではありません。コマンドはコンソールに送信して初めて実行される点に注意が必要です。
RStudioでは、Ctrl +
Enterキーのショートカット(MacではCmd +
Return)を使って、スクリプトエディタから直接コマンドを実行できます。スクリプトの現在行(カーソルで示される位置)のコマンド、または現在選択されているテキスト内のすべてのコマンドが、Ctrl
+
Enterを押すことでコンソールに送信され実行されます。その他のキーボードショートカットについては、RStudio
IDEに関するこのチートシートを参照してください。
分析作業中に、スクリプトに記録を残す必要はなくても、変数の内容を調べたりオブジェクトの構造を確認したい場合があります。このような場合はコンソールに直接コマンドを入力して実行できます。RStudioでは、Ctrl
+ 1とCtrl +
2のショートカットで、スクリプトとコンソールパネルを切り替えることができます。
Rがコマンドの入力を受け付けられる状態にある場合、コンソールには>プロンプトが表示されます。コマンドが入力されると(キーボード入力、コピー&ペースト、またはスクリプトエディタからCtrl
+
Enterで送信した場合)、Rはそのコマンドを実行しようとします。準備が整うと結果が表示され、新しい>プロンプトに戻って次のコマンドを待ちます。
Rがまだ完全なコマンドの入力を待っている場合、コンソールには+プロンプトが表示されます。これは、括弧や引用符が正しく閉じられていないことを意味します。つまり、開き括弧と閉じ括弧の数が一致していない、または開き引用符と閉じ引用符の数が一致していない状態です。この状態になった場合、コマンドの入力が完了したと思ったら、コンソールウィンドウ内でクリックしてEscキーを押してください。これにより不完全なコマンドがキャンセルされ、>プロンプトに戻ります。
コース中およびコース終了後の学習方法
本コースで扱う内容は、Rを使って自身の研究データを分析する方法の基本的な理解を得るのに役立ちます。しかし、データのクリーニング、統計手法の適用、美しいグラフの作成といった高度な操作を行うには、さらに学ぶ必要があります。Rを他のツールと同様に熟練して効率的に使いこなす最善の方法は、実際の研究課題に適用することです。初心者にとって、一からスクリプトを書くのは気が遠くなる作業に思えるかもしれませんが、多くの人がオンラインでコードを公開しているため、既存のコードを自分の目的に合わせて修正する方が、学習の第一歩として取り組みやすいでしょう。

ヘルプの活用方法
RStudio内蔵のヘルプインターフェースを使って、R関数に関する詳細情報を検索する
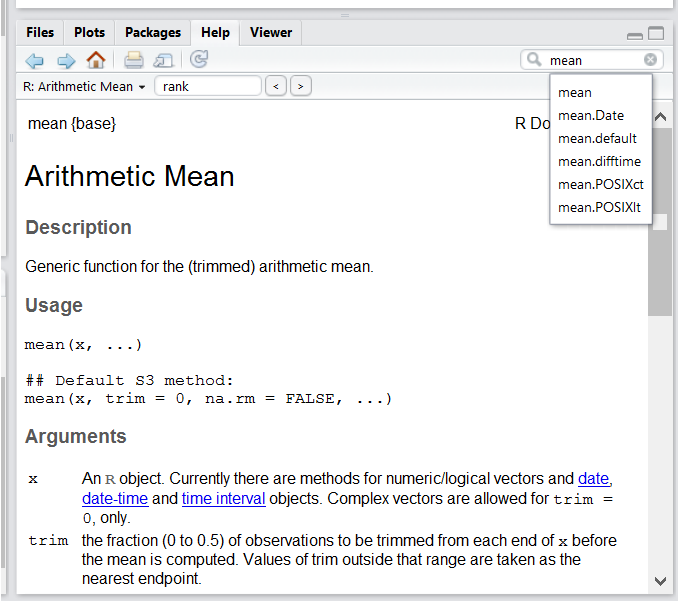
最も迅速なヘルプ取得方法の一つは、RStudioのヘルプインターフェースを使用することです。このパネルはデフォルトでRStudioの右下パネルに表示されます。スクリーンショットで示されているように、「Mean」(平均)という単語を入力すると、RStudioは関連する可能性のある複数の提案を表示します。その後、説明が表示ウィンドウに表示されます。
使用したい関数の名前は分かっているが、その使い方が分からない場合
特定の関数(例えば
barplot())の使用方法について知りたい場合は、以下のように入力してください:
R
?barplot
引数の名前だけを確認したい場合は、以下のコマンドが便利です:
R
args(lm)
Xのような処理を行う関数を使いたいが、該当する関数がどれか分からない場合
特定のタスクを実行するための関数を探している場合、help.search()関数を使用できます。これはダブル疑問符
??
で呼び出すことができます。ただし、この機能はインストール済みパッケージ内のヘルプページのみを検索し、検索キーワードに一致するものを探します
R
??kruskal
探している情報が見つからない場合は、rdocumentation.org というウェブサイトが便利です。このサイトでは利用可能なすべてのパッケージのヘルプファイルを検索できます。
最終的には、一般的なGoogle検索やインターネットで「R <タスク名>」と検索すると、適切なパッケージのドキュメントや、既に誰かが同じ質問をしている有益なフォーラムにたどり着けることが多いです。
行き詰まっている…理解できないエラーメッセージが表示される場合
まずはエラーメッセージをそのままGoogleで検索してみましょう。ただし、この方法が常に効果的とは限りません。多くの場合、パッケージ開発者はRのエラー処理機能に依存しているため、「subscript out of bounds」のような一般的なエラーメッセージが表示され、問題の診断に役立たないことがあります。エラーメッセージが非常に一般的な場合は、使用している関数やパッケージ名も検索クエリに含めると良いでしょう。
ただし、Stack
Overflowも必ず確認してください。[r]タグで検索すれば、ほとんどの質問にはすでに回答が付いています。重要なのは、適切なキーワードを使って検索し、答えを見つけることです:
https://stackoverflow.com/questions/tagged/r
R入門 はプログラミング経験が少ない方にはやや難解かもしれませんが、R言語の基礎を理解するには良い資料です。
R FAQ は技術的で密度の高い内容ですが、非常に有用な情報が満載です。
助けを求める際のポイント
誰かに助けを求める際の鍵は、問題を迅速に理解してもらうことです。問題の原因を特定しやすいように、できるだけ分かりやすく説明しましょう。
問題を説明する際には、正しい用語を使うことが重要です。例えば、「パッケージ」と「ライブラリ」は同じものではありません。ほとんどの人は意図を理解してくれるでしょうが、この2つの違いに強いこだわりを持つ人もいます。重要なのは、助けようとする人を混乱させないことです。問題を説明する際は、可能な限り具体的に記述しましょう。
可能であれば、動作しない部分をシンプルな「再現可能な例」に簡略化してください。50,000行10,000列のデータフレームではなく、ごく小規模なデータフレームで問題を再現できる場合は、問題の説明と一緒にその小規模なデータフレームを提供しましょう。適切な場合は、自分の専門分野以外の人でも理解できるように、できるだけ一般的な表現を使うようにしてください。例えば、実際のデータセットの一部を使う代わりに、3列5行程度の汎用的なデータセットを作成するなどです。再現可能な例の書き方については、Hadley Wickham氏のこちらの記事 が参考になります。
他の人とオブジェクトを共有する場合、オブジェクトが比較的小さい場合は、dput()関数を使用できます。これはメモリ上のオブジェクトと完全に同じオブジェクトを再作成できるRコードを出力します:
R
## irisはRに付属するサンプルデータフレームで、head()はデータフレームの先頭部分を返す関数です
dput(head(iris))
OUTPUT
structure(list(Sepal.Length = c(5.1, 4.9, 4.7, 4.6, 5, 5.4),
Sepal.Width = c(3.5, 3, 3.2, 3.1, 3.6, 3.9), Petal.Length = c(1.4,
1.4, 1.3, 1.5, 1.4, 1.7), Petal.Width = c(0.2, 0.2, 0.2,
0.2, 0.2, 0.4), Species = structure(c(1L, 1L, 1L, 1L, 1L,
1L), levels = c("setosa", "versicolor", "virginica"), class = "factor")), row.names = c(NA,
6L), class = "data.frame")オブジェクトが大きい場合は、生のファイル(つまりCSVファイル)と、エラーが発生する直前までのスクリプト全体(問題に関係のない部分は削除したもの)を提供するか、特にデータフレーム以外の質問の場合は、Rオブジェクトをファイルに保存することができます[^export]:
R
saveRDS(iris, file="/tmp/iris.rds")
ただし、このファイルの内容は人間が直接読める形式ではなく、Stack
Overflowに直接投稿することはできません。代わりに、メールなどで他の人に送信し、readRDS()コマンドで読み込ませることができます(ここではダウンロードしたファイルがユーザーのホームディレクトリのDownloadsフォルダにあることを前提としています):
R
some_data <- readRDS(file="~/Downloads/iris.rds")
最後に、必ずsessionInfo()の出力結果を含めてください。これは使用しているプラットフォーム、Rのバージョン、使用しているパッケージなど、問題を理解する上で非常に有用な情報を含んでいます。
R
sessionInfo()
OUTPUT
R version 4.5.2 (2025-10-31)
Platform: x86_64-pc-linux-gnu
Running under: Ubuntu 22.04.5 LTS
Matrix products: default
BLAS: /usr/lib/x86_64-linux-gnu/blas/libblas.so.3.10.0
LAPACK: /usr/lib/x86_64-linux-gnu/lapack/liblapack.so.3.10.0 LAPACK version 3.10.0
locale:
[1] LC_CTYPE=C.UTF-8 LC_NUMERIC=C LC_TIME=C.UTF-8
[4] LC_COLLATE=C.UTF-8 LC_MONETARY=C.UTF-8 LC_MESSAGES=C.UTF-8
[7] LC_PAPER=C.UTF-8 LC_NAME=C LC_ADDRESS=C
[10] LC_TELEPHONE=C LC_MEASUREMENT=C.UTF-8 LC_IDENTIFICATION=C
time zone: UTC
tzcode source: system (glibc)
attached base packages:
[1] stats graphics grDevices utils datasets methods base
loaded via a namespace (and not attached):
[1] assertthat_0.2.1 R6_2.6.1 xfun_0.52
[4] magrittr_2.0.4 glue_1.8.0 knitr_1.50
[7] sandpaper_0.17.2.9000 lifecycle_1.0.4 xml2_1.4.1
[10] ps_1.9.1 cli_3.6.5 processx_3.8.6
[13] callr_3.7.6 vctrs_0.6.5 renv_1.1.5
[16] withr_3.0.2 compiler_4.5.2 purrr_1.2.0
[19] tools_4.5.2 tinkr_0.3.0 evaluate_1.0.3
[22] yaml_2.3.10 BiocManager_1.30.25 pegboard_0.7.9
[25] rlang_1.1.6 サポートを受けるには?
- コース中に隣に座っている人:ワークショップ中は遠慮なく隣の人と話し、解答を比較したり、質問したりしてください。
- 経験豊富な同僚:もしあなたよりも経験豊かな人がいれば、その人が喜んで助けてくれるかもしれません。
- Stack Overflow:もしあなたの質問がまだ回答されておらず、よく練られたものであれば、5分以内に回答が得られる可能性が高いです。適切な質問の仕方のガイドラインに従うことを忘れないでください。
- R-helpメーリングリスト:このリストは多くのユーザー(Rコアチームの主要メンバーを含む)に読まれており、多くの投稿がありますが、やや事務的なトーンで、新規ユーザーに対して必ずしも親しみやすいとは限りません。質問が妥当なものであれば、迅速に回答が得られる可能性が高いですが、絵文字付きの回答は期待しないでください。また、他のどの場所よりも正確に専門用語を使用することが重要です(そうしないと、質問そのものではなく言葉の使い方についての回答が返ってくる可能性があります)。特定のパッケージに関する質問よりも、基本機能に関する質問の方がより適切な回答が得られる傾向があります。
- 特定のパッケージに関する質問の場合は、そのパッケージ用のメーリングリストがあるかどうかを確認してください。通常、パッケージの
DESCRIPTIONファイルに記載されており、packageDescription("パッケージ名")コマンドでアクセスできます。また、パッケージの著者に直接メールを送ったり、コードリポジトリ(例:GitHub)で問題をオープンにしたりすることも有効です。 - GISや系統発生学など特定のトピックに特化したメーリングリストもあります(完全なリストはRプロジェクトのメーリングリストページで確認できます)。
その他のリソース
Rヘルプの求め方:役立つガイドライン集
Jon Skeet氏によるこのブログ記事では、プログラミングに関する質問の仕方について非常に包括的なアドバイスが提供されています。
reprexパッケージは、サポートを求める際に再現可能な例を作成するのに非常に役立ちます。rOpenSciコミュニティが作成した「質問の仕方:回答を得るための方法」(GitHubリンクおよびビデオ録画)では、reprexパッケージとその哲学についてのプレゼンテーションが行われています。
Rパッケージ
パッケージの読み込み
前述の通り、RパッケージはRにおいて非常に重要な役割を果たします。インストール済みのパッケージの機能を利用するには、まずそのパッケージを読み込む必要があります。これはlibrary()関数を使用して行います。以下ではggplot2パッケージを読み込んでいます。
R
library("ggplot2")
パッケージのインストール
デフォルトのパッケージリポジトリはComprehensive R Archive
Network(CRAN)であり、CRANで利用可能なパッケージはすべてinstall.packages()関数でインストールできます。例えば以下では、後で学習するdplyrパッケージをインストールしています。
R
install.packages("dplyr")
このコマンドはdplyrパッケージ自体だけでなく、その動作に必要なすべての依存パッケージもインストールします。
もう一つの主要なRパッケージリポジトリはBioconductorが管理しています。Bioconductorパッケージは、専用パッケージであるBiocManagerを使用して管理およびインストールできます。これはCRANから以下のようにインストールできます:
R
install.packages("BiocManager")
その後、SummarizedExperiment(後で使用します)、DESeq2(RNA-Seq解析用)など、BioconductorまたはCRANの個別パッケージはBiocManager::installでインストール可能です。
R
BiocManager::install("SummarizedExperiment")
BiocManager::install("DESeq2")
デフォルトでは、BiocManager::install()はインストール済みパッケージをすべてチェックし、新しいバージョンが利用可能かどうかを確認します。新しいバージョンがあれば、それらを表示して「すべて/一部/すべて更新しますか?
[a/s/n]:」と尋ね、回答を待ちます。可能な限り最新のパッケージバージョンを使用することが望ましいですが、実際にはRセッションを新しく開始し、他のパッケージを読み込む前にパッケージを更新することを推奨します。
- RとRStudioを使い始める
