データから始める
Last updated on 2025-11-11 | Edit this page
Overview
Questions
- Rによる最初のデータ分析
Objectives
-
data.frameの概念について説明してください。 - .csv ファイルから外部データを読み込み、データフレームに格納する方法を示してください。
- データフレームの内容を要約する方法を説明してください。
- 因子(factor)の概念について説明してください。
- 文字列と因子間の変換方法を示してください。
- 因子の順序変更と名前変更の方法を説明してください。
- 日付のフォーマット方法を示してください。
- データのエクスポートと保存方法について説明してください。
このエピソードは、Data Carpentriesの_Data Analysis and Visualisation in R for Ecologists_レッスンに基づいています。
遺伝子発現データの説明
本研究では、Blackmoreら(2017)が公表したデータの一部を使用します。論文タイトルは『上気道感染症が中枢神経系のトランスクリプトーム変化に及ぼす影響』です。この研究の目的は、上気道感染症が感染後の小脳および脊髄におけるRNA転写変化に及ぼす影響を明らかにすることでした。性別を一致させた8週齢のC57BL/6マウスを用い、生理食塩水またはインフルエンザA型ウイルスを経鼻投与し、感染0日目、4日目、および8日目における小脳および脊髄組織のトランスクリプトーム変化をRNA-seq法で評価しました。
データセットはカンマ区切り値(CSV)ファイルとして保存されています。各行は単一のRNA発現測定値に関する情報を保持しており、最初の11列は以下の項目を表します:
| 列名 | 説明 |
|---|---|
| gene | 測定対象とした遺伝子名 |
| sample | 遺伝子発現測定を実施したサンプル名 |
| expression | 遺伝子発現値 |
| organism | 生物種/分類群 - 本データはすべてマウス由来です |
| age | マウスの年齢(すべてのマウスは8週齢) |
| sex | マウスの性別 |
| infection | マウスの感染状態 - インフルエンザA型に感染しているか否か |
| strain | インフルエンザA型の株 |
| time | 感染期間(日数) |
| tissue | 遺伝子発現実験に使用した組織 - 小脳または脊髄 |
| mouse | マウスの一意識別子 |
遺伝子発現データを含むCSVファイルのダウンロードにはR関数download.file()を使用し、取得したCSVファイルの内容をread.csv()でメモリ上にdata.frameクラスのオブジェクトとして読み込みます。download.file()コマンドの第1引数にはソースURLを指定する文字列を指定します。このURLはGitHubリポジトリからCSVファイルをダウンロードします。カンマの後に続くテキスト("data/rnaseq.csv")が、ローカルマシン上のファイル保存先パスとなります。事前に"data"という名前のフォルダを作成しておく必要があります。このコマンドはリモートファイルをダウンロードし、"rnaseq.csv"という名前を付けて既存の"data"フォルダに追加します。
R
download.file(url = "https://github.com/carpentries-incubator/bioc-intro/raw/main/episodes/data/rnaseq.csv",
destfile = "data/rnaseq.csv")
これでデータの読み込み準備が整いました。以下のようにデータを読み込めます:
R
rna <- read.csv("data/rnaseq.csv")
このコマンドは出力を表示しません。これは割り当て操作自体が出力を生成しないためです。データが正しく読み込まれたことを確認するには、データフレームの名前を直接入力してその内容を確認できます:
R
rna
うわっ…かなりの量の出力ですね。少なくともデータが正常に読み込まれたことを示しています。head()関数を使用してこのデータフレームの先頭6行を確認してみましょう:
R
head(rna)
OUTPUT
gene sample expression organism age sex infection strain time
1 Asl GSM2545336 1170 Mus musculus 8 Female InfluenzaA C57BL/6 8
2 Apod GSM2545336 36194 Mus musculus 8 Female InfluenzaA C57BL/6 8
3 Cyp2d22 GSM2545336 4060 Mus musculus 8 Female InfluenzaA C57BL/6 8
4 Klk6 GSM2545336 287 Mus musculus 8 Female InfluenzaA C57BL/6 8
5 Fcrls GSM2545336 85 Mus musculus 8 Female InfluenzaA C57BL/6 8
6 Slc2a4 GSM2545336 782 Mus musculus 8 Female InfluenzaA C57BL/6 8
tissue mouse ENTREZID
1 Cerebellum 14 109900
2 Cerebellum 14 11815
3 Cerebellum 14 56448
4 Cerebellum 14 19144
5 Cerebellum 14 80891
6 Cerebellum 14 20528
product
1 argininosuccinate lyase, transcript variant X1
2 apolipoprotein D, transcript variant 3
3 cytochrome P450, family 2, subfamily d, polypeptide 22, transcript variant 2
4 kallikrein related-peptidase 6, transcript variant 2
5 Fc receptor-like S, scavenger receptor, transcript variant X1
6 solute carrier family 2 (facilitated glucose transporter), member 4
ensembl_gene_id external_synonym chromosome_name gene_biotype
1 ENSMUSG00000025533 2510006M18Rik 5 protein_coding
2 ENSMUSG00000022548 <NA> 16 protein_coding
3 ENSMUSG00000061740 2D22 15 protein_coding
4 ENSMUSG00000050063 Bssp 7 protein_coding
5 ENSMUSG00000015852 2810439C17Rik 3 protein_coding
6 ENSMUSG00000018566 Glut-4 11 protein_coding
phenotype_description
1 abnormal circulating amino acid level
2 abnormal lipid homeostasis
3 abnormal skin morphology
4 abnormal cytokine level
5 decreased CD8-positive alpha-beta T cell number
6 abnormal circulating glucose level
hsapiens_homolog_associated_gene_name
1 ASL
2 APOD
3 CYP2D6
4 KLK6
5 FCRL2
6 SLC2A4R
## 以下の方法もお試しください
## View(rna)
注意
read.csv()関数はデフォルトでカンマをフィールド区切り文字として認識しますが、一部の国ではカンマが小数点区切りとして使用され、セミコロン(;)がフィールド区切り文字として用いられています。このような形式のファイルをRで読み込む場合は、read.csv2()関数を使用できます。この関数はread.csv()と完全に同様の動作をしますが、小数点区切り文字とフィールド区切り文字のパラメータが異なります。別の形式のファイルを扱う場合、これらの区切り文字はユーザーが指定可能です。詳細な情報については、?read.csvと入力してヘルプを参照してください。タブ区切りデータファイルを読み込む場合はread.delim()関数も利用可能です。これらの関数はすべて、異なる引数を持つ主要なread.table()関数のラッパー関数であることに注意が必要です。したがって、前述のデータはread.table()関数を使用し、区切り文字として,を指定することでも読み込むことができます。以下がそのコード例です:
R
rna <- read.table(file = "data/rnaseq.csv",
sep = ",",
header = TRUE)
header引数はデフォルトでread.table()がFALSEに設定されているため、ヘッダーを正しく読み込むためにはTRUEに設定する必要があります。
データフレームとは?
データフレームは、ほとんどの表形式データにおいて事実上の標準データ構造であり、統計処理やグラフ作成の際に用いられる基本的なデータ形式です。
データフレームは手動で作成することも可能ですが、実際にはread.csv()やread.table()といった関数を用いて作成されることが一般的です。つまり、ハードディスクやウェブ上のスプレッドシートデータをインポートする際に生成される形式です。
データフレームとは、列がすべて同じ長さのベクトルで構成される表形式のデータ表現です。列がベクトルであるため、各列には単一のデータ型(例:文字列、整数、因子など)のみが含まれます。例えば、以下の図は数値型、文字列型、論理型のベクトルで構成されるデータフレームの概念図です。
データフレームの構造をstr()関数で確認すると、この概念が明確に理解できます:
R
str(rna)
OUTPUT
'data.frame': 32428 obs. of 19 variables:
$ gene : chr "Asl" "Apod" "Cyp2d22" "Klk6" ...
$ sample : chr "GSM2545336" "GSM2545336" "GSM2545336" "GSM2545336" ...
$ expression : int 1170 36194 4060 287 85 782 1619 288 43217 1071 ...
$ organism : chr "Mus musculus" "Mus musculus" "Mus musculus" "Mus musculus" ...
$ age : int 8 8 8 8 8 8 8 8 8 8 ...
$ sex : chr "Female" "Female" "Female" "Female" ...
$ infection : chr "InfluenzaA" "InfluenzaA" "InfluenzaA" "InfluenzaA" ...
$ strain : chr "C57BL/6" "C57BL/6" "C57BL/6" "C57BL/6" ...
$ time : int 8 8 8 8 8 8 8 8 8 8 ...
$ tissue : chr "Cerebellum" "Cerebellum" "Cerebellum" "Cerebellum" ...
$ mouse : int 14 14 14 14 14 14 14 14 14 14 ...
$ ENTREZID : int 109900 11815 56448 19144 80891 20528 97827 118454 18823 14696 ...
$ product : chr "argininosuccinate lyase, transcript variant X1" "apolipoprotein D, transcript variant 3" "cytochrome P450, family 2, subfamily d, polypeptide 22, transcript variant 2" "kallikrein related-peptidase 6, transcript variant 2" ...
$ ensembl_gene_id : chr "ENSMUSG00000025533" "ENSMUSG00000022548" "ENSMUSG00000061740" "ENSMUSG00000050063" ...
$ external_synonym : chr "2510006M18Rik" NA "2D22" "Bssp" ...
$ chromosome_name : chr "5" "16" "15" "7" ...
$ gene_biotype : chr "protein_coding" "protein_coding" "protein_coding" "protein_coding" ...
$ phenotype_description : chr "abnormal circulating amino acid level" "abnormal lipid homeostasis" "abnormal skin morphology" "abnormal cytokine level" ...
$ hsapiens_homolog_associated_gene_name: chr "ASL" "APOD" "CYP2D6" "KLK6" ...データフレームオブジェクトの検査
すでにhead()関数とstr()関数がデータフレームの内容と構造を確認するのに有用であることを説明しました。以下に、データの内容/構造を把握するための代表的な関数を非網羅的に紹介します。実際に試してみましょう!
サイズ情報:
-
dim(rna)- 行数を第1要素、列数を第2要素とするベクトルを返します(オブジェクトの次元情報)。 -
nrow(rna)- 行数を返します。 -
ncol(rna)- 列数を返します。
内容情報:
-
head(rna)- 最初の6行を表示します。 -
tail(rna)- 最後の6行を表示します。
名前情報:
-
names(rna)- 列名を返します(data.frameオブジェクトの場合、colnames()関数と同じ機能です)。 -
rownames(rna)- 行名を返します。
要約情報:
-
str(rna)- オブジェクトの構造と、各列のクラス、長さ、内容に関する情報を提供します。 -
summary(rna)- 各列に対する要約統計量を表示します。
注記:
これらの関数の多くは「汎用関数」であり、data.frame以外の他のデータ型に対しても使用できます。
課題:
str(rna)
の出力結果に基づいて、以下の質問に答えてください:
- オブジェクト
rnaのクラスは何ですか? - このオブジェクトには行がいくつ、列がいくつありますか?
- class: データフレーム
- 行数: 32428、列数: 19
データフレームの索引付けと部分抽出
rna
データフレームは行と列を持つ2次元構造です。特定のデータを抽出する場合、取得したい「座標」を明示的に指定する必要があります。まず行番号を指定し、その後に列番号を指定します。ただし、これらの座標を指定する方法によって結果のクラスが異なる点に注意してください。
R
# データフレームの最初の行・最初の列の要素(ベクトルとして)
rna[1, 1]
# データフレームの6番目の列の最初の要素(ベクトルとして)
rna[1, 6]
# データフレームの最初の列全体(ベクトルとして)
rna[, 1]
# データフレームの最初の列全体(データフレームとして)
rna[1]
# データフレームの7番目の列の最初の3要素(ベクトルとして)
rna[1:3, 7]
# データフレームの3行目全体(データフレームとして)
rna[3, ]
# head_rna <- head(rna) と同等
head_rna <- rna[1:6, ]
head_rna
:
は特殊な関数で、整数の数値ベクトルを昇順または降順で生成します。例えば
1:10 や 10:1 のように使用します。詳細はセクション@ref(sec:genvec)を参照してください。
また、“-”
記号を使用してデータフレームの特定のインデックスを除外することも可能です:
R
rna[, -1] ## 最初の列を除いたデータフレーム全体
rna[-c(7:66465), ] ## head(rna) と同等
データフレームの部分抽出は、前述のようにインデックスを指定する方法のほか、列名を直接指定する方法でも行えます:
R
rna["gene"] # 結果はデータフレーム
rna[, "gene"] # 結果はベクトル
rna[["gene"]] # 結果はベクトル
rna$gene # 結果はベクトル
RStudioでは、自動補完機能を利用することで、列の完全かつ正確な名前を簡単に取得できます。
課題
rnaデータセットの200行目のみを含むdata.frameオブジェクトrna_200を作成してください。nrow()関数がdata.frameの行数を返す仕組みに注目してください。
この行数を利用して、初期の
rnaデータフレームから最後の行だけを抽出してください。tail()関数で表示される最後の行と比較し、期待通りの結果が得られていることを確認してください。行番号ではなく、
nrow()関数を使って最後の行を抽出してください。この最後の行から新しいデータフレーム
rna_lastを作成してください。
nrow()関数を使用して、rnaデータフレームの中央に位置する行を抽出してください。この行の内容をrna_middleという名前のオブジェクトに格納してください。nrow()関数と前述の-表記を組み合わせて、head(rna)関数と同様の動作を実現し、rnaデータセットの最初の6行のみを保持してください。
R
## 1.
rna_200 <- rna[200, ]
## 2.
## 可読性向上と重複削減のため `n_rows` を保存
n_rows <- nrow(rna)
rna_last <- rna[n_rows, ]
## 3.
rna_middle <- rna[n_rows / 2, ]
## 4.
rna_head <- rna[-(7:n_rows), ]
Factors
因子はカテゴリカルデータを表します。これらはラベルと関連付けられた整数として格納され、順序付きまたは順序なしのいずれかとして扱うことができます。 因子は見た目(そして多くの場合動作も)文字ベクトルのように見えますが、Rでは実際には整数ベクトルとして扱われます。したがって、文字列として扱う際には細心の注意が必要です。
一度作成された因子は、事前に定義された値のセット(レベルと呼ばれる)のみを含むことができます。デフォルトでは、Rは常にレベルをアルファベット順に並べ替えます。例えば、2つのレベルを持つ因子がある場合:
R
sex <- factor(c("male", "female", "female", "male", "female"))
Rはレベル "female" に 1 を、レベル
"male" に 2
を割り当てます(このベクトルの最初の要素が "male"
であっても、f は m
よりもアルファベット順で先に来るため)。levels()
関数を使用するとこの割り当てを確認でき、nlevels()
でレベルの総数を取得できます:
R
levels(sex)
OUTPUT
[1] "female" "male" R
nlevels(sex)
OUTPUT
[1] 2因子の順序が重要でない場合もありますが、意味的な意味を持つ場合(例:「低」「中」「高」)、可視化が向上する場合、あるいは特定の分析手法で必要とされる場合には、順序を指定したいことがあります。ここでは、sex
ベクトルのレベル順序を並べ替える方法の一例を示します:
R
sex ## 現在の順序
OUTPUT
[1] male female female male female
Levels: female maleR
sex <- factor(sex, levels = c("male", "female"))
sex ## 順序変更後
OUTPUT
[1] male female female male female
Levels: male femaleRのメモリ内では、これらの因子は整数(1, 2,
3)として表現されますが、因子は自己記述的であるため整数よりも情報量が豊富です:"female"
や "male" というラベルは、1 や 2
という数値よりもはるかに説明的です。どちらが「男性」を表すのか、整数値だけでは判断できません。一方、因子にはこの情報が組み込まれています。特にレベル数が多い場合(例:本例の遺伝子バイオタイプ)にこの機能は非常に有用です。
データが因子として格納されている場合、plot()
関数を使用することで、各因子レベルが表す観測値の数を素早く把握できます。ここでは、データ内の男性と女性の数を確認してみましょう。
R
plot(sex)
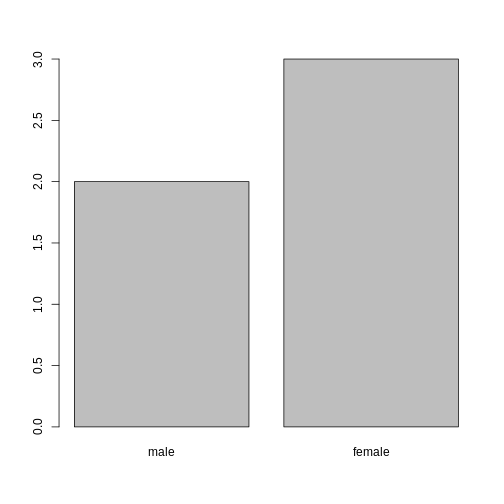
因子を文字ベクトルに変換する場合
因子を文字ベクトルに変換する必要がある場合は、as.character()関数を使用します。
R
as.character(sex)
OUTPUT
[1] "male" "female" "female" "male" "female"因子名の変更
これらの因子名を変更する場合、必要なのは因子の 水準を変更することだけです:
R
levels(sex)
OUTPUT
[1] "male" "female"R
levels(sex) <- c("M", "F")
sex
OUTPUT
[1] M F F M F
Levels: M FR
plot(sex)
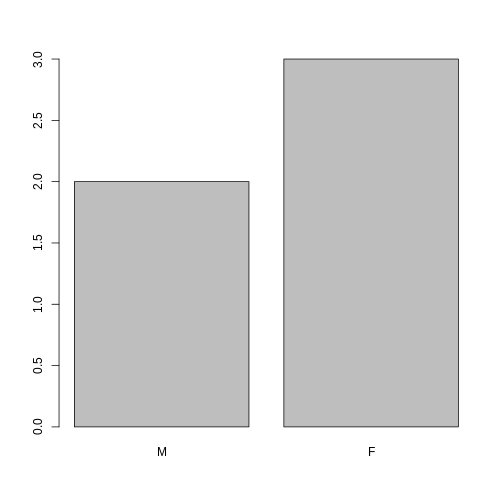
課題:
- 変数「F」と「M」をそれぞれ「Female」(女性)と「Male」(男性)に名称変更してください。
R
levels(sex)
OUTPUT
[1] "M" "F"R
levels(sex) <- c("Male", "Female")
課題:
read.csv()
関数を使用した場合のデータフレームの作成方法は理解できたと思いますが、
data.frame()
関数を使えば手動でデータフレームを作成することも可能です。
以下の手動で作成された data.frame
にはいくつかの誤りが含まれています。これらの誤りを見つけて修正できますか?
遠慮せずに試行錯誤してみてください!
- 動物の名称の前後に引用符が欠落している箇所がある
- 「感触」欄の1つのエントリーが欠落している(おそらく毛皮を持つ動物の1つに関するもの)
- 重量欄のカンマが1つ欠落している箇所がある
課題:
以下の例において、各列のデータ型を予測できますか?
str(country_climate)
コマンドを使用して予測結果を確認してください。
予測結果は予想通りでしたか?その理由は?予想と違った場合、その理由は?
データフレーム作成時に最後の変数の後に
stringsAsFactors = TRUEを追加してみてください。現在どのような現象が起こっていますか?stringsAsFactorsは、read.csv()関数を使用してテキストベースのスプレッドシートをRに読み込む際にも設定可能です。
R
country_climate <- data.frame(
country = c("カナダ", "パナマ", "南アフリカ", "オーストラリア"),
climate = c("寒冷", "温暖", "温帯", "温暖/温帯"),
temperature = c(10, 30, 18, "15"),
northern_hemisphere = c(TRUE, TRUE, FALSE, "FALSE"),
has_kangaroo = c(FALSE, FALSE, FALSE, 1)
)
R
country_climate <- data.frame(
country = c("カナダ", "パナマ", "南アフリカ", "オーストラリア"),
climate = c("寒冷", "温暖", "温帯", "温暖/温帯"),
temperature = c(10, 30, 18, "15"),
northern_hemisphere = c(TRUE, TRUE, FALSE, "FALSE"),
has_kangaroo = c(FALSE, FALSE, FALSE, 1)
)
str(country_climate)
OUTPUT
'data.frame': 4 obs. of 5 variables:
$ country : chr "カナダ" "パナマ" "南アフリカ" "オーストラリア"
$ climate : chr "寒冷" "温暖" "温帯" "温暖/温帯"
$ temperature : chr "10" "30" "18" "15"
$ northern_hemisphere: chr "TRUE" "TRUE" "FALSE" "FALSE"
$ has_kangaroo : num 0 0 0 1データ型の自動変換は、時には便利な機能ですが、時には厄介な問題を引き起こすこともあります。この機能が存在することを認識し、その規則を理解し、Rにインポートするデータがデータフレーム内で正しい型であることを必ず確認してください。もしそうでない場合は、データ入力時に生じた可能性のある誤り(例えば数値のみが含まれるべき列に文字データが含まれている場合など)を検出するためにこの機能を活用しましょう。
詳細については、RStudioのチュートリアルをご覧ください。
行列
データフレームについて学んだところで、次はパッケージのインストール方法を復習し、新しいデータ型である「行列」について学びましょう。行列はデータフレームと同様に2次元構造を持ち、行と列で構成されます。しかし決定的な違いは、行列のすべてのセルは同じ型でなければならないという点です:数値型、文字型、論理型など。この点において、行列はデータフレームよりもベクトルに近い性質を持っています。
行列のデフォルトコンストラクタは「matrix」関数です。この関数は、行列を埋めるための値のベクトルと、行数および/または列数1を引数として取ります。値は以下のように列方向に並べられます。
R
m <- matrix(1:9, ncol = 3, nrow = 3)
m
OUTPUT
[,1] [,2] [,3]
[1,] 1 4 7
[2,] 2 5 8
[3,] 3 6 9課題:
installed.packages()
関数を使用して、現在コンピュータにインストールされているすべてのパッケージに関する情報を含む
character
型の行列を作成してください。作成した行列を調査・分析してください。
R
## 行列の作成
ip <- installed.packages()
head(ip)
## View(ip) での表示もお試しください
## インストール済みパッケージの総数
nrow(ip)
## すべてのインストール済みパッケージ名
rownames(ip)
## 各パッケージについて取得可能な情報の種類
colnames(ip)
テストデータとして、大規模なランダムデータ行列を作成することは有用であることが多い。以下の演習では、平均
0、標準偏差 1
の正規分布からランダムに生成されたデータを含むこのような行列を作成することが求められる。これは
rnorm() 関数を使用して実行可能である。
課題:
平均0、標準偏差1の正規分布に従うデータからなる、1000行3列の行列を作成してください
R
set.seed(123)
m <- matrix(rnorm(3000), ncol = 3)
dim(m)
OUTPUT
[1] 1000 3R
head(m)
OUTPUT
[,1] [,2] [,3]
[1,] -0.56047565 -0.99579872 -0.5116037
[2,] -0.23017749 -1.03995504 0.2369379
[3,] 1.55870831 -0.01798024 -0.5415892
[4,] 0.07050839 -0.13217513 1.2192276
[5,] 0.12928774 -2.54934277 0.1741359
[6,] 1.71506499 1.04057346 -0.6152683日付のフォーマット処理
Rを使い始めたばかりのユーザー(そして経験豊富なユーザーも!)が直面する最も一般的な問題の一つが、日付と時刻情報を分析に適した変数形式に変換する作業です。
表計算ソフトにおける日付の取り扱いについて
表計算ソフトでは通常、日付情報は単一の列に格納されます。一見するとこれが自然な方法のように思えますが、実は最適な方法とは言えません。表計算ソフトは人間の目には正しく表示されたように見えますが、実際に日付をどのように処理・保存しているかについては問題が生じる可能性があります。日付を分析する際には、年・月・日を別々の列に格納するか、年と年の何日目かを別々の列に分ける方法の方がはるかに安全です。
LibreOffice、Microsoft Excel、OpenOffice、Gnumericなどの表計算ソフトでは、日付のエンコード方法が異なり(同じソフトでもバージョンやOSによって互換性がない場合もあります)、特にExcelでは日付でないデータを日付として処理してしまう問題が報告されています(@Zeeberg:2004)。例えば、MAR1、DEC1、OCT4といった名前や識別子も日付として扱われる可能性があります。そのため、日付形式そのものを避けることで、こうした問題を未然に防ぐことができます。
Data Carpentryの「日付データの扱い」セクションでは、表計算ソフトにおける日付処理の注意点について詳しく解説しています。
本解説では、tidyverseパッケージ群の一部であるlubridateパッケージのymd()関数を使用します(tidyverseの詳細については公式ウェブサイトをご覧ください)。lubridateはtidyverseのインストール時に自動的にインストールされます。tidyverseを読み込むと(library(tidyverse))、ほとんどのデータ分析で使用されるコアパッケージが自動的にロードされますが、lubridateはコアパッケージではないため、library(lubridate)で明示的に読み込む必要があります。
まず必要なパッケージを読み込みましょう:
R
library("lubridate")
ymd()関数は年・月・日を表すベクトルを受け取り、Date型のベクトルに変換します。DateはRが日付データとして認識するデータクラスで、これを適切に操作できます。関数の引数は柔軟な仕様となっていますが、ベストプラクティスとして「YYYY-MM-DD」形式の文字列ベクトルを使用することが推奨されます。
日付オブジェクトを作成し、構造を確認してみましょう:
R
my_date <- ymd("2015-01-01")
str(my_date)
OUTPUT
Date[1:1], format: "2015-01-01"次に、年・月・日を個別に結合してみます。同じ結果が得られることが確認できます:
R
# sepは各要素を区切る文字を指定します
my_date <- ymd(paste("2015", "1", "1", sep = "-"))
str(my_date)
OUTPUT
Date[1:1], format: "2015-01-01"ここでは一般的な日付操作の手順について説明します。以下のサンプルデータでは、日付が「year」「month」「day」の各列に別々に格納されています。
R
x <- data.frame(year = c(1996, 1992, 1987, 1986, 2000, 1990, 2002, 1994, 1997, 1985),
month = c(2, 3, 3, 10, 1, 8, 3, 4, 5, 5),
day = c(24, 8, 1, 5, 8, 17, 13, 10, 11, 24),
value = c(4, 5, 1, 9, 3, 8, 10, 2, 6, 7))
x
OUTPUT
year month day value
1 1996 2 24 4
2 1992 3 8 5
3 1987 3 1 1
4 1986 10 5 9
5 2000 1 8 3
6 1990 8 17 8
7 2002 3 13 10
8 1994 4 10 2
9 1997 5 11 6
10 1985 5 24 7この関数をxデータセットに適用します。まずpaste()関数を使ってxの「year」「month」「day」列から文字列ベクトルを作成します:
R
paste(x$year, x$month, x$day, sep = "-")
OUTPUT
[1] "1996-2-24" "1992-3-8" "1987-3-1" "1986-10-5" "2000-1-8" "1990-8-17"
[7] "2002-3-13" "1994-4-10" "1997-5-11" "1985-5-24"この文字列ベクトルをymd()関数の引数として使用できます:
R
ymd(paste(x$year, x$month, x$day, sep = "-"))
OUTPUT
[1] "1996-02-24" "1992-03-08" "1987-03-01" "1986-10-05" "2000-01-08"
[6] "1990-08-17" "2002-03-13" "1994-04-10" "1997-05-11" "1985-05-24"処理結果のDate型ベクトルは、xデータセットに新しい列「date」として追加できます:
R
x$date <- ymd(paste(x$year, x$month, x$day, sep = "-"))
str(x) # 新たに追加された列と、クラスが「date」となっていることを確認してください
OUTPUT
'data.frame': 10 obs. of 5 variables:
$ year : num 1996 1992 1987 1986 2000 ...
$ month: num 2 3 3 10 1 8 3 4 5 5
$ day : num 24 8 1 5 8 17 13 10 11 24
$ value: num 4 5 1 9 3 8 10 2 6 7
$ date : Date, format: "1996-02-24" "1992-03-08" ...正しく処理されているか確認しましょう。新しい列を確認する方法の一つとしてsummary()関数を使用する方法があります:
R
summary(x$date)
OUTPUT
Min. 1st Qu. Median Mean 3rd Qu. Max.
"1985-05-24" "1988-01-11" "1993-03-24" "1993-03-18" "1997-01-20" "2002-03-13" ymd()関数は年・月・日の順序で入力されることを前提としています。例えば日・月・年の順でデータがある場合にはdmy()関数を使用する必要があります。
R
dmy(paste(x$day, x$month, x$year, sep = "-"))
OUTPUT
[1] "1996-02-24" "1992-03-08" "1987-03-01" "1986-10-05" "2000-01-08"
[6] "1990-08-17" "2002-03-13" "1994-04-10" "1997-05-11" "1985-05-24"lubridateパッケージには、あらゆる種類の日付バリエーションに対応するための豊富な関数が用意されています。
Rオブジェクトの概要
これまでに、次元数や単一データ型・複数データ型の格納可否が異なる様々な種類のRオブジェクトを見てきました。主なものは以下の通りです:
-
ベクトル:1次元構造(長さを持つ)、単一データ型 -
行列:2次元構造、単一データ型 -
データフレーム:2次元構造、列ごとに異なるデータ型
リスト
これまで取り上げてこなかったものの、理解しておくと有用なデータ型として「リスト」があります。これは先ほどの概要から自然に導かれる概念です:
-
リスト:1次元構造で、各要素は異なるデータ型を取り得る
以下に、数値ベクトル、文字ベクトル、行列、データフレーム、および別のリストを含むリストを作成してみましょう:
R
l <- list(1:10, ## 数値データ
letters, ## 文字データ
installed.packages(), ## 行列データ
cars, ## データフレーム
list(1, 2, 3)) ## リスト
length(l)
OUTPUT
[1] 5R
str(l)
OUTPUT
List of 5
$ : int [1:10] 1 2 3 4 5 6 7 8 9 10
$ : chr [1:26] "a" "b" "c" "d" ...
$ : chr [1:162, 1:16] "abind" "askpass" "backports" "base64enc" ...
..- attr(*, "dimnames")=List of 2
.. ..$ : chr [1:162] "abind" "askpass" "backports" "base64enc" ...
.. ..$ : chr [1:16] "Package" "LibPath" "Version" "Priority" ...
$ :'data.frame': 50 obs. of 2 variables:
..$ speed: num [1:50] 4 4 7 7 8 9 10 10 10 11 ...
..$ dist : num [1:50] 2 10 4 22 16 10 18 26 34 17 ...
$ :List of 3
..$ : num 1
..$ : num 2
..$ : num 3リストの部分抽出には []
を使用して新しいサブリストを抽出するか、[[]]
を使用してリスト内の単一要素を抽出します(インデックス指定または名前指定が可能で、リストに名前が付けられている場合)。
R
l[[1]] ## 最初の要素を抽出
OUTPUT
[1] 1 2 3 4 5 6 7 8 9 10R
l[1:2] ## 長さ2のリストを抽出
OUTPUT
[[1]]
[1] 1 2 3 4 5 6 7 8 9 10
[[2]]
[1] "a" "b" "c" "d" "e" "f" "g" "h" "i" "j" "k" "l" "m" "n" "o" "p" "q" "r" "s"
[20] "t" "u" "v" "w" "x" "y" "z"R
l[1] ## 長さ1のリストを抽出
OUTPUT
[[1]]
[1] 1 2 3 4 5 6 7 8 9 10表形式データのエクスポートと保存
テキストベースのスプレッドシートをRに読み込む方法として、read.tableファミリーの関数を使用する方法を説明しました。data.frameをテキストベースのスプレッドシートとしてエクスポートする場合は、write.table関数群(write.csv、write.delimなど)を使用します。これらの関数はいずれも、エクスポート対象の変数と出力先ファイルを引数として受け取ります。例えば、rnaデータフレームをdata_outputディレクトリ内のmy_rna.csvファイルにエクスポートするには、以下のように実行します:
R
write.csv(rna, file = "data_output/my_rna.csv")
このようにして作成した新しいCSVファイルは、Rに詳しくない他の共同作業者と共有することが可能です。なお、data.frameの一部のフィールドにカンマが含まれている場合(例えば「product」列など)でも、Rはデフォルトで各フィールドを引用符で囲みます。そのため、列区切り文字としてもカンマを使用している場合でも、正しくRに再読み込みすることができます。
- Rでの表形式データ
行数か列数のいずれか一方を指定すれば十分で、もう一方は値の長さから自動的に計算されます。値の数と行数/列数が一致しない場合に何が起こるか、実際に試してみてください。↩︎
